논문 보러가기
저는 논문을 GPT로 읽는것에 익숙해서 이에 이질감에 드신다면 다른 리뷰를 찾아보는것이 좋습니다.
Abstarct
유전체 편집 기술, 특히 원핵 생물의 CRISPR-Cas 시스템에서 파생된 기술들은 생명 과학 연구에 혁명을 일으켰으며, 의학과 농업에도 큰 변화를 가져올 가능성이 있습니다.
Streptococcus pyogenes에서 유래한 Cas9 뉴클레아제(SpCas9)를 포함한 단일 단백질 CRISPR-Cas 효과기는 그 단순성, 강력함, 그리고 컴팩트한 형태로 인해 생명공학 분야에서 널리 사용되고 있습니다.
CRISPR 도구 상자를 다양화하고 편집 능력을 확장하기 위해 다양한 미생물 및 바이러스 유전체에서 새로운 시스템들이 발굴되었습니다. 이러한 새로운 시스템들은 작고, 생물학적 유체에서 단백질 안정성이 우수한 등 특정 속성을 위해 추구되었으나, 종종 목표 세포에서의 기본 활동성, PAM 선택성, 온도 최적화, 또는 시험관 내 생화학적 속성 등 중요한 속성에서 타협이 발생해 도달 범위가 제한됩니다.
단백질 언어 모델은 명시적인 구조 가설을 피하고, 대신 공동 진화적 청사진을 학습하여 단백질 기능을 학습합니다. 다양한 단백질 서열 세트에 대해 사전 학습된 언어 모델은 구조와 기능을 감독 없이 표현하는 방법을 학습합니다. 이러한 모델을 미세 조정하면 새로운 단백질을 생성할 수 있으며, 이들은 가족의 기능적 제약을 준수하면서도 서열 공간에서 상당히 다를 수 있습니다. 이러한 접근법은 기능적 리소자임을 설계하는 데 검증되었으며, 자연 단백질과의 서열 동일성이 31%에 불과한 상태에서도 성공적으로 시뮬레이션 되었습니다.
이 연구에서 우리는 언어 모델이 광범위한 CRISPR-Cas 단백질을 효과적으로 생성할 수 있음을 입증했습니다. 더욱이, 생성된 II형 효과 단백질이 수백 개의 돌연변이가 있는 상태에서도 인간 세포에서 기능하는 유전자 편집기로 조립될 수 있음을 보여주었습니다.
Abstarct 설명
우선 기본 용어부터 짚고 넘어 가보겠습니다.
CRISPR-Cas9 시스템은 유전자 편집 도구로 널리 사용되며, 이 시스템의 핵심 요소 중 하나가 바로 SpCas9입니다. SpCas9은 Streptococcus pyogenes라는 박테리아에서 유래한 Cas9 뉴클레아제를 의미합니다. 이 SpCas9과 관련된 다양한 개념을 구체적으로 설명하겠습니다.
1. SpCas9이란 무엇인가?
- SpCas9은 Streptococcus pyogenes에서 유래한 Cas9 단백질의 약칭입니다.
- Cas9은 CRISPR 시스템에서 핵심 역할을 하는 효소(뉴클레아제)로, DNA를 특정 위치에서 절단할 수 있는 능력을 가지고 있습니다.
- Streptococcus pyogenes는 인간에게 질병을 일으킬 수 있는 박테리아로, 이 박테리아의 CRISPR 시스템에서 Cas9이 처음 발견되었습니다. SpCas9은 특히 NGG PAM 서열을 인식하는 특징이 있으며, 유전자 편집 연구에서 가장 널리 사용되는 Cas9 변종입니다.
이미지 출처
2. SpCas9의 기능
- 유전자 편집: SpCas9은 가이드 RNA(sgRNA)와 결합하여 특정 DNA 서열을 타겟팅하고 절단할 수 있습니다. 이 기능을 통해 연구자들은 특정 유전자를 비활성화하거나 변경할 수 있습니다.
- PAM 서열 인식: SpCas9은 NGG라는 특정 PAM(Protospacer Adjacent Motif) 서열이 있는 DNA 부위를 인식하고 절단합니다. PAM 서열이 없다면, SpCas9은 해당 위치에서 DNA를 절단하지 못합니다.
3. 목표 세포에서의 기본 활동성 (Intrinsic Activity in Target Cells)
- 기본 활동성이란 SpCas9이 특정 목표 세포 내에서 얼마나 효율적으로 작동하는지를 의미합니다. 이는 SpCas9이 가이드 RNA와 결합하여 타겟 DNA를 절단하는 능력을 나타냅니다.
- 세포 내부의 다양한 환경 조건(예: pH, 이온 농도 등)은 SpCas9의 효율성에 영향을 미칠 수 있습니다. 예를 들어, SpCas9이 세포 내부에서 제대로 기능하지 않으면, 유전자 편집의 효율성이 저하될 수 있습니다.
4. PAM 선택성 (PAM Specificity)
- PAM 선택성이란 SpCas9이 어떤 PAM 서열을 인식할 수 있는지를 의미합니다. SpCas9은 NGG라는 특정 PAM 서열을 필요로 합니다.
- PAM 선택성은 유전자 편집의 정확성과 관련이 있습니다. 특정 PAM 서열을 인식함으로써 SpCas9은 DNA에서 정확한 위치를 타겟팅할 수 있습니다. 만약 PAM 선택성이 낮다면, 의도하지 않은 위치에서 DNA 절단이 발생할 수 있습니다(오프타겟 효과).
5. 온도 최적화 (Temperature Optimization)
- 온도 최적화는 SpCas9이 최적의 활동성을 발휘하는 데 필요한 온도 조건을 의미합니다.
- Cas9 단백질은 효소로서 특정 온도에서 가장 잘 작동합니다. SpCas9의 경우, 일반적으로 37°C(인체의 체온)가 최적의 활동성을 나타내는 온도로 알려져 있습니다. 온도가 이 범위를 벗어나면 Cas9의 효율성이 저하될 수 있습니다.
- 연구자들은 특정 실험 조건에서 SpCas9의 성능을 최적화하기 위해 온도를 조정할 수 있습니다.
6. 시험관 내 생화학적 속성 (In Vitro Biochemical Properties)
- 시험관 내 생화학적 속성이란 실험실에서 시험관 내에서(즉, 세포 외부에서) SpCas9의 효율성과 기능을 테스트하는 과정을 의미합니다.
- 여기에는 SpCas9이 특정 DNA 서열을 절단하는 효율성, 가이드 RNA와의 결합 강도, PAM 인식 능력, 그리고 절단 후 DNA 수리 메커니즘 등 다양한 생화학적 속성을 평가하는 것이 포함됩니다.
- 시험관 내 실험을 통해, SpCas9이 세포 내에서 어떻게 작동할지를 예측하고 최적화할 수 있습니다.
정리
- SpCas9은 Streptococcus pyogenes에서 유래한 DNA 절단 효소로, CRISPR-Cas9 유전자 편집 기술에서 핵심적인 역할을 합니다.
- 기본 활동성은 SpCas9이 목표 세포 내에서 얼마나 효율적으로 작동하는지를 의미하며, PAM 선택성은 SpCas9이 특정 DNA 서열을 인식하고 절단할 수 있는 능력을 나타냅니다.
- 온도 최적화는 SpCas9이 최적의 효율을 발휘하는 온도를 의미하며, 시험관 내 생화학적 속성은 실험실 환경에서 SpCas9의 기능과 성능을 평가하는 것을 말합니다.
AlphaFold2가 생각나는 논문입니다. alphafold2에서는
단백질의 구조를 분석했다면 이 논문은 새로운 단백질을 생성하는 것이 주 기능인 것 같습니다.
Result

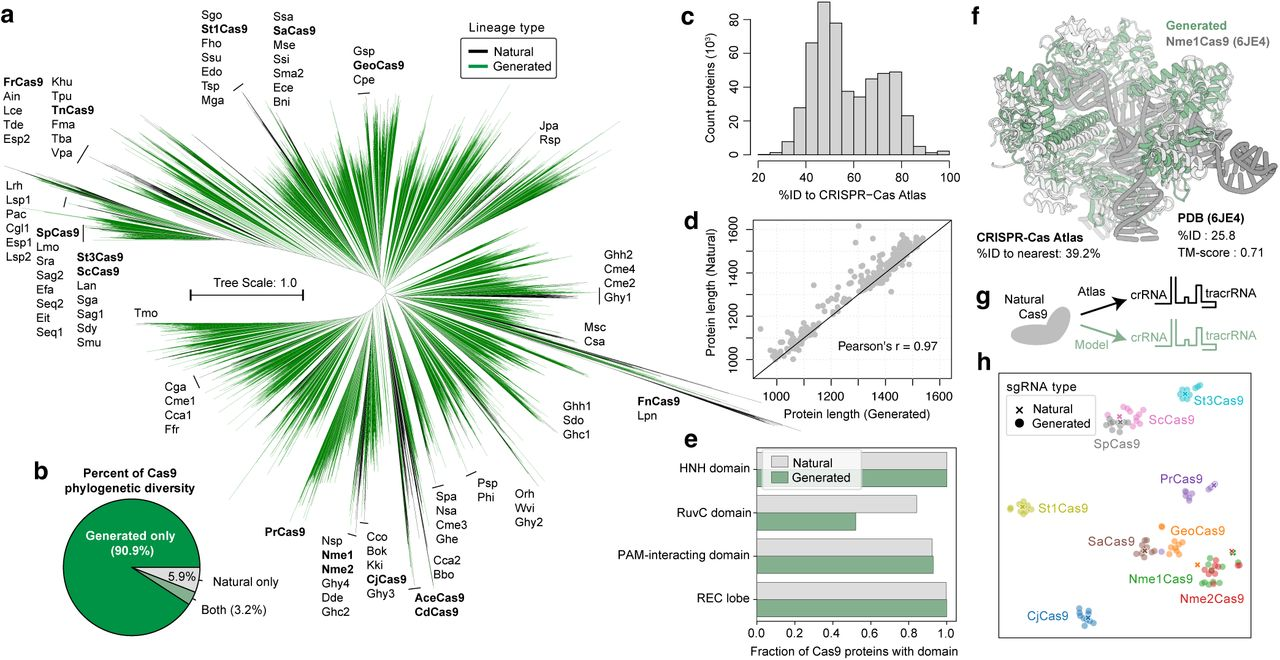
많은 CRISPR-Cas 단백질이 유전자 편집에 활용되고 있지만, Cas9은 여전히 가장 널리 사용되고 있다고 합니다. 새로운 Cas9 유사 서열을 생성하기 위해, 연구진은 CRISPR-Cas 모델에 CRISPR-Cas 아틀라스에서 샘플링한 Cas9의 N- 또는 C-말단에서 50개의 잔기를 제공하여 생성 방향을 유도했습니다. 그러나 이러한 유도 생성의 경우 단 27.6%만이 우리의 엄격한 서열 생존 필터를 통과했습니다. 보다 효율적이고 정확하게 생존 가능한 Cas9 유사 서열을 생성하기 위해, CRISPR-Cas 아틀라스의 238,917개의 Cas9 서열만을 사용하여 또 다른 언어 모델을 미세 조정했습니다(Fig. 1a 및 Fig. S1). 이 모델은 CRISPR-Cas 모델보다 두 배 높은 비율로 생존 가능한 Cas9 유사 서열을 생성했으며, 유도 생성이 필요하지 않았다고 합니다.
이 부분에서 설명하고 있는 것은, 딥러닝 기반의 단백질 언어 모델을 사용하여 Cas9 유사 단백질을 설계하는 과정에서 효율성과 정확성을 높이기 위해 어떻게 접근했는지를 설명하고 있습니다. 이를 좀 더 구체적으로 분석해보겠습니다.
1. 서열 생존 필터
- 서열 생존 필터란, 딥러닝 모델이 생성한 Cas9 유사 단백질 서열 중에서 기능적으로 의미가 있는 서열만을 선택하는 과정입니다. 이 필터는 모델이 생성한 서열들이 실제로 생물학적으로 유효한지(즉, 기능할 수 있는지)를 평가하는 단계입니다.
- 처음에 생성된 서열 중 7.6%만이 이 필터를 통과했음을 언급하고 있습니다. 이는 초기 모델이 생성한 대부분의 서열이 기능적으로 유효하지 않거나 충분히 안정적이지 않았다는 것을 의미합니다.
2. 보다 효율적이고 정확한 서열 생성
- 처음에는 다양한 CRISPR-Cas 서열을 포함하는 데이터를 사용하여 모델을 학습시켰지만, 그 결과 효율성이 낮고 대부분의 생성된 서열이 생존 필터를 통과하지 못했습니다.
- 이를 개선하기 위해 연구팀은 CRISPR-Cas 아틀라스라는 대규모 데이터베이스에서 238,917개의 Cas9 서열만을 사용하여 모델을 다시 미세 조정(재학습)했습니다. 즉, 이번에는 Cas9 서열에만 집중하여 모델을 학습시킨 것입니다.
- 이 과정에서 모델의 생성 효율과 정확성이 크게 향상되었습니다. 구체적으로, 이 모델은 이전보다 두 배 높은 비율로 생존 가능한 Cas9 유사 서열을 생성할 수 있었습니다.
3. 유도 생성이 필요하지 않았음
- 유도 생성이란, 모델이 특정 방향으로 서열을 생성하도록 인위적인 단서를 제공하는 방법입니다. 예를 들어, 특정 서열의 일부를 미리 제공하여 나머지 서열을 완성하도록 하는 방식입니다.
- 새롭게 미세 조정된 모델은 이러한 유도 생성 없이도 효율적으로 생존 가능한 서열을 생성할 수 있었습니다. 이는 모델이 Cas9 서열을 충분히 학습했으며, 자체적으로 기능적으로 유효한 서열을 생성할 수 있는 능력이 강화되었음을 의미합니다.
이 부분은 초기 모델이 생성한 서열 중 대부분이 생존 필터를 통과하지 못했기 때문에, Cas9 서열에 초점을 맞춰 모델을 다시 학습시켜 더 효율적이고 정확하게 생존 가능한 서열을 생성한 방법을 설명합니다. 이 재학습된 모델은 유도 생성 없이도 두 배 더 많은 생존 가능한 Cas9 유사 서열을 생성할 수 있었으며, 이는 모델의 성능이 크게 향상되었음을 보여줍니다.
.
논문의 "Results" 부분은 딥러닝을 활용하여 새로운 CRISPR-Cas 단백질을 설계하고, 그 단백질들이 실제로 기능할 수 있는지를 검증하는 과정을 설명합니다. 이 과정에서 얻어진 핵심적인 발견과 결과는 다음과 같습니다.
1. 딥러닝을 통한 CRISPR-Cas 단백질 생성
- 연구진은 단백질 언어 모델(ProGen2)을 사용하여 다양한 Cas 단백질 서열을 생성했습니다. 이 모델은 대규모의 자연 단백질 서열 데이터셋을 학습하여 새로운 단백질 서열을 창출하는 능력을 가지고 있습니다.
- 생성된 서열은 기존의 자연 서열에 비해 다양성이 4.8배 증가했으며, 특히 Cas9 및 Cas12a와 같은 계열에서는 6-8배의 다양성이 증가했습니다. 이는 딥러닝이 자연적으로 존재하지 않는 새로운 단백질을 설계할 수 있음을 보여줍니다.
2. Cas9 유사 단백질의 설계 및 검증
- 연구팀은 딥러닝 모델을 통해 100만 개 이상의 Cas9 유사 단백질 서열을 생성하고, 이 중 생존 가능한 서열을 선택했습니다. 선택된 서열들은 자연에서 발견된 Cas9 단백질보다 다양한 특성을 가지고 있었으며, 기존의 CRISPR-Cas 아틀라스 데이터베이스보다 10배 이상 확장된 계통수를 나타냈습니다.
- 실험적으로, 생성된 Cas9 유사 단백질들이 인간 세포에서 유전자 편집 기능을 수행할 수 있음을 확인했습니다. 특히, OpenCRISPR-1이라는 단백질은 높은 온타겟 특이성과 낮은 오프타겟 활동을 보여주며, SpCas9과 비교해도 뛰어난 성능을 보였습니다.
3. 베이스 에디팅과의 응용 가능성
- 연구팀은 OpenCRISPR-1 단백질을 베이스 에디팅(Base Editing) 시스템과 결합하여 테스트했습니다. 베이스 에디터는 DNA의 특정 염기를 변환하는 데 사용되며, OpenCRISPR-1은 SpCas9과 유사한 성능을 보여주었습니다.
- 이 결과는 딥러닝으로 설계된 새로운 단백질이 기존의 유전자 편집 도구들과 호환되어 효과적으로 사용될 수 있음을 입증합니다.
4. 가이드 RNA 설계 및 효율성
- 연구팀은 딥러닝 모델을 사용하여 새로운 Cas9 유사 단백질에 맞는 가이드 RNA(sgRNA)도 설계했습니다. 설계된 sgRNA는 SpCas9의 sgRNA와 유사한 성능을 보였으며, 일부는 더 높은 편집 효율성을 나타냈습니다.
- 이는 딥러닝을 통해 특정 단백질에 맞춘 가이드 RNA를 설계함으로써 유전자 편집의 정확성과 효율성을 높일 수 있음을 보여줍니다.
핵심 결론
- 딥러닝을 활용하여 기존의 CRISPR-Cas 단백질보다 더 다양한 특성을 가진 새로운 단백질을 설계할 수 있으며, 이러한 단백질들이 인간 세포에서 실제로 기능할 수 있음을 입증했습니다.
- OpenCRISPR-1과 같은 새로운 단백질은 기존의 SpCas9과 유사하거나 더 나은 성능을 보였으며, 이는 딥러닝 기반 단백질 설계의 실질적인 응용 가능성을 보여줍니다.
- 이러한 연구 결과는 유전자 편집 기술의 새로운 가능성을 열어주며, 맞춤형 단백질 설계를 통해 다양한 생물학적 및 의학적 응용이 가능하다는 것을 시사합니다.
알기 쉽게 요약하면 이렇게 요약 가능할 것 같습니다.
- 이전에 CRISPR-Cas 계열의 단백질을 훈련시켰다.
- 망했다.
- Cas9 서열만 추출해서 다시 fine tuning 했다.
- 생존 가능한 서열(성공한 단백질) 생성 확률이 증가했다.
- 이 과정에서 유도생성 없이도 생존 가능한 단백질이 만들어졌다.
근데 과연 이 모델을 어떻게 평가했을까 궁금했는데 여러 evaluation 방법들과 AlphaFold2를 사용했습니다.
1. Pearson 상관 계수
- Pearson 상관 계수는 두 변수 간의 선형 상관 관계를 측정하는 통계적 지표입니다. 값은 -1에서 1 사이로 나타나며, 1에 가까울수록 두 변수 간의 강한 양의 상관관계를 의미합니다.
- 이 연구에서는 생성된 단백질 서열과 자연 단백질 서열의 길이 간의 상관 관계를 평가했습니다. Pearson 상관 계수가 0.97이라는 것은 두 서열 길이 간의 관계가 매우 강하게 일치함을 의미합니다. 즉, 생성된 서열이 자연 서열의 길이와 매우 유사하다는 것을 나타냅니다.
2. pLDDT (Predicted Local Distance Difference Test)
- pLDDT는 단백질 구조 예측에서 사용되는 신뢰도 점수로, AlphaFold2와 같은 단백질 구조 예측 모델이 각 아미노산의 위치를 얼마나 정확하게 예측했는지를 나타냅니다. pLDDT 점수는 0에서 100까지의 범위로 나타나며, 값이 높을수록 예측된 구조의 신뢰도가 높습니다.
- 여기서 99.4%의 구조가 pLDDT 80 이상을 기록했다는 것은 생성된 단백질 서열이 매우 정확하게 예측된 구조를 가지고 있음을 시사합니다.
3. tracrRNA (Trans-activating CRISPR RNA)
- tracrRNA는 CRISPR 시스템에서 중요한 역할을 하는 RNA 분자입니다. tracrRNA는 crRNA(CRISPR RNA)와 결합하여 Cas 단백질과 함께 기능하며, 타겟 DNA 서열을 인식하고 절단합니다.
- tracrRNA는 crRNA와 함께 Cas 단백질에 결합하여 복합체를 형성하고, 이 복합체가 타겟 DNA 서열을 인식하고 정확하게 절단할 수 있도록 돕습니다.
4. gRNA (Guide RNA)
- gRNA는 가이드 RNA로, CRISPR-Cas 시스템에서 타겟 DNA 서열을 인식하는 역할을 합니다. gRNA는 crRNA와 tracrRNA를 결합하여 하나의 RNA 분자로 만든 것입니다.
- sgRNA (single guide RNA)는 gRNA의 한 형태로, crRNA와 tracrRNA가 하나의 분자로 합쳐진 구조입니다. sgRNA는 Cas 단백질과 함께 타겟 DNA 서열을 인식하고, 이 서열에 결합하여 Cas 단백질이 정확한 위치에서 DNA를 절단하도록 안내합니다.
5. crRNA (CRISPR RNA)
- crRNA는 CRISPR 시스템의 일부분으로, 특정 DNA 서열을 인식하는 RNA입니다. crRNA는 타겟 DNA 서열과 상보적인 서열을 가지고 있으며, 이는 gRNA의 일부가 됩니다.
- crRNA는 tracrRNA와 결합하여 Cas 단백질과 함께 작동하며, 타겟 DNA를 정밀하게 인식하고 절단하는 데 중요한 역할을 합니다.
6. t-SNE (t-distributed Stochastic Neighbor Embedding)
- t-SNE는 고차원 데이터(예: 유전자 서열)를 2차원 또는 3차원 공간으로 시각화하는 데 사용되는 기법입니다. t-SNE는 데이터의 유사성을 보존하면서 복잡한 데이터 구조를 시각화하는 데 유용합니다.
- 이 연구에서 t-SNE는 gRNA 서열 간의 유사성을 시각화하는 데 사용되었습니다. 이를 통해 모델에서 설계된 sgRNA가 자연적으로 유래된 sgRNA와 얼마나 유사한지를 평가했습니다.
7. 생성된 단백질의 구조적 생존 가능성 평가
- 연구진은 AlphaFold2를 사용하여 생성된 Cas9 유사 단백질과 자연 단백질의 3D 구조를 예측했습니다. 대부분의 예측된 구조가 높은 신뢰도(pLDDT 80 이상)를 보였으며, 이는 생성된 서열이 자연 단백질과 유사한 구조를 형성할 가능성이 높음을 의미합니다.
- 또한, 실험적으로 결정된 구조와의 비교를 통해 생성된 단백질이 자연적으로 존재하는 단백질과 얼마나 유사한지를 평가했습니다. 연구진은 생성된 단백질이 Cas9의 핵심 기능을 담당하는 도메인(예: HNH 및 RuvC 뉴클레아제 도메인)을 보존하고 있음을 확인했습니다.
8. gRNA 모델의 검증
- 연구진은 특정 Cas9 유사 단백질에 대해 gRNA를 설계하고, 이들이 타겟 DNA 서열을 인식하고 절단할 수 있는지를 검증했습니다. 모델에서 설계된 gRNA는 자연적으로 유래된 gRNA와 유사한 기능을 나타냈으며, 이는 모델이 새로운 Cas9 유사 단백질에 맞는 기능적 gRNA를 설계할 수 있음을 시사합니다.
이 부분은 생성된 Cas9 유사 단백질의 생존 가능성과 구조적 유사성, 그리고 이들이 CRISPR-Cas 시스템에서 실제로 기능할 수 있는지를 평가하는 과정을 설명합니다.
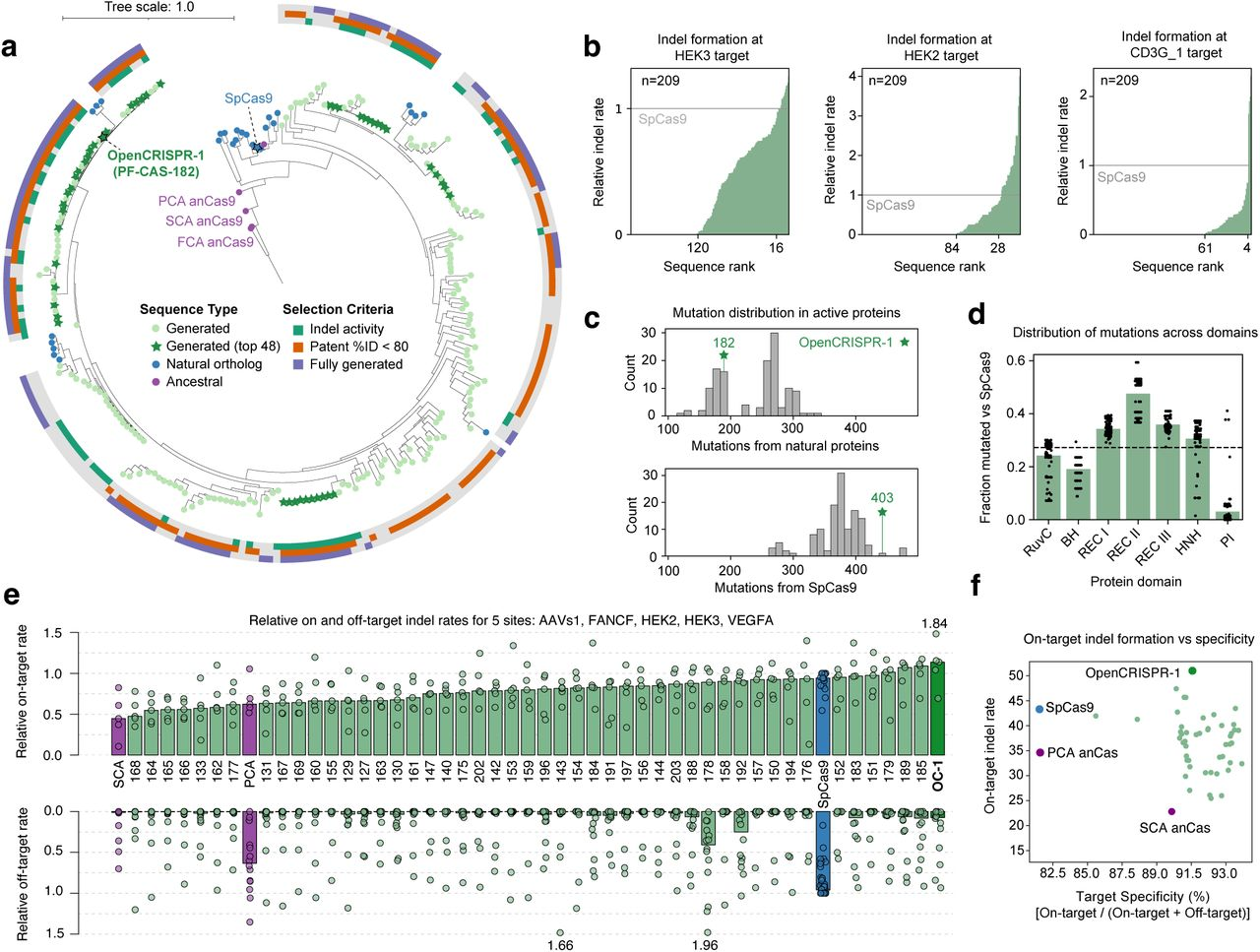
다음 파트에서는 Cas9 유사 단백질을 인간 세포에서 유전자 편집 도구로 사용할 수 있는지 평가하는 과정과 결과를 설명했습니다.
-
Cas9 유사 단백질의 인간 세포 내 테스트:
- 연구팀은 209개의 Cas9 유사 단백질 서열을 인간 코돈에 최적화하고, C-말단에 1x SV40 NLS 태그(핵으로 단백질을 유도하는 신호)를 포함시켜 인간 세포에서 발현할 수 있도록 설계했습니다.
- 이 단백질들은 HEK293T 세포(인간 신장 세포 유래)에서 SpCas9 sgRNA(특정 DNA 서열을 타겟팅하는 RNA)와 함께 공동 형질전환되어 기능을 테스트했습니다.
- 실험 결과, 이들 Cas9 유사 단백질 중 일부는 SpCas9와 유사하거나 더 높은 유전자 편집 효율을 보였습니다.
-
활성 뉴클레아제의 분석:
- 연구팀은 생성된 단백질의 활성과 SpCas9와의 비교를 통해, 어떤 단백질이 높은 활성을 보이는지 평가했습니다. 이때, AUC ROC 값이라는 지표를 사용하여 활성이 높은 효소와 낮은 효소를 구분했습니다. 여기서 HEK3라는 특정 타겟 부위에서 AUC ROC 값이 0.82로 나타났습니다. 이는 모델이 효소 활성을 예측하는 데 매우 높은 정확도를 보였음을 의미합니다.
- Cas9 유사 단백질의 서열 분석 결과, SpCas9와의 서열 유사성은 낮았지만, 이들 중 상당수가 높은 활성을 나타냈습니다.
-
PF-CAS-182: 최적의 단백질 발견:
- 연구팀은 PF-CAS-182라는 단백질을 발견했는데, 이 단백질은 온타겟(정확한 타겟 위치)에서 SpCas9과 유사한 성능을 보였지만, 오프타겟(의도하지 않은 위치)에서의 편집이 95%나 감소했습니다.
- PF-CAS-182는 기존의 SpCas9과 비교해도 서열 유사성이 낮았지만, 더 높은 활성을 보였습니다. 이 단백질은 OpenCRISPR-1로 명명되었습니다.
-
PAM 서열에 대한 특성화:
- 연구팀은 OpenCRISPR-1이 NGG PAM뿐만 아니라 비-NGG PAM 서열에서도 어떻게 작동하는지를 테스트했습니다. 결과적으로, OpenCRISPR-1은 NGG PAM을 포함한 위치에서 높은 활성을 보였지만, PAM 서열이 맞지 않는 위치에서는 활성이 크게 감소했습니다. 이는 OpenCRISPR-1이 특정한 PAM 서열에 대해 높은 특이성을 가진다는 것을 시사합니다.
주요 전문 용어 해설:
-
코돈 최적화 (Codon Optimization):
- 코돈은 DNA에서 아미노산을 지정하는 세 개의 염기 서열입니다. 코돈 최적화는 특정 생물체에서 유전자가 더 효율적으로 발현되도록 DNA 서열을 변경하는 과정을 의미합니다. 여기서는 인간 세포에서 Cas9 단백질이 효율적으로 발현될 수 있도록 Cas9 서열을 인간 코돈에 맞춰 조정했습니다.
-
1x SV40 NLS 태그:
- NLS (Nuclear Localization Signal)는 단백질이 핵으로 이동하도록 유도하는 신호입니다. SV40은 원숭이 바이러스에서 유래한 NLS 태그의 일종으로, 단백질을 세포 핵으로 운반하는 역할을 합니다. Cas9 단백질에 이 태그를 추가함으로써, 세포 내에서 Cas9이 핵으로 이동해 유전자를 편집할 수 있도록 합니다.
-
HEK293T 세포:
- HEK293T는 인간 신장 세포에서 유래된 세포주로, 연구에서 유전자 발현과 유전자 편집을 연구하기 위해 널리 사용됩니다. 이 세포주를 사용하여 Cas9 유사 단백질의 기능을 테스트했습니다.
-
SpCas9 sgRNA:
- SpCas9(Streptococcus pyogenes Cas9)은 가장 널리 사용되는 Cas9 단백질로, sgRNA (single guide RNA)는 이 Cas9 단백질이 특정 DNA 서열을 인식하고 절단하도록 유도하는 RNA입니다. sgRNA는 타겟 DNA와 상보적인 서열을 가지며, Cas9과 결합해 유전자를 편집합니다.
-
AUC ROC 값:
- AUC (Area Under the Curve) ROC (Receiver Operating Characteristic) 값은 이진 분류 모델의 성능을 평가하는 데 사용되는 지표입니다. 0.5는 무작위 분류를, 1.0은 완벽한 분류를 의미합니다. 여기서 0.82라는 값은 모델이 효소 활성을 예측하는 데 높은 정확도를 가졌음을 의미합니다.
-
NGG PAM 및 비-NGG PAM:
- PAM(Protospacer Adjacent Motif) 서열은 Cas9 단백질이 DNA를 인식하고 절단하기 위해 필요한 짧은 DNA 서열입니다. NGG는 SpCas9이 인식하는 특정 PAM 서열로, 여기서 N은 A, T, C, G 중 어떤 염기든 가능함을 의미합니다. 비-NGG PAM은 이와 다른 서열을 의미하며, Cas9 변종이 다양한 PAM 서열을 인식할 수 있도록 연구가 진행됩니다.
결론
이 부분은 Cas9 유사 단백질의 인간 세포 내 유전자 편집 성능을 평가하는 과정을 다루고 있으며, 이를 통해 연구팀은 기존의 SpCas9과 비교해도 뛰어난 성능을 보이는 OpenCRISPR-1이라는 단백질을 발견했습니다. 이 연구는 Cas9 유사 단백질이 특정 조건에서 높은 효율성과 특이성을 보이며, 특히 비-NGG PAM 서열에서도 효율적으로 작동할 수 있음을 보여줍니다.
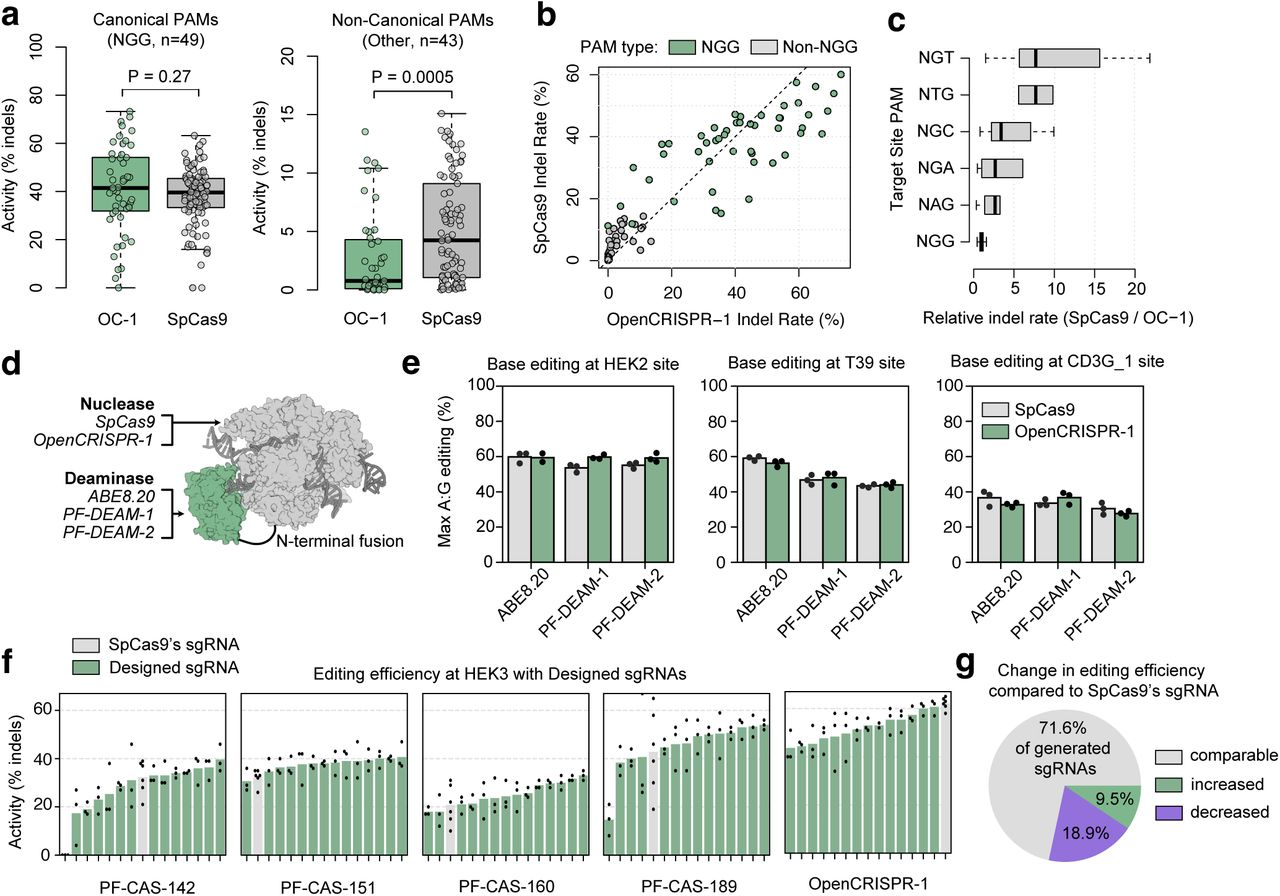
이 부분은 OpenCRISPR-1 단백질이 염기 편집(Base Editing) 시스템에서 어떻게 사용될 수 있는지를 탐구하는 연구 결과를 설명하고 있습니다. 또한, OpenCRISPR-1을 기반으로 한 합성 염기 편집 시스템의 개발과, sgRNA(가이드 RNA) 설계에 대한 추가 실험을 다루고 있습니다.
핵심 내용 요약:
-
OpenCRISPR-1의 염기 편집 시스템에서의 활용:
- 연구팀은 OpenCRISPR-1 단백질을 염기 편집에 사용할 수 있도록 니카제(nickase)로 변환했습니다. 이는 DNA 이중 가닥을 절단하지 않고 한 가닥만을 절단하는 형태로, D10A 돌연변이를 포함하여 변형되었습니다.
- OpenCRISPR-1을 아데닌을 구아닌으로 변환하는 아데노신 데아미나제(ABE8.20)와 결합시켜 염기 편집기를 만들었습니다.
- HEK293T 세포에서 이 시스템을 테스트한 결과, OpenCRISPR-1을 기반으로 한 염기 편집기가 35-60%의 높은 A-to-G 변환 효율을 보였으며, 이는 SpCas9 기반 염기 편집 시스템과 유사한 성능을 나타냈습니다.
-
합성 아데닌 데아미나제 개발:
- 연구팀은 합성 염기 편집 시스템을 설계하기 위해, 다양한 데이터베이스(UniProtKB, BFD)에서 아데닌 데아미나제(TadA) 유사 단백질을 기반으로 딥러닝 모델을 훈련시켰습니다.
- 이를 통해 PF-DEAM-1과 PF-DEAM-2라는 두 가지 합성 아데닌 데아미나제를 개발했으며, 이들 단백질은 SpCas9 또는 OpenCRISPR-1과 결합하여 높은 A-to-G 편집 효율을 나타냈습니다.
-
sgRNA 설계와 테스트:
- 연구팀은 SpCas9의 sgRNA가 OpenCRISPR-1과 같은 Cas9 유사 단백질에 최적화되지 않을 수 있다고 가정하고, 새로운 sgRNA를 설계했습니다.
- 5개의 Cas9 유사 단백질에 대해 14개의 새로운 sgRNA를 설계하고, HEK293T 세포에서 편집 효율을 테스트했습니다. 일부 sgRNA는 SpCas9의 기존 sgRNA보다 더 높은 편집 효율을 보였습니다.
- OpenCRISPR-1은 기존의 SpCas9 sgRNA와도 유사한 성능을 보였으며, 새로운 sgRNA와도 호환되는 것으로 나타났습니다.
전문 용어 설명:
-
염기 편집(Base Editing):
- 염기 편집은 DNA 이중 가닥을 절단하지 않고, 특정 염기(A, T, C, G)를 다른 염기로 변환하는 기술입니다. 이는 유전자 교정의 정확성을 높이고, 오프타겟 효과를 줄이는 데 중요한 역할을 합니다.
-
니카제(Nickase):
- 니카제는 DNA 이중 가닥 중 한 가닥만을 절단하는 효소입니다. Cas9 단백질은 원래 이중 가닥을 절단하지만, 특정 돌연변이(예: D10A)를 도입하면 니카제로 변환되어 한 가닥만을 절단할 수 있습니다.
-
D10A 돌연변이:
- D10A 돌연변이는 Cas9 단백질의 특정 아미노산(D 아스파트산)을 A 알라닌으로 변형시키는 돌연변이입니다. 이 돌연변이는 Cas9을 니카제로 전환하여 DNA의 한 가닥만 절단할 수 있게 만듭니다.
-
아데노신 데아미나제(ABE8.20):
- 아데노신 데아미나제(ABE)는 DNA의 아데닌(A) 염기를 구아닌(G)으로 변환하는 효소입니다. ABE8.20은 이러한 효소의 특정 변종으로, 염기 편집에 사용됩니다.
-
SpCas9 sgRNA 스캐폴드:
- sgRNA (single guide RNA)는 Cas9 단백질을 특정 DNA 서열로 유도하는 RNA입니다. 스캐폴드(scaffold)는 이 sgRNA의 구조적 뼈대를 의미하며, Cas9 단백질과의 결합을 돕습니다. SpCas9 sgRNA 스캐폴드는 SpCas9 단백질에 최적화된 구조를 가지고 있습니다.
-
HEK293T 세포:
- HEK293T는 인간 신장 세포에서 유래된 세포주로, 유전자 발현 실험에서 널리 사용됩니다. 여기서는 OpenCRISPR-1의 기능을 테스트하는 데 사용되었습니다.
-
루프 삽입 (Loop Insertion):
- 루프 삽입은 단백질 서열에 새로운 아미노산 루프(구조적 돌출부)를 추가하는 것을 의미합니다. 이는 단백질의 구조적 안정성이나 기능을 조절하는 데 사용될 수 있습니다.
-
gRNA 모델:
- gRNA 모델은 특정 Cas9 단백질에 맞춘 가이드 RNA(sgRNA)를 설계하기 위해 훈련된 딥러닝 모델입니다. 이 모델은 특정 Cas9 변종에 최적화된 sgRNA를 설계하는 데 사용됩니다.
결론
이 부분에서는 OpenCRISPR-1을 염기 편집 시스템에서 사용하기 위한 실험이 다루어졌으며, 높은 효율성과 특이성을 가진 합성 염기 편집 시스템의 개발이 보고되었습니다. 또한, 새로운 sgRNA 설계를 통해 Cas9 유사 단백질의 편집 효율을 더욱 개선할 수 있는 가능성이 제시되었습니다.
마무리
우선 논문의 핵심내용은 이것이 전부이며 나머지는 응용방법 및 윤리에 관한 이야기가 대부분이였습니다.
