1. Power transform(멱함수)
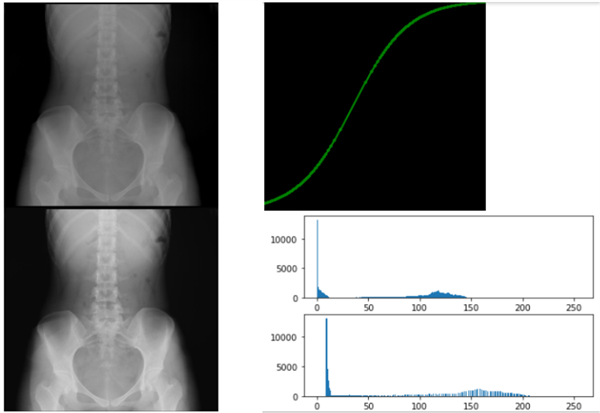
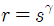 입력 영상 및 결과 영상의 범위는 0~1로 정규화 되어 있다.
입력 영상 및 결과 영상의 범위는 0~1로 정규화 되어 있다.
감마 값이 1보다 작으면 영상은 밝아지고 1보다 크면 어두워진다.
미리 LUT(look up table)을 만들어 연산을 해놓으면 프로그램을 더 효율적으로 사용 가능.
연산을 해서 LUT에 넣어주면 할때마다 power_transform을 호출할 필요가 없다.
import cv2
import numpy as np
from google.colab.patches import cv2_imshow
# 멱함수 변환 함수
def power_transform(gamma, LUT):
i = 0
for i in range(0, 256):
#0~255 -> 0~1로 만들어주고 255를 곱해서 결과값은 0~255
tmp = 255.0 * pow((i/255.0), gamma)
if tmp>255:
tmp = 255
LUT[i] = tmp
# 그래프 생성
def view_1d(mat, size, data, DX, DY, yscale):
cv2.rectangle(mat, (0,0), (256,256), (0,0,0), -1)
i=0
for i in range(0, size-1):
# data[i]~data[i+1], 즉 현재 LUT와 다음 LUT의 모든 점을 하나의 선으로 이어준다.
cv2.line(mat,(DX+i,DY-yscale*data[i]),(DX+i+1,DY-yscale*data[i+1]),(0,128,0),2)
# 2차원 컬러이미지 생성
view = np.zeros((256,256,3), np.uint8)
LUT = np.zeros(256, np.uint8) # 1차원의 LUT
gamma = 0.5
power_transform(gamma, LUT)
view_1d(view, 256, LUT, 0, 256, 1) # view에 LUT를 심어준다.
img = cv2.imread('abdomen SUPINE sens-197 highdose.bmp', cv2.IMREAD_GRAYSCALE)
image = cv2.resize(img, (256,256), cv2.INTER_AREA)
dst = image.copy()
w = image.shape[1]
h = image.shape[0]
for i in range(0,h):
for i in range(0,w):
dst[i][j] = LUT[image[i][j]] # dst 이미지 변환
cv2_imshow(image)
cv2_imshow(dst)
cv2_imshow(view)output
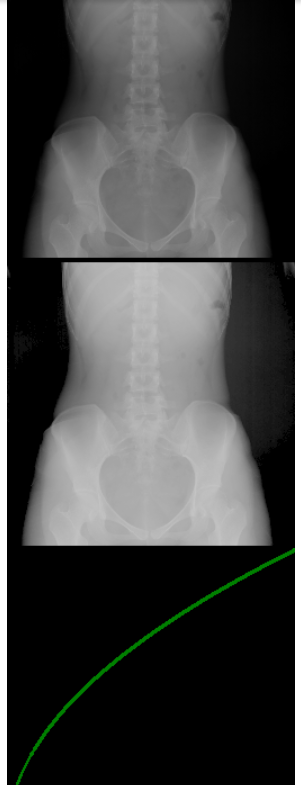
2. Sigmoid transform
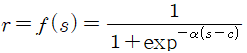 sigmoid에서 line이 급격하게 변하는 부분을 펼쳐줘서 강조할 수 있다.
sigmoid에서 line이 급격하게 변하는 부분을 펼쳐줘서 강조할 수 있다.
α: 기울기를 결정
α 크게하면 line이 급격해져서 대비가 강해진다.
import cv2
import numpy as np
import math
import matplotlib.pyplot as plt
from google.colab.patches import cv2_imshow
# sigmoid 함수 변환 함수
def sigmoid_transform(a, c, LUT):
i = 0
for i in range(0, 256):
s = i/255.0
x = math.exp(-a*(s-c))
tmp = 255.0*(1.0/(1+x)) # 0~255 -> 0~1로 만들어주고 255를 곱해서 결과값은 0~255
if tmp>255:
tmp = 255
LUT[i] = tmp
# 그래프 생성
def view_1d(mat, size, data, DX, DY, yscale):
cv2.rectangle(mat, (0,0),(256,256),(0,0,0), -1)
i=0
for i in range(0, size-1):
#data[i]~data[i+1], 즉 현재 LUT와 다음 LUT의 모든 점을 다 하나의 line으로 이어준다.
cv2.line(mat,(DX+i,DY-yscale*data[i]),(DX+i+1,DY-yscale*data[i+1]),(0,128,0),2)
# 2차원 컬러이미지 생성
view = np.zeros((256,256,3),np.uint8)
LUT = np.zeros(256,np.uint8) # 1차원의 LUT
a = 8 # 대비(숫자가 커지면 대비가 강해진다)
c = 0.4 # 밝기(숫자가 작아질수록 밝아진다)
sigmoid_transform(a, c, LUT)
view_1d(view, 256, LUT, 0, 256, 1) # view에 LUT를 심어준다.
img = cv2.imread('abdomen SUPINE sens-197 highdose.bmp', cv2.IMREAD_GRAYSCALE)
image = cv2.resize(img, (256,256), cv2.INTER_AREA)
dst = image.copy()
w = image.shape[1]
h = image.shape[0]
for i in range(0,h):
for j in range(0,w):
dst[i][j] = LUT[image[i][j]] # dst 이미지 변환
plt.subplot(211) # 2행 1열로 잡은 공간에 1번째로 지정
plt.hist(image.ravel(), 256,[0,256]) #histogram
plt.subplot(212) # 2행 1열로 잡은 공간에 2번째로 지정
plt.hist(dst.ravel(), 256,[0,256]) #histogram
cv2_imshow(image)
cv2_imshow(dst)
cv2_imshow(view)output: