차원축소
수많은 데이터의 저장공간을 절약하기 위하여 차원축소를 할 수 있다
데이터는 14.k-평균의 데이터를 활용하였다
압축하기
기본적으로 fruits_2d는 300개의 100*1000 이미지이다
print(fruits_2d.shape) # (300, 10000)
fruits_pca = pca.transform(fruits_2d)
print(fruits_pca.shape) # (300, 50)이를 50개의 이미지로 압축해보자
from sklearn.decomposition import PCA
pca = PCA(n_components=50)
pca.fit(fruits_2d)
print(pca.components_.shape) # (50, 10000)
draw_fruits(pca.components_.reshape(-1,100,100))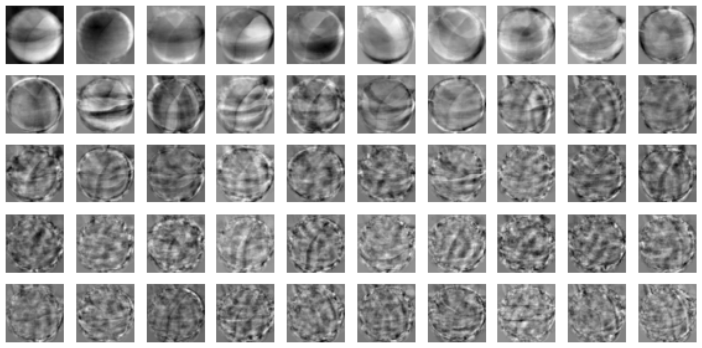
이 이미지는 일종의 데이터 셋의 특징으로 이해 할 수 있다
재구성하기
fruits_inverse = pca.inverse_transform(fruits_pca)
print(fruits_inverse.shape) # (300, 10000)
fruits_reconstruct = fruits_inverse.reshape(-1,100,100)
for start in [0,100,200]:
draw_fruits(fruits_reconstruct[start:start+100])
print("\n")print(np.sum(pca.explained_variance_ratio_)) # 0.921...
plt.plot(pca.explained_variance_ratio_)
plt.show()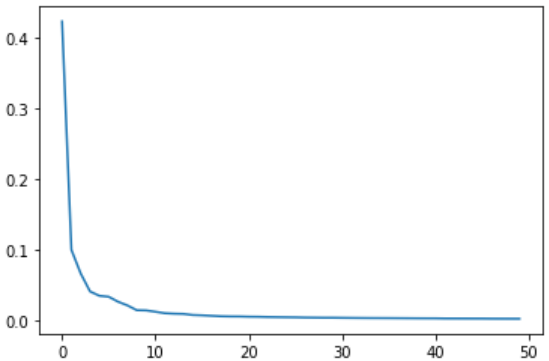
90%가 넘는 분산을 유지한다
그래프를 참고하면 처음 10개의 주성분이 대부분의 분산을 표현중이다
기타 알고리즘
로지스틱회귀에 PCA를 활용하여 차이점을 확인해보자
로지스틱 회귀 학습
from sklearn.linear_model import LogisticRegression
lr = LogisticRegression()
target = np.array([0]*100 + [1]*100 + [2]*100)로지스틱 회귀는 지도학습이므로 타겟을 설정 후 훈련을 진행하였다
훈련시간 측정
from sklearn.model_selection import cross_validate
scores = cross_validate(lr, fruits_2d, target)
print(np.mean(scores['test_score'])) # 0.996...
print(np.mean(scores['fit_time'])) # 0.461...검증점수는 97%로 매우 높으며, 수행시간은 0.94초가 걸렸다
이제 PCA를 통해 압축 후 진행해보자
scores = cross_validate(lr, fruits_pca, target)
print(np.mean(scores['test_score'])) # 1.0
print(np.mean(scores['fit_time'])) # 0.021...정확도는 100%에, 훈련시간은 0.02초로 매우 감소하였다
pca = PCA(n_components=0.5)
pca.fit(fruits_2d)
print(pca.n_components_) # 2분산이 50%인 주성분을 찾아 PCA 모델을 만들었고, 2개의 특성만으로 훈련이 가능하다
fruits_pca = pca.transform(fruits_2d)
print(fruits_pca.shape) # (300,2 )
scores = cross_validate(lr,fruits_pca, target)
print(np.mean(scores['test_score'])) # 0.99
print(np.mean(scores['fit_time'])) # 0.030...2개의 특성만으로 99%의 정확도와 0.04초의 훈련시간을 보인다
KMeans
이번에는 축소된 데이터를 통해 KMeans 알고리즘을 수행해보자
from sklearn.cluster import KMeans
km = KMeans(n_clusters=3, random_state=42)
km.fit(fruits_pca)
print(np.unique(km.labels_,return_counts=True))
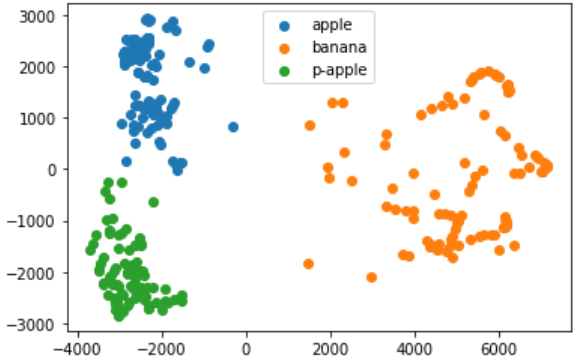
# (array([0, 1, 2]), array([110, 99, 91], dtype=int64))위 데이터를 km.labels_를 통해 산점도를 그려보았다
for label in range(0,3):
data = fruits_pca[km.labels_ == label]
plt.scatter(data[:,0], data[:,1])
plt.legend(['apple','banana','p-apple'])
plt.show