스터디때 발표를 위해 리뷰 시작!
npj digital medicine에 올라온 paper.
제목을 일단 봐보면 큰 topic은 self-supervised transfer learning 을 통해서 physiological signal을 통해 수술의 adverse event를 예측하는 주제이다.
그렇다면, self-supervised learning 에 대해 공부해야할 것이다.
self-supervised learning 참고
이 글에서도 정리를 했지만, self-supervised learning은 비교적 데이터셋이 큰 데이터로 학습을 시킨 모델을 통해서 pre-trained model을 바탕으로 dataset을 representation learning하여 다음 step에 전이 시키는 방식이므로, physiological signal을 큰 데이터셋으로 부터 pre-train 한 후, 전이학습을 통해 수술 후 adverse event를 예측했겠구나. 정도로 논문을 시작하면 되겠다.
Introduction
의료기관은 소규모 기관부터 대규모 기관까지 크기가 다양한데, 소규모 기관은 정확한 모델을 훈련하기 위한 충분한 데이터나 계산 리소스가 부족할 수 있다.
그리고, 개인정보관련해서 대규모 공개 EHR 데이터셋의 존재 가능성은 매우 낮으므로 많은 기관에서 자체적으로 성능 모델을 만들기에는 현실적인 어려움이 있음.
이러한 상황에서도 정확한 예측을 하는 방법은 transfer learning 인데, transfer learning 참고 이 transfer learning은 이미 의료 이미지와 임상 텍스트에서 성능이 잘 나왔음.
이 transfer learning 에서 이미지 혹은 시계열 데이터에서 일반화 가능한 feature를 추출하는 방법을 학습하는 deep embedding model이 유망한 것으로 알려져있다.
(신호계열의 데이터는 보통 EMA와 같은 간단한 방식으로 embedding해옴. 본 연구는 좀 더 potential한 feature를 뽑아낼 수 있는 embedding 을 하기 위한 시도를 함.)
그래서 본 논문에서는 PHASE(PhysiologicAl Signal Embeddings)라는 모델을 제시하는데
이 모델은 physiological signals에 대해 deep embedding model을 통해 임베딩한 후 예측모델으 돌리면 성능이 더 좋으며, 컴퓨팅 리소스가 풍부한 기관에서 리소스가 적은 기관으로 transfer함으로써 computation cost도 줄일 수 있다고 주장함.(즉, 데이터를 공유하지않고 모델을 공유함으로써 의료 도메인에서 활용하기 적당하였음.)
본 논문은 3개의 다른 의료기관의 데이터(2개는 수술방(AIMS),1개는 중환자실데이터(MIMIC-III))를 이용하여 transfer learning 을 평가하였다.
feature attributions을 validate하기 위한 embedding model을 self supervised approache를 통해 접근함.
input으로 15개의 physiological signal, 6개의 Static variables 를 사용하여
6가지의 output
(1. hypoxemia, 2. hypocapnia, 3. hypotension, 4. hypertension, 5. phenylephrine administration, 6. epinephrine administration)을 예측함
Upstream model(embedding model을 평가하기 위해 downstream task(GBT model)을 사용하는데, 해당 embedding model을 비교함.
1) standard embedding setting 에서 어떤 model이 제일 성능이 좋은 지 판별
2) 해당 모델(PHASE)들을 transfer embedding 하여 성능이 좋은 지 확인
3) 가장 성능이 좋았던 embedding model(next)를 fine-tuned embedding setting에서 비교
standard embedding setting 에서 PHASE가 기존 접근법보다 예측성능이 더 좋았음.(최신 machine learning model(hand engineered feartures이용한 GBT보다 4% 더 hypoxemia를 더 잘 예측했음)
또한 PHASE가 LSTM embedding model이 하나의 data set에서 feature를 추출하는데 사용되는 transferred embedding setting 에서 성능을 향상시키는 것을 확인함. -> unseen data에 대한 LSTM을 fine tunning 하면 모든 결과에서 randomly initialized model보다 빠른 convergence와 더 나은 예측성능으로 이어진다는 것을 확인함. 마지막으로 SHAPLY 이용해서 중요한 variables를 식별하여 모델을 검증함. 이로 explainable model이라는 것을 입증.
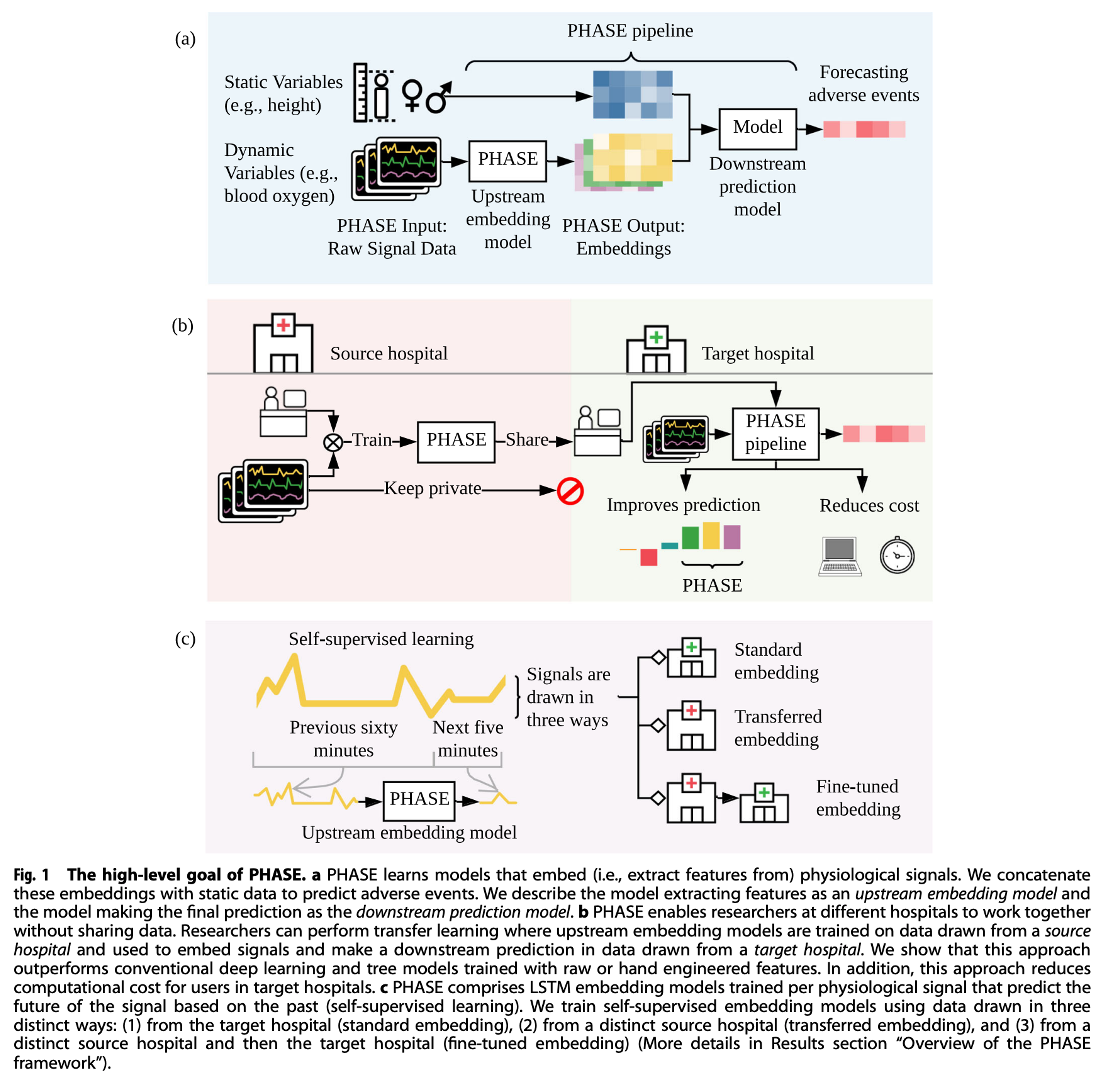
-그림1 설명-
a : PHASE는 physiological signal을 embedding 하는 모델을 학습(feature extraction)
이 embedding된 vector를 static variable과 연결하여 수술 후 부작용을 예측함.
feature extraction하는 모델을 "upstream model", 최종 예측을 수행하는 모델을 "Downstream model"로 명명함
b : PHASE를 공유(not 데이터 공유)하는 모식도.
source hospital에서 가져온 데이터에 대해 upstream embedding model을 훈련하고
target hospital에서 가져온 데이터에서 신호를 임베딩하고 down-stream prediction을 하는데 사용되는 transfer learning 을 수행할 수 있음.
이 방식이 raw data or hand-engineered feautre로 훈련된 기존의 deep learning or tree model보다 성능이 우수한 것을 확인.
c : PHASE는 physiological signal로 훈련된 LSTM embedding(self supervised learning)으로 구성됨.
데이터를 가져오는 방식은 세가지 방식이 있음
1) target hospital로부터 (standard embedding),
2) distinct source hospital로부터 (transfereed embedding)
3) from a distinct source hospital and then the target hospital (fine-tuned embeding)
Methods
1. datasets
수술실(OR) 데이터 세트는 AIMS을 통해 수집되었으며, 여기에는 static information과 분 단위로 샘플링된 physiological signal의 실시간 측정이 포함됨. 은 학술 의료 센터에서, 은 트라우마 센터에서 가져옴.
과 의 환자 분포 사이의 두 가지 현저한 차이점은 1. 성별 비율(학계 의료 센터의 여성 57% 대 외상 센터의 38%)과 2. 응급 상황으로 분류되는 ASA 코드의 비율(응급 상황 대 7.65%)임. 15.31%).
은 PhysioNet의 공개적으로 사용 가능한 MIMIC 데이터 세트에서 추출한 하위 샘플 버전으로, 매사추세츠주 보스턴에 있는 중환자실(ICU)에서 얻은 데이터가 포함되어 있음.
데이터에는 여러 생리적 신호가 포함되어 있지만 많은 생리적 신호에서 누락이 상당했음. 따라서 분 단위 SAO2 신호만 사용함.
세 가지 데이터 세트 모두에서 신호 특성의 나머지 누락된 값은 평균으로 imputation 하였고, 각 feature는 neural network 학습을 위한 단위 평균과 분산을 갖도록 standardized함.
2. set-up
입원 분포 : P (아래와 같이 LOS의 분포를 P로 표현)
hospital stay를 P ~ P 로 표현 할 수 있음
그 후,time point를 t ~ (1, ⋯ , len (P)).로 그리고 t와 P를 이용하여 i 샘플을 정의한다.
3. variables
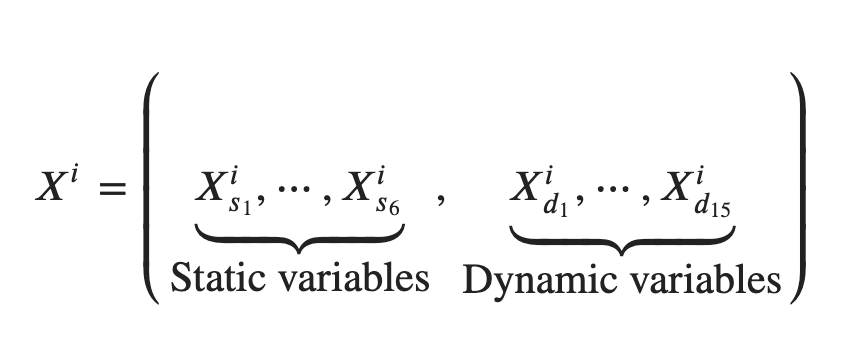
static variable : 입원기간 내내 일정하고 P에 의해서만 결정됨
dynamic variable : 시간이 지남에 따라 변하고 P or t 에 의해서 결정됨
각 sample은 (i는 P 및 t 에 따라서 결정됨) 변수를 static/dynamic으로 분할함.
-
Static variable : Height, Weight, ASA Code, ASA Code Emergency, Gender, and Age.
-
dynamic variable :
● SAO2—Blood oxygen saturation
● ETCO2—End-tidal carbon dioxide
● NIBP[S/M/D]—Non-invasive blood pressure (systolic, mean, diastolic)
● FIO2—Fraction of inspired oxygen
● ETSEV/ETSEVO—End-tidal sevoflurane
● ECGRATE—Heart rate from ECG
● PEAK—Peak ventilator pressure
● PEEP—Positive end-expiratory pressure
● PIP—Peak inspiratory pressure
● RESPRATE—Respiration rate
● TEMP1—Body temperature
● PHENYL—Whether phenylephrine was administered. We only use this
as an output variable and not as an input.
● EPINE—Whether epinephrine was administered. We only use this as an
output variable and not as an input.
dynamic variable을 인덱싱 하기 위해서 이러한 annotation으로 표현하였고 해당 dynamic variable은 b-a 차원임을 수식에서 알 수 있다.
4. outcomes
binary outcomes로 outcomes를 설정함.
부작용 이벤트는 앞으로의 5분후의 physiological signal() 의 함수로서 (g(⋅)) 로 정의.

5. embeddings(i.e. features)
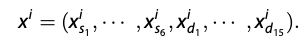
는 가 embedding 된 것을 annotation 한 것.
static variable은 embedding 거치지 않고, 그대로 사용하므로 =
dynamic variable을 함수 를 통해 과거 60분 간의 physiological signal variabled을 embedding 하여 로 annotation
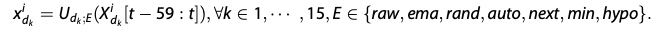
downstream model의 경우 static variable를 그대로 사용했음(static variable에서 feature extraction 시행하여도 성능에 큰 차이 없었음)
그러나 LSTM downstream model의 경우 normalize함
6. LSTM embedding
각 physiological variable()로부터 embedding vector를 잘 extract하기 위해서
source hospital ()에서 훈련한 per-signal neural networks (LSTMs)을 사용함.
200개의 node의 embedding dimension과 final time step의 embedding을 활용함. LSTM( ) 은 각각의 physiological signal을 60분 이전의 signal()를 input으로 하여 loss function(dependent on the embedding type E)을 최소화하도록 훈련한다.
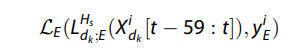
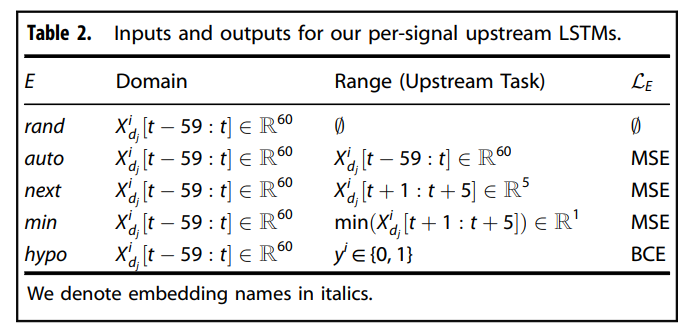
위의 표는 PHASE의 self-supervised label인 next, min, hypo 를 포함한 upstream embedding model LSTM을 훈련하는데 사용한 서로 다른 task를 표현함
꼼꼼히 들여다보면,
1) rand는 untrained LSTM with random weight. (즉, upstream model 훈련하지 않음)
2) auto는 auto-encoder 모델로 60차원 그대로 임베딩됨.
3) next 앞으로의 5분간의 신호를 embedding 해내므로 5차원의 임베딩을 하고,
4) min은 가장 최소값이므로 1차원,
5) hypo는 traditional supervised approach to transfer learning where the embedding model has the same output as the downstream prediction model이라 binary outcomes를 도출해낸다.
이러한 upstream model의 임베딩 과정을 로 annotation 가능.
이 식의 은 의 지난 60분의 L의 final hidden layer에 mapping 하는 함수를 얻기 위해 L의 출력 레이어를 제거하는 것을 의미.
는 source hospital()의 를 이용하여 LSTM을 convergence하도록 위 표에 맞는 output으로 train하는 notation이다.
neural networtk embedding 접근 방식과 관련된 feature를 설명하기 위해 network의 output layer를 제거하고 네트워크의 final hidden layer를 사용하여 각 신호를 embedding함. 이 과정을
의 식으로 표현가능하다.
여기서 h는 network L의 output layer를 제거하는 함수이고, 200의 의미는 hidden node 갯수임.
7. Downstream prediction model
Downstream model (D)는 다양한 유형의 embedding model을 평가하는 데 사용됨.
모델 D는 Target Hospital 으로 부터 추출한 embedded sample 에 대해 훈련됨.
D는 부작용을 예측하기 위해 binary cross entropy loss를 최소화함.
이때 부작용 outcome()는 physiological signal의 미래 5분의 함수에 의해 정의됨 (위에 참고)
8. dynamic embedding
Dynamic variable의 경우 1) 사용할 signal의 양 2) signal(X_{d_j}^i$)을 embedding 하는 방법 을 먼저 정함.
그래서 signal을 얼마나 각 모델에게 줄 것인지와 어떻게 embedding 할 지에 대해 먼저 정함.
1) 사용할 signal의 양
공정한 비교를 위해서 모든 모델에 결과 이전의 60분에만 access 하도록 함.
2) signal()을 어떻게 embedding 할 지에 대해서 결정해야했는데,
보통은 EMA와 같은 비교적 간단한 embedding을 함. 따라서, 비교를 위해
1) 60mins을 그대로 사용 (raw)하여 embedding하는 모델을 로 annotation.
로 embedding 함. 해당 annotation 을 보면 알겠지만, 임베딩 된 는 60차원이다.
2) exponential moving average and variances을 이용한 (ema) :
9. Transferred embedding
- target dataset() : embedding variable에 대한 down stream predict model을 훈련하는 domain
- source dataset() : upstream embedding model을 훈련한 domain
Transference experiment로 로 케이스를 나누어 실험을 진행함.
두 케이스 모두 인데, 에서 embedding 파트를 훈련하고 로 test만 수행하는 것이다.
- : H_s=OR_0이면 H_t = OR_1 임.
- : SAO2에 대한 LSTM embedding model의 source domain 은 , 나머지 model의 source domain은 target domain과 같음() (d/t MIMIC데이터에 SAO2 제외하고는 결측치가 너무 많아서)
10.Fine-tuned embedding
fine-tunning approach denote =
fine-tunning은 OR dataset에서 고려되는 접근법인데,
target dataset ()에서 예측 task를 수행하고 싶은데, 다른 데이터셋()에서 embedding 파트를 훈련 후, 다시 target hospital에서 target dataset으로 좀 더 다듬은 후 예측 수행하는 것.
이 과정을 로 표현 가능.
fine-tunning을 위해 에서 추출한 sample(target dataset와 다름!!)로 훈련된 LSTM을 이용하여 LSTM을 initialize한 후 OR_0에서 추출한 sample에 수렴할 때 까지 훈련하는 것.
에 대한 fine-tuning approach의 dynamic variables의 feautre는 아래와 같다.
11. Jointly Trained Upstream Model
본 연구는 signal마다 embedding한 것이 jointly trained upstream model()보다 성능이 좋았음을 확인하기 위해 모든 physiological signal을 다음 5분을 예측하기 위해 LSTM을 최적화하였음. 해당 과정을 아래의 수식으로 표현 가능.
: jointly trained upstream model(LSTM)
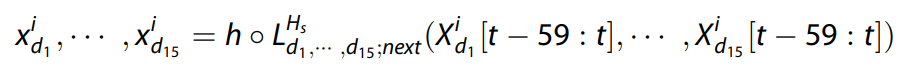
12. Local Feature Attributions
explainable model을 위해, complex tree-based models대한 interventional conditional expectation set function와 함께 exact Shapley values을 제공하는 Interventional Tree Explainer를 활용함.
Shapley value는 의 each feature가 single down stream prediction 에 얼마나 기여했는지 나타내는 local feature attributions 으로 사용됨.
- Positive attribution은 일반적으로 feature가 모델의 출력을 증가시킨다는 것을 의미함(risk of adverse events)
- Negative attribution은 일반적으로 feauture가 출력을 감소시킨다는 것을 의미.
efficiency를 유지하기 위해 local feature attributions을 summation 하는데, 이는 Saxioms Shapley values satisfy 중 하나임. efficiency는 특정 표본에 대한 속성이 model의 예측과 baseline(기준선)에 대한 average model output간의 차이를 sum up한다고 느슨하게 기술함. 효율성은 local feature attribution이 이 모델의 출력(log-odds, probability-space 등)과 대략 같은 척도에 있음을 암시하기 때문에 desirable함. 특정 신호에 대한 attribution을 평균화하는 경우, 해당 attribution은 더 이상 효율성을 만족시키지 못할 것이고 신호에 대한 속성은 평균이 아닌 static attribution(높이, 무게 등)에 대한 속성과는 다른 척도가 될 것이다. efficiency를 보장하기 위해 대신 dynamic variable(생리적 신호 기능)에 대한 attribution을 sum over하여 static feautre에한 attribution을 비교할 수 있도록 함.
Results
1. Five perioperative outcomes from three hospital datasets
surgical morbidity와 관련된 중요한 outcomes를 예측하는 것에 관심있음.
1. hypoxemia (=low blood oxygen level)
2. hypocapnia (=low blood carbon dioxide)
3. hypotension (=low blood pressure)
4. hypertension (=high blood pressure)
5. phenylephrine : hypotension시 자주 사용되는 약물
6. epinephrine : local anesthetics의 additive & reduce bleeding위해 사용
이러한 outcome으로 methodology를 평가하기 위해서 서로 다른 세기관에서 data를 사용함.
#data source에 대한 statistics
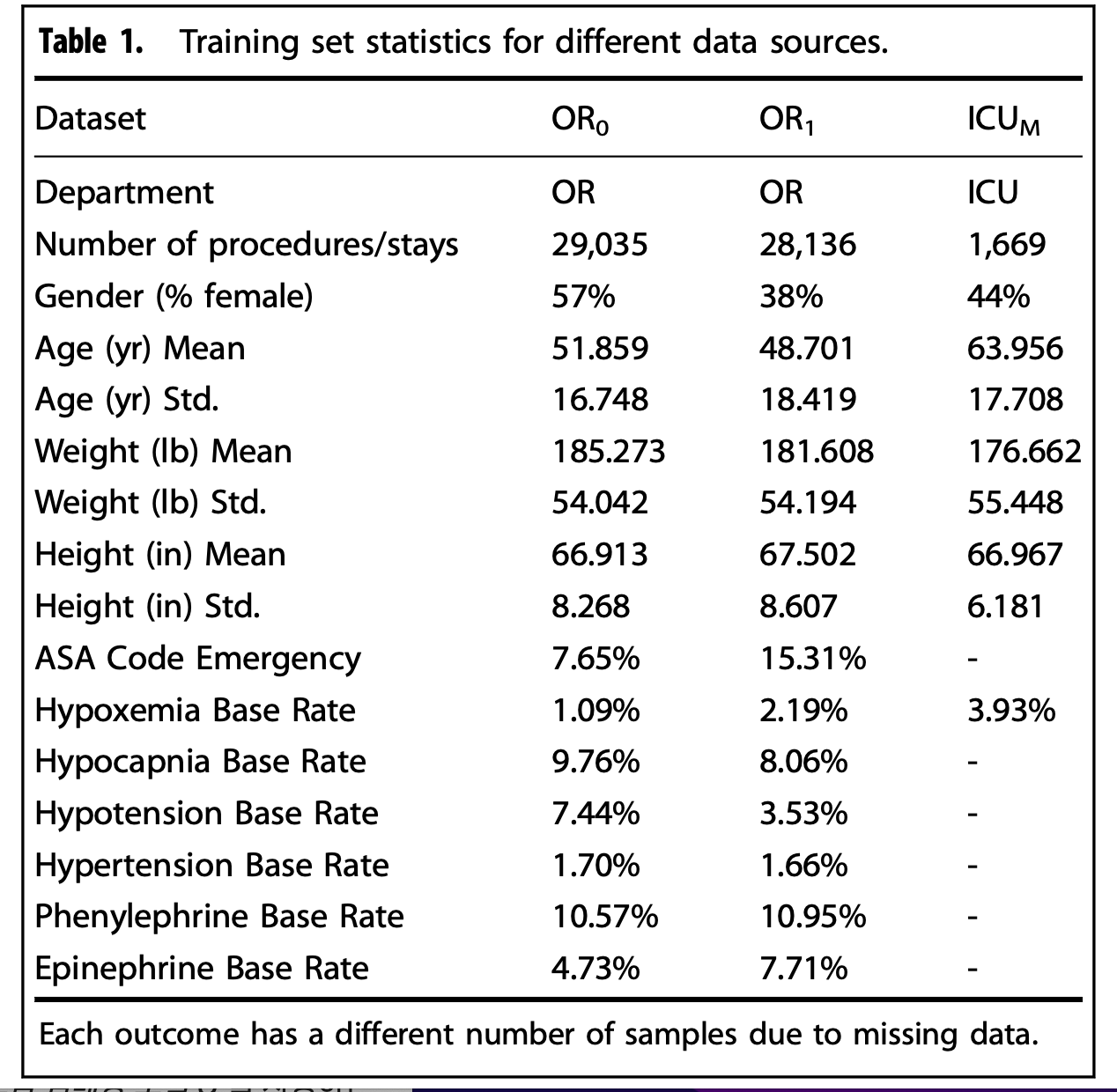
위 표를 보면, ICU(M)은 MIMIC-III의 중환자실 데이터고 OR0,OR1은 각 다른 의료기관의 수술방 데이터이다. MIMIC-III 데이터는 SAO2만 관측되어서 hypoxemia base rate 만 결과가 있다.
OR0,1의 데이터에서 static variable에서 gender, ASA code를 제외하고는 데이터의 분포가 비슷함을 알 수 있다. 또한, 각 6가지의 예측 output에 대한 Base Rate를 살펴보면, hypotension과 epinephrine을 제외한 요소들은 비슷하거나 1%차이였다.
평가 metric으로 area under a precision recall curve 사용하였다. (=average precision)
2. Overveiw of the PHASE framework
(용어 정리)
PHASE : approach to embed physiological signals.
U :(=upstream embedding models) source hospital dataset(Hs)의 physiological signal로 부터 train한 model
D : (=downstream prediction model) 모델 D의 input은 static variable과 앞선 U로 부터 나온 embedding된 벡터(embedded physiological signals)임. 모델 D는 Ht(target Hospital)에서 훈련함.
본 연구의 모델을 3가지 방식으로 평가하는데
1) standard embedding setting
: target 병원(예측 task를 하고자 하는 병원)에서 embedding 파트를 훈련시켜서 target 병원 데이터로 test를 수행하는 경우
2) transferred embedding setting
: source 병원에서 embedding 파트를 훈련해서 target 병원데이터로 test만 수행 (보통 target hospital의 resource가 부족한 경우에 target hospital 보다 규모가 큰 source hospital에서 embedding 훈련)
3) fine-tuned embedding setting
: source 병원에서 embedding파트를 훈련후, target 병원에서 target병원데이터로 조금 더 다듬은 후 target 병원데이터로 test 수행
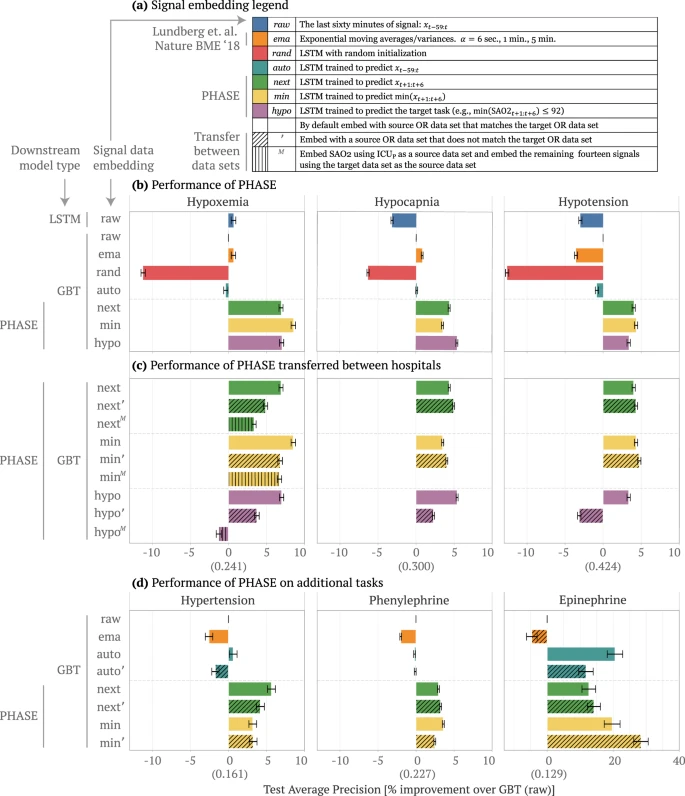
per-signal upstream embedding의 modeling decision의 advantages
1) per-signal embedding models은 the raw signals or hand-engineered signal features로 훈련한 downstream prediction model보다 성능이 뛰어남.
2) 모든 신호로 훈련한 single embedding model보다도 per-signal embedding models의 성능이 뛰어남.
3) per-signal embedding models이 heterogeneous setting(source hospital의 변수가 target hospital에서의 변수가 다른 상황)에서도 잘 작동함.
embedding 설명
1. raw, ema : 딥러닝 모델 아님
1) raw는 그냥 원본 신호 그 자체
2) ema는 exponential moving average (= 원 신호를 smoothing 한 데이터)
- Hs의 데이터에서 훈련되는 LSTM의 final hidden layer로 사용되는 embeddings
3) rand : random weight로 untrained LSTM에 이용됨.
4) auto : input을 autoencode하기 위해 훈련된 LSTM (unsupervised approach)
5) next & min : (self-supervised : the LSTM outputs are drawn from the same physiological signal variable as the input, but are taken from different parts of the signal)
6) hypo : embedding model이 downstream predict model(hypoxemia, hypocapnia, hypotension)과 같은 동일한 output을 가지는 tranfer learning 에 대한 traditional supervised approach.1) next : next 5 min of a particular signal을 예측하도록 훈련된 LSTM에 사용
2) min : next 5 min of a particular signaldml minimum을 예측하도록 훈련된 LSTM에 사용
3. comparing approaches to embed physiological signals
이 파트에서는 standard embedding setting 에서 각각의 임베딩 방식을 비교함
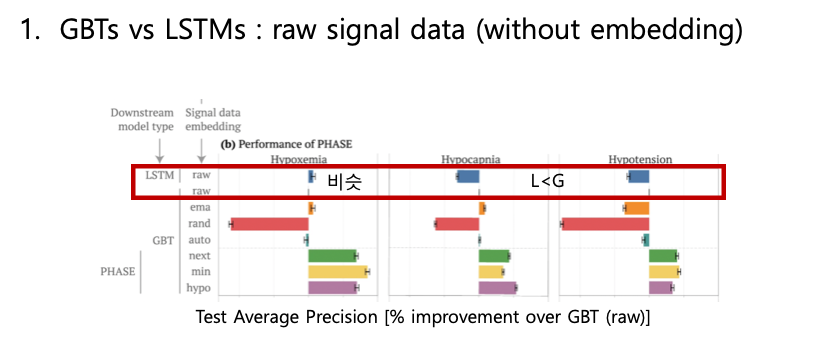
이 그림을 보면 GBT raw는 LSTM raw와 비슷하거나 더 나은 성능을 보임.
사실, LSTM은 time series data의 패턴을 잘 챕쳐하고, GBT는 static variable을 더 잘 capture함을 착안하여 "LSTM을 사용하여 physiological signal을 embedding 하고 GBT를 이용하여 static variable과 embedding vector를 사용하여 최종 예측을 수행하는 PHASE모델을 제안하게 됐다.
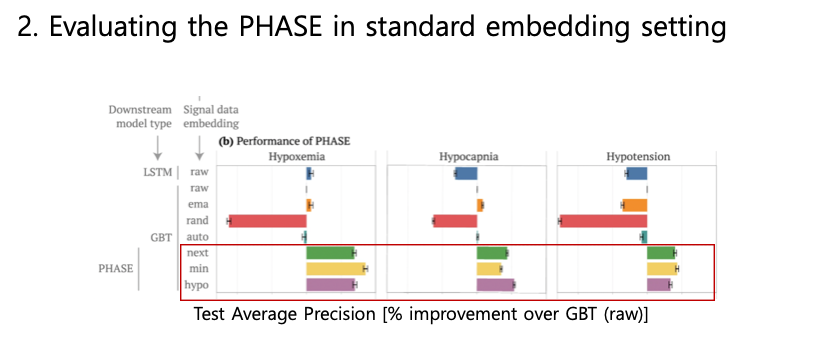
이 그림을 보면 ema, auto는 눈에 띄게 raw에 비해서 성능이 좋거나 나쁘지 않고, rand LSTM은 엉망이었음(🤫). PHASE의 next/min/hypo는 성능이 전반적으로 다 좋았다. 그런데 min/hypo는 upstream embedding model의 outcomes를 down stream predict model의 결과와 비슷하게 도출해냈지만(의미론적으로도 min,hypo는 최저값, 임계값보다 아래라는 의미로 outcome과 비슷한 결과로 도출), 성능이 지속적으로 next처럼 좋지 못했음. 그러한 이유로 앞으로는 next/min을 비교하고 fine tunned embedding 비교에서는 next로만 비교함.
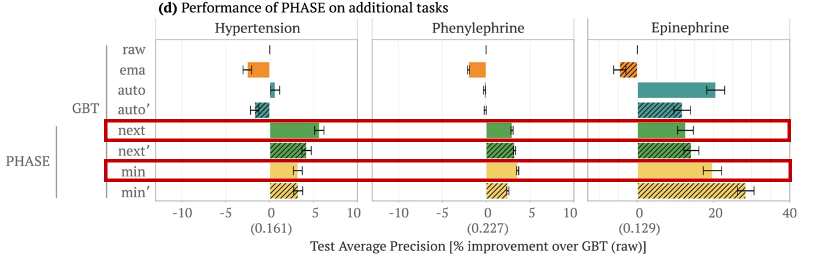
이 그림을 보면 이해가 쉬울텐데, min은 hypertension(고혈압)을 예측할때에는 성능이 안좋았는데 이는 앞에서 말했던 min(최솟값)의 의미적 유사성때문으로 간주 가능. 그런데 phenylephrine에서는 왜 성능이 더 좋았느냐? phenylephrine는 보통 저혈압일때 투약하는 약물이라서 min과 관련이 있어서..ㅎㅎ
Epinephrine 예측의 경우에는 전체적으로 모든 임베딩 모델이 성능이 좋았는데 이건 epi 관련한 sample size가 적어서 그런것 으로 추정했음.
4. Evaluating upstream embedding models on unseen data
Transfer embedding setting하에서 비교한 실험임. (앞의 method를 잘 읽고 오시길 바랍니다.🙃)
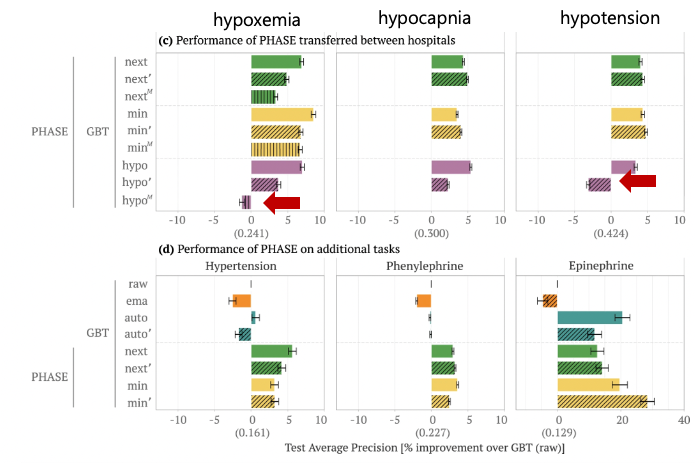
min과 next는 ‘ M 모두 어떤 outcome에서든지 raw GBT 보다 성능이 좋았다.
하지만, hypo’는 hypoxemia, hypocapnia에서는 성능이 더 좋았으나, hypotension에서는 성능이 낮았음.
게다가 은 hypoxemia에서 raw GBT 보다 성능이 낮았는데, 이것은 LSTM embedding model output이 supervised learning outcome(hypo)가 다른 데이터셋에서는 self-supervised approach처럼 일반화 시키지 못한다는 것을 알 수 있다.
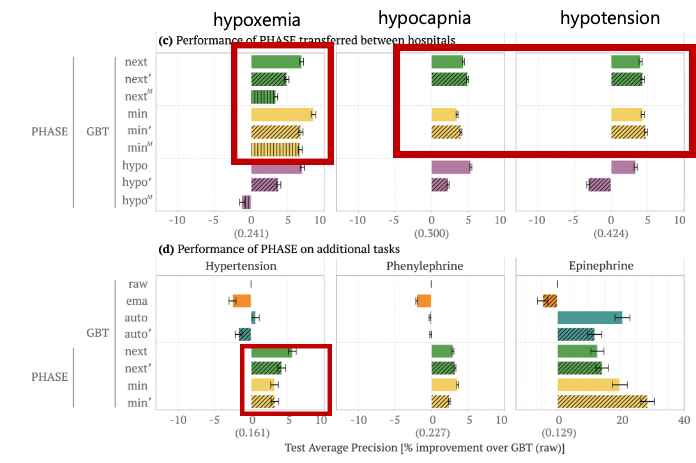
', M vs standard emedding : next/min은 hypocapnia, hypotension은 성능이 거의 비슷함. But, hypoxemia나 hypertnesion은 병원 데이터 간의 차이때문에 성능이 감소한 것이라고 추정.
그래도 transfer learning 을 통해서 리소스가 적은 병원에서도 비용이 덜 들면서 예측을 할 수 있다는 것을 알 수 있음.
transfer learing에서 고려해야 할 사항이 두가지가 있는데,
1) 본 연구는 GBT downstream model을 사용하는 것에 focus한 것임. (down stream model을 MLP와 GBT를 비교하였고 GBT의 성능이 더 좋아서 downstream model로 채택함)
2) per-signal LSTM embedding model은 모든 signal을 함께 훈련한 single LSTM보다 성능이 뛰어나는 것이었음. 게다가 per-signal embedding 이다보니 variables이 source hospital 과 target hospital이 정확하게 match하지 않아도(feature heterogeneity에서) 작동하는 이점이 있었다. Per-signal LSTM embedding model은 end user가 그들의 기관에서 가능한 신호에 상응하는 모델을 고르고 선택하기 때문에 heterogeneous setting 에서도 작동함.
5.Fine-tuning upstream embedding models imporves performance and reduces computational cost
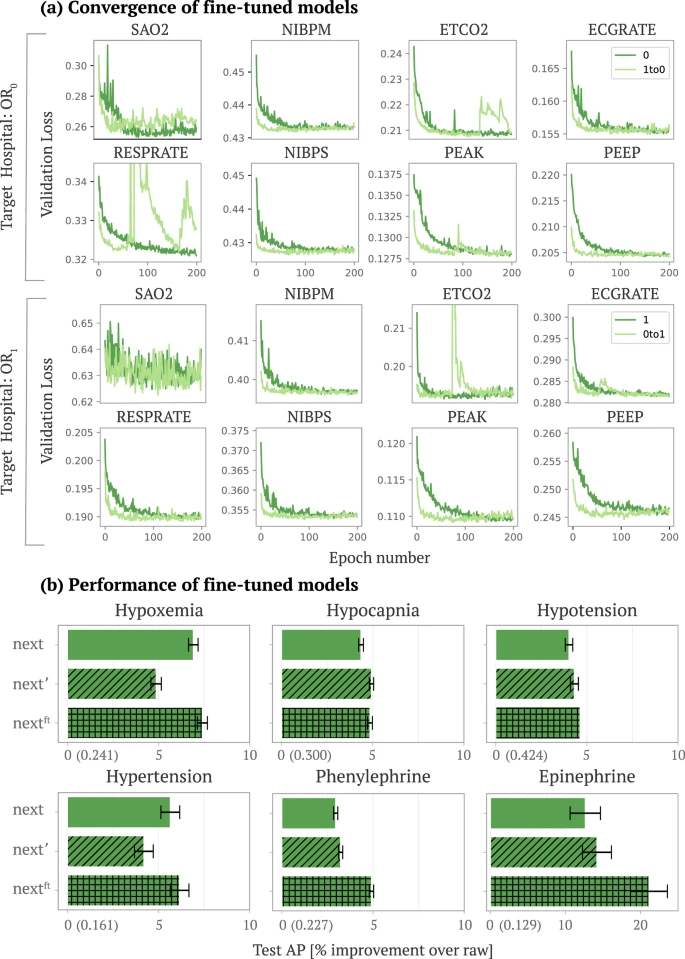
Standard embedding model이 hypoxemia, hypertension에서 성능이 더 좋은 것을 대안하기 위해 fine-tunning embedding approach를 제안했음.
그림 b를 보면 fine tuned embeeding setting에서 standard or transfer보다 성능이 훨씬 더 좋은 것을 알 수 있음.
그림 a에서는 짙은 녹색 : randomly initailized LSTM/ 연두색 : target hospital로 부터 best model의 weight을 이용한 모델의 epoch 수에 따른 convergence를 나타내고 있음. 딱 봐도 연두색라인이 convergence 속도가 더 빠른 것을 알 수 있음.
이 결과들은 end users가 fine-tune PHASE LSTMs가 더 적은 계산비용으로도 좋은 성능을 낼 수 있음을 시사함
6.Validating models with local feature atrributions
summary plot을 사용하여 downstream GBT model에서 사용된 key variable을 요약함.(그림4)
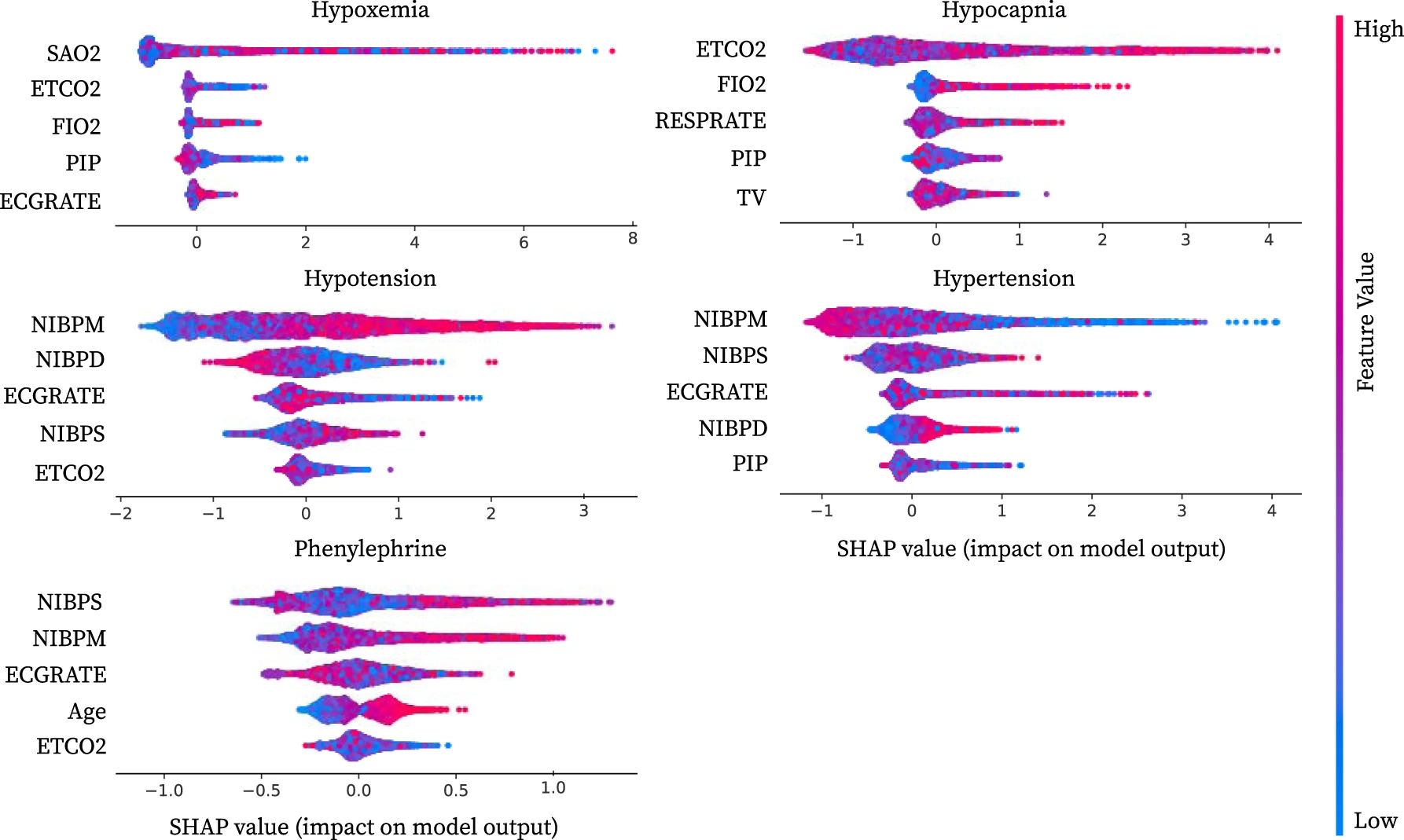
그림4 설명
target dataset ()에서 next embedding 으로 훈련된 GBT 모델의 가장 중요한 상위 5개의 변수에 대한 local feautrue attribution summary plot이다.
각 변수의 속성을 얻기 위해서 Interventional Tree Explainer을 사용하여 각 GBT를 설명함.
이것은 15개의 physiological variable(각 200차원의 embedding)과 6개의 static variable에 대한 next embedding에 대한 attribution을 제공. 특정 physiological variable의 중요성을 얻기 위해서 embedding attribution을 합산함. attribution을 합산하고 나면 axiom of efficiency를 유지할 수 있음. x축에서는 모델 출력에 대한 변수의 누적 영향을 나타내는 aggregated attribution을 나타냄. 포인트의 색상은 static variable에 대한 feature값이거나 주어진 physiological variable에 대한 모든 next embedding의 합임.
이 plot에서 각 포인트는 단일 sample에 대한 feature의 중요도를 나타내며, x 축은 feature가 모델의 output에 미치는 영향을 나타내고, 색상은 기능의 값을 나타냄. PHASE next embedding에 대해 훈련된 GBT model을 각 변수 측면에서 설명하는데에 중점을 둠. 왜냐하면, embeddings는 대부분의 outcome에서 서능이 좋았기 때문. 그림의 색은 embedding model에 따라 embedding values가 임의로 양수 혹은 음수일 수 있기 때문에 자연스럽게 해석할 수 없는 single signal variable (200개의 추출된 feature)과 관련된 모든 feautre의 합이다.
embedding model을 training하는 standard approach 는 모든 signal variable을 single model의 input으로 사용됨. 각 embedding의 dimension은 동시에 multiple signal에 따라 달라지기 때문에 이러한 접근은 해석하기가 더 어렵다. PHASE에서와 같이 per-signal embedding model을 사용하면 각 embedding이 single physiological signal variable에 의존하는 것으로 명확하게 해석가능함.
5개의 oucomes 모두에 대한 next embedding에 대해 훈련된 모델은 이전 문헌에 대해서 중요한 변수를 검증함.(그림4)
Discussion
- LSTM을 사용한 표준 임베딩이 다운스트림 모델(GBT 및 MLP)의 성능을 향상시킨 실험 진행한 토대로 PHASE model을 통한 생리적 신호 임베딩이 여러 outcome에서 효과적임.
- PHASE는 transferred 임베딩 설정에서도 거의 똑같이 잘 작동하며, 실제로 fine-tunning하면 무작위로 초기화된 모델보다 더 잘 작동. 이는 사전 훈련된 모델을 공유함으로써 계산 요구 사항 및 예측 성능 측면에서 다운스트림 모델을 개선할 수 있음.
- ICU 데이터()에 대해 훈련된 임베딩 모델이 놀라울 정도로 잘 수행되었음을 발견
-> 이 두 결과(2,3) 모두 PHASE에서 성능 개선이 adverse event에 대해 유사한 분포가 반드시 있기보다는 미래 신호에 대한 self-supervision 때문인 것을 알 수 있음.
- PHASE는 각 신호 변수에 대해 독립적으로 훈련된 LSTM을 사용함으로써 성능도 우수했을 뿐만 아니라 데이터셋의 이질성을 해결하기 위해 소스 병원에 대해 다르지만 겹치는 변수를 가진 대상 병원은 둘 다 가지고 있는 변수에 대한 임베딩 모델을 사용할 수 있음.
#limitaions
모델 공유가 데이터 공유보다 적은 정보를 나타냄.
우리 데이터의 또 다른 한계는 임베딩 모델이 분당 한 번 샘플링된 생리적 신호에만 적용된다는 것.
개인적인 느낀점.
큰 저널의 논문, 그리고 딥러닝 관련 논문을 처음 읽어본 것인데 시간이 참 오래걸렸다. 여러 사람들의 도움덕분에 이해할 수 있었다.
이 논문은 딥러닝의 모델을 개발 및 조정해서 좀 더 나은 성능을 보여주는 모델 개발의 논문이라기보단 의료환경이라는 도메인에 대한 이해도가 높아서 그런지 해당 모델을 실제 의료 현장에 사용할 수 있도록 많은 고민이 엿보였던 논문인 것 같다. (특히 리소스가 부족한 병원에서 사용하도록 노력한 시도..멋져..)
appendix까지 매우 꼼꼼히 읽었어야해서 좀 힘들었지만..? 연구가 어떻게 진행되고 얼마나 많은 실험을 하는 지 알게 됐고 다소 내 미래에 대한 두려움에대한 생각이 0.0000000000001초 들었다. (왜냐면, 난 이게 재밌어서 두렵지만 흥미로워 ㅎㅎ)
논외로, 이 논문을 읽기 위해 이수인교수님의 유튜브 강의들을 여러번 봤는데 교수님 멋있는 것 같다..이 세상에 왜이렇게 똑똑하고 멋있는 사람이 많은지..ㅎㅎ
