https://89douner.tistory.com/298
https://www.youtube.com/watch?v=fWmRYmjF-Xw&t=100s
위 자료를 참고했다.
한동안은 위 유튜브 영상들의 과정을 따라가면서 공부할듯!
사용 데이터셋
- ISBI 2012 EM segmentation Challenge (membrane 데이터셋)
- 위 데이터셋은 규모가 작아 github에 업로드 되어있다.
데이터셋 다운로드
- https://github.com/alexklibisz/isbi-2012
- 위 깃헙에서 zip 파일로 다운받아 data 폴더 내 파일들을 사용하면 된다.
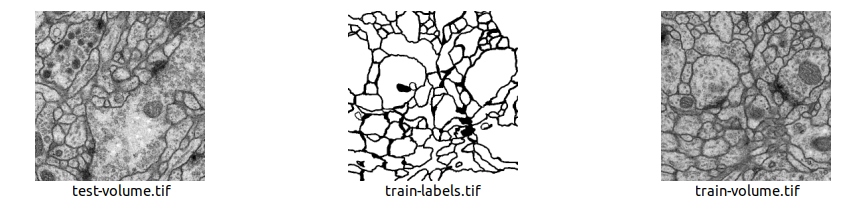
data 폴더 내부에 위와 같은 파일 세 개가 존재한다.
- train-volume.tif
- training을 위한 input data
- train-label.tif
- training을 위한 label image data
- test-volue.tif
- test image data

이미지 속성을 보면, 각각 512x512 사이즈 이미지 30장으로 이뤄진 것을 알 수 있다. (8-bit grayscale image)
이미지에서 까만 선 부분이 세포막에 해당한다.
데이터셋 재구축
위에서 봤다시피, 하나의 tif 파일에 30개의 개별 이미지가 들어있었다. 이 파일들에서 개별 이미지를 추출하는 과정을 거칠 것이다.
참고한 영상의 과정을 따를 것이므로, 경로도 참고 글과 똑같이 설정해준다.

이미지 전처리 코드 작성
개별 이미지로 나눠 주는 코드를 작성해준다. train하기 쉽도록 데이터 형태를 바꿔주는 과정에 해당한다.
# 필요한 패키지 import
import os
import numpy as np
from PIL import Image
import matplotlib.pyplot as plt
# 데이터 불러오기
dir_data = os.path.join(".", "data")
name_label = "train-labels.tif" # label, input 파일 이름
name_input = "train-volume.tif"
img_label = Image.open(os.path.join(dir_data, name_label))
img_input = Image.open(os.path.join(dir_data, name_input))
ny, nx = img_label.size # label 이미지의 사이즈 알기 위함
nframe = img_label.n_frames # 레이블의 프레임 수
##
nframe_train = 24
nframe_val = 3
nframe_test = 3
# 24, 3, 3개의 프레임을 각각 train, val, test data로 사용
# 각 데이터셋이 속해있는 디렉토리 경로 변수 지정
dir_save_train = os.path.join(dir_data, "train")
dir_save_val = os.path.join(dir_data, "val")
dir_save_test = os.path.join(dir_data, "test")
# train, validation, test 데이터 디렉토리 생성 코드
if not os.path.exists(dir_save_train):
os.makedirs(dir_save_train)
if not os.path.exists(dir_save_val):
os.makedirs(dir_save_val)
if not os.path.exists(dir_save_test):
os.makedirs(dir_save_test)
##데이터 저장
# 랜덤하게 데이터 분배하기 위한 코드
id_frame = np.arange(nframe)
np.random.shuffle(id_frame)
# trainset 저장
offset_nframe = 0
for i in range(nframe_train):
img_label.seek(id_frame[i + offset_nframe])
img_input.seek(id_frame[i + offset_nframe])
label_ = np.asarray(img_label)
input_ = np.asarray(img_input)
np.save(os.path.join(dir_save_train, "label_%03d.npy" % i), label_)
np.save(os.path.join(dir_save_train, "input_%03d.npy" % i), input_)
# validation set 저장
offset_nframe += nframe_train
for i in range(nframe_val):
img_label.seek(id_frame[i + offset_nframe])
img_input.seek(id_frame[i + offset_nframe])
label_ = np.asarray(img_label)
input_ = np.asarray(img_input)
np.save(os.path.join(dir_save_val, "label_%03d.npy" % i), label_)
np.save(os.path.join(dir_save_val, "input_%03d.npy" % i), input_)
# test set 저장
offset_nframe += nframe_val
for i in range(nframe_test):
img_label.seek(id_frame[i + offset_nframe])
img_input.seek(id_frame[i + offset_nframe])
label_ = np.asarray(img_label)
input_ = np.asarray(img_input)
np.save(os.path.join(dir_save_test, "label_%03d.npy" % i), label_)
np.save(os.path.join(dir_save_test, "input_%03d.npy" % i), input_)
##시각화
plt.subplot(121)
plt.imshow(label_, cmap="gray")
plt.title("label")
plt.subplot(122)
plt.imshow(input_, cmap="gray")
plt.title("input")
plt.show()
# label은 input의 segmentation map. 흰 부분이 1, 검은 부분이 0
데이터셋 재구축 결과

요런 식으로 train, val, test set이 각각 나뉘게 된다.
그나저나.. test-volume 파일이 있는데 test set을 굳이 별개로 둬야 하나?
다음에 할 것

다음에는 오른쪽 input을 받아 왼쪽의 segmentation map을 출력하는 segmentation model인 Unet을 구현하게 될 것!
Unet 논문도 간단히 읽어보도록 하자.
