https://arxiv.org/abs/1505.04597
U-Net: Convolutional Networks for Biomedical Image Segmentation
2015 MICCAI
Abstract
- Data augmentation에 기반한 학습 전략 및 네트워크 제시
- 매우 적은 이미지로 end to end로 학습시킴으로써 ISBI challenge(segmentation of neuronal structure in electron microsopic stacks)에서 이전 방법보다 좋은 성능 보임
Introduction
-
(이때 시점 기준) CNN은 보통 classification task에 많이 사용되었으나, biomedical image processing 시에는 output이 localization 결과를 포함하고 있어야 할 필요성 존재
- 즉, class label이 픽셀 하나하나당 부여된 형태
- 해당 논문이 나오기 전에는 각 픽셀의 class를 예측하기 위해 sliding window 방식으로 접근한 방법이 있었음
- 각 픽셀 근처의 patch(local region)를 input으로 해 localize하는 것
- 그렇게 함으로써 실제 train data가 적더라도 생성된 patch 수가 많아 이를 보완할 수 있었음
- 위 접근 방식이 ISBI 2012에서의 EM segmentation challenge에서 SOTA였음
- 그러나, 각 패치마다 처리해야 하기 때문에 느리고 overlapping된 패치들이 있어 redundancy가 크다는 문제가 있었음
- 또, 설정마다 localization accuracy에 있어 trade off 존재
- 패치를 크게 할 경우, max pooling layer를 많이 필요로 하기 떄문에 localization accuracy 하락하고, 패치를 작게 할 경우 네트워크가 너무 작은 context만을 고려하게 됨
- 위 문제를 해결하기 위해 여러 layer로부터 feature를 고려하는 classifier도 제안되었음
- localization 성능을 높일 뿐 아니라, context를 더 많이 활용할 수 있게됨!
- 각 픽셀 근처의 patch(local region)를 input으로 해 localize하는 것
-
본 논문에서는 "fully convolutional network" 제안
- 해당 모델 구조를 통해 적은 train image로도 더 정확히 segmentation을 수행할 수 있다는 것 확인
모델 구조
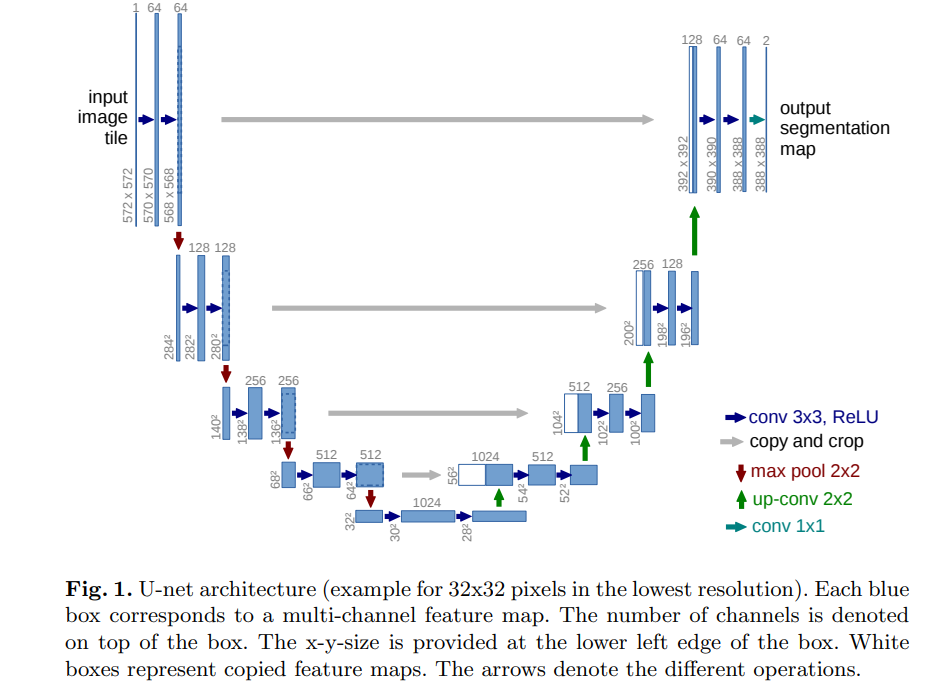
- 파란 박스는 multi channel feature map이고, 옆의 값들은 각각 x,y,z값을 나타냄
- 하얀 박스는 copy된 feature map
- 왼쪽 path를 contracting path, 오른쪽을 expansive path라고 함
- 활성화함수로는 ReLU 사용
- Downsampling step에서는 feature channel 수를 두 배씩 증가시킴
- Upsampling convolution에서는 2x2 convolution 사용
- 이때, contracting path로부터 대응되는 crop된 feature들을 concat해주면서 증가한 채널 수를 줄여줌
- 여기서도 ReLU 사용
- 매 convolution마다 border 부분의 픽셀에 있어 정보 손실이 있기 때문에 중앙부를 중점으로 해 crop해주는 것
- 마지막 1x1 convolution은 각 feature vector를 class number로 변환해주기 위함 (foreground, background)
- 총 23개의 convolutional layer로 이뤄져있음
- 그림에서 알 수 있듯, 대칭 구조로 서로 contracting하는 것을 알 수 있음.
이때, pooling layer는 upsampling layer로 대체됨- output의 해상도를 높이는 역할
- localize를 하기 위해서는 contracting path로부터의 high resolution feature들이 upsampling된 output과 섞임
- 그렇게 함으로써 연속된 convolutional layer가 해당 정보를 통해 더 정확한 output을낼 수 있도록 학습될 수 있음
- upsampling part 또한 많은 feature channel 수를 갖는 게 특징
- 더 높은 해상도를 갖는 layer에 context information을 더 잘 전달하도록 함
- FC layer가 전혀 없음
- 각 convolution으로부터의 결과만을 사용
- 즉, segmentation map은 입력 이미지의 모든 context 정보를 갖는 픽셀들로 이뤄짐
- overlap-tile 전략으로 임의의 큰 사이즈의 이미지에서도 segmentation이 잘 수행됨
- border 부분의 픽셀들을 예측하기 위해서는 input image를 mirroring함으로써 추정
- 이를 통해 손실되는 정보를 보완함
- GPU 메모리 제한으로 인해 해상도에 있어 제한 존재. 그렇기에 큰 이미지에 모델을 적용할 때 해당 전략이 중요하게 작용함
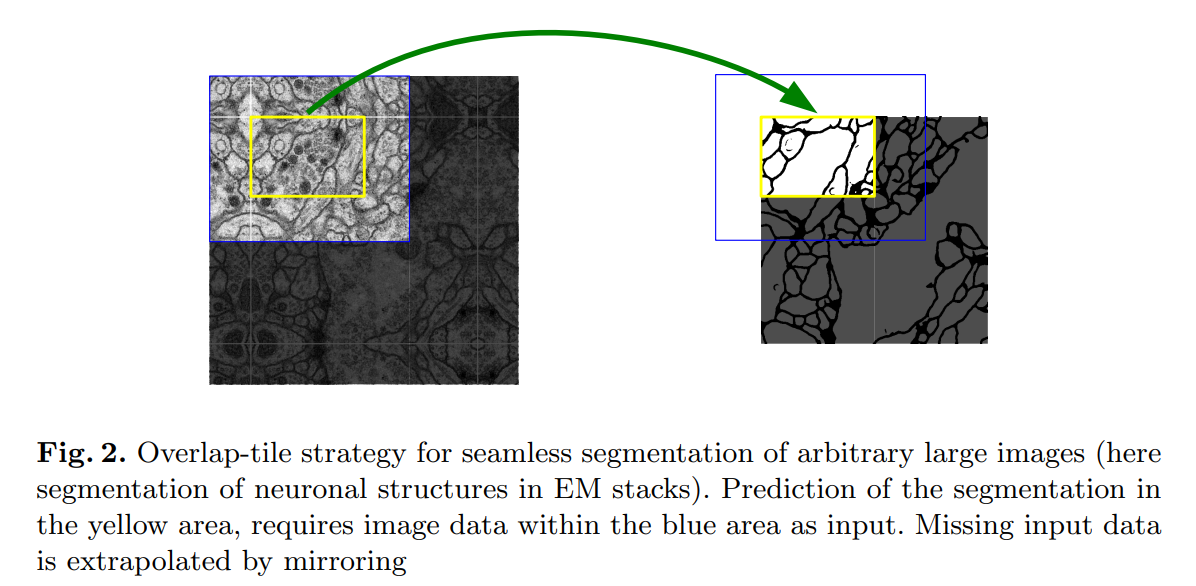
- 각 convolution으로부터의 결과만을 사용
- 입력 이미지가 적을 때를 고려해 다양한 data augmentation 적용
- biomedical segmentation에 있어 특히 중요
- 실제에서 있을법한 변형들을 적용 (tissue 이미지)
- biomedical segmentation에 있어 특히 중요
- Cell segmentation에 있어 어려움 중 하나는 맞닿아있는, 같은 class에 속하는 object들을 어떻게 나눌 것인가에 대한 것
- 이를 보완하기 위해 weighted loss 제안
- 맞닿아있는 cell 간의 background label에 더 큰 weight를 부여 -> 잘 분리할 수 있을 것!
- 이를 보완하기 위해 weighted loss 제안
- 해당 모델 구조는 다양한 biomedical segmentation 문제에 적용 가능
- ISBI 2012 challenge의 EM stack에서의 neuronal structure segmentation 결과를 보여줌
- ISBI cell tracking challenge 2015에서의 light microscopy image cell segmentation의 결과도 보여줌
2. Network Architecture
위에 설명했으므로 패스
3. Training
- GPU 메모리를 최대한 활용하기 위해, 배치 사이즈를 크게 하는 것 보다 input tile을 크게 하는 것을 선택함
- 즉, single image를 하나의 배치로서 사용
- 높은 momentum 값(0.99) 사용
- Energy function으로는 최종 feature map에 대한 pixel wise softmax함수와 결합된 cross entropy loss 사용
Energy Function
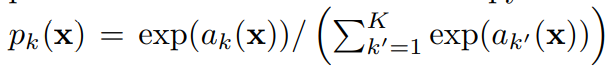

Weight map
- training 이미지 상에서 foreground label을 갖는 픽셀 수와 background label을 갖는 픽셀 수 간 차이가 큼
- 이를 보정하기 위해 weight map을 미리 계산
- (touching cell들에 대한)small separation에 대해서도 학습을 원활히 할 수 있도록 강제하는 역할
- 이를 보정하기 위해 weight map을 미리 계산
- morphological operation을 통해 separation border 게산
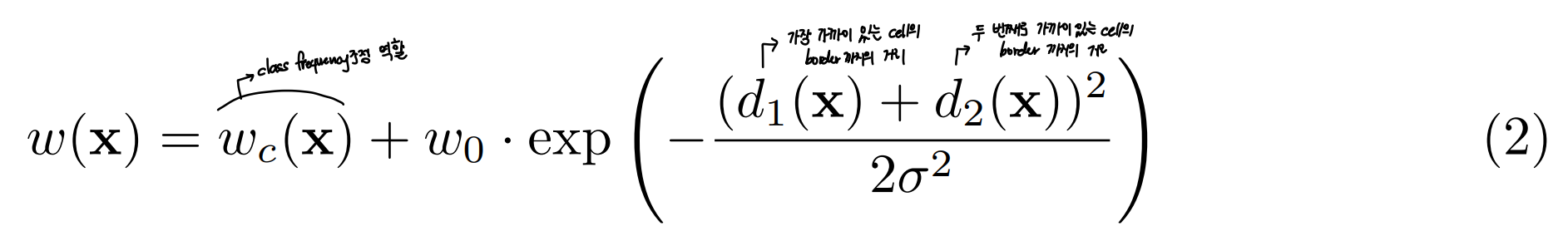
- 본 논문에서는 값으로 10을, 값으로 5를 사용
- Conovlution layer를 많이 사용하는 deep한 네트워크일수록 가중치 초기화가 매우 중요!
- 어디는 엄청 활성화된데 반해, 다른 곳에서는 전혀 기여하지 못할 수도..
- 각 feature map이 unit variance를 갖도록 하는 게 이상적이지만..
- 본 논문에서는 의 표준편차 값을 갖는 Gaussian distribution으로부터 가중치를 초기화하는 방안 채택
- 이때, N은 input의 노드 수를 의미

- a는 원본 이미지, b는 ground truth를 표시한 것.
- 이때, 다른 색은 다른 instance를 나타냄
- c는 생성된 segmentation mask. 하얀 색이 foreground
- d가 pixel-wise loss weight인데, border에 대해 weight가 강하게 주어진 것을 알 수 있음
3.1 Data Augmentation
-
모델의 강건성을 위해 중요
-
microsopical image의 경우, shift와 rotation에 대한 변형이 현실적.
-
추가적으로 elastic distortion적용
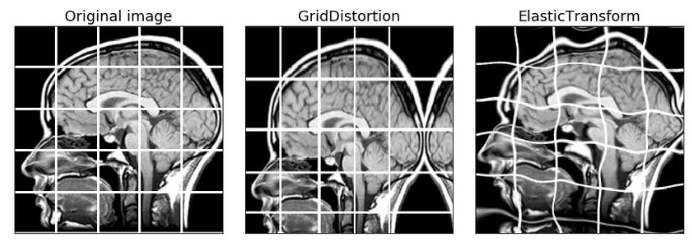
Elastic distortion이라는 용어를 처음 접해봤는데, 이런 느낌인가보다. 추가적으로 공부하자 -
coarse 3x3 grid 상에서의 random displacement vector를 사용해 smooth deformation을 생성한다고 한다.
- 이때, displacement들은 10 pixel 표준편차를 갖는 Gaussian distribution에서 샘플링된다고 한다.
- Per-pixel displacement들은 bicubic interpolation으로 계산된다고 한다.
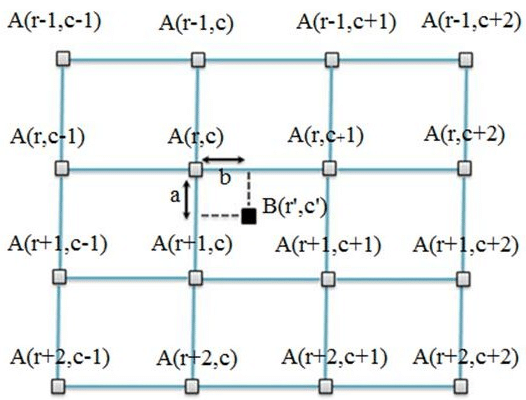
요런 느낌
-
contracting path의 마지막에 Drop out layer를 적용함으로써 data augmentation의 효과를 추가적으로 줬다고 한다.
4. Experiments
세 가지 segmentation task에 적용
EM segmentation challenge
- 30개의 training image(512 x 512)
- Ventral nerve cord(VNC)
- cell에 대한 groundtruth가 white색상으로, membrane에 대한 groundtruth가 black색상으로 되어있음
- evaluation은 10가지의 다른 레벨에서 map을 thresholding함으로써 진행
- warping error, Rand error, pixel error 계산 (이 기준은 잘 모르겠음.. 설명이 제대로 안되어있다.)
Warping error?
- 객체를 잘 분할했는가
Pixel error?
- 맞고 틀린 픽셀 수에 대한 것
Rand error?
- 두 데이터 클러스터 간 similarity 측정 위함
자세한 건 추가적으로 공부하자.
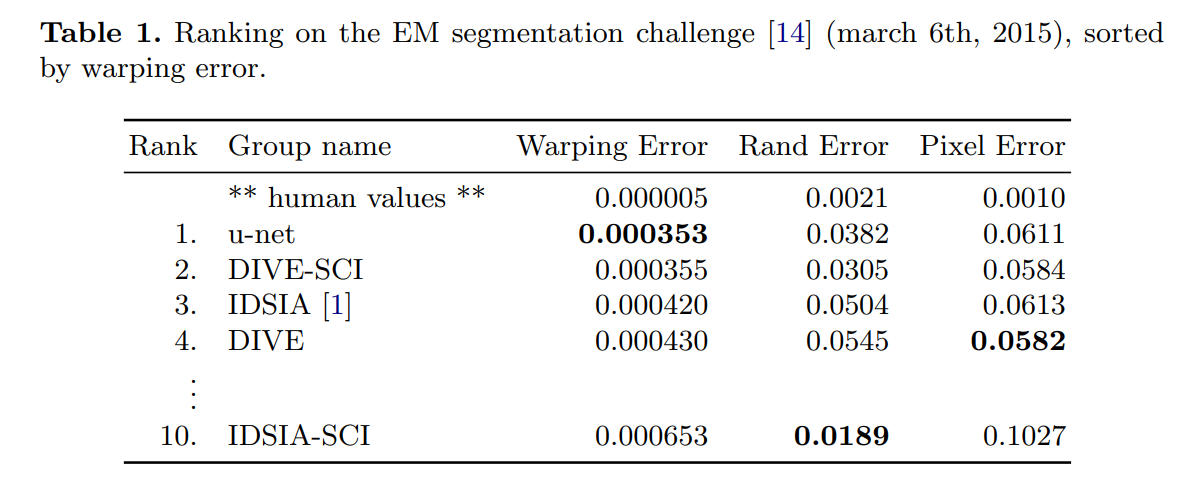
EM segmentation challenge에서 U-net의 warping error가 가장 낮다.
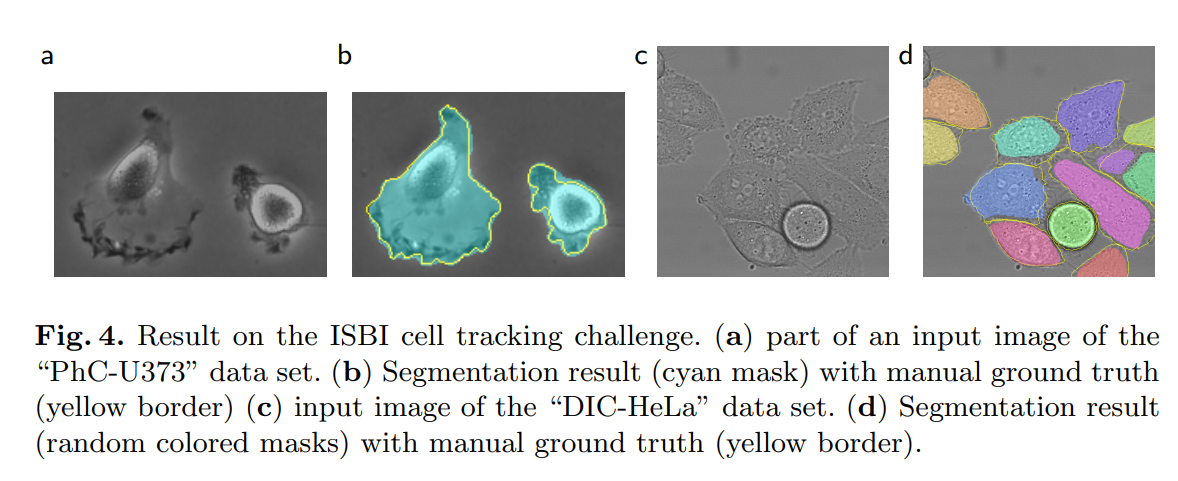
- 위 그림은 ISBI cell tracking challenge의 결과를 보여준다.
- PhC-U373 dataset, DIC-HeLa dataset 사용
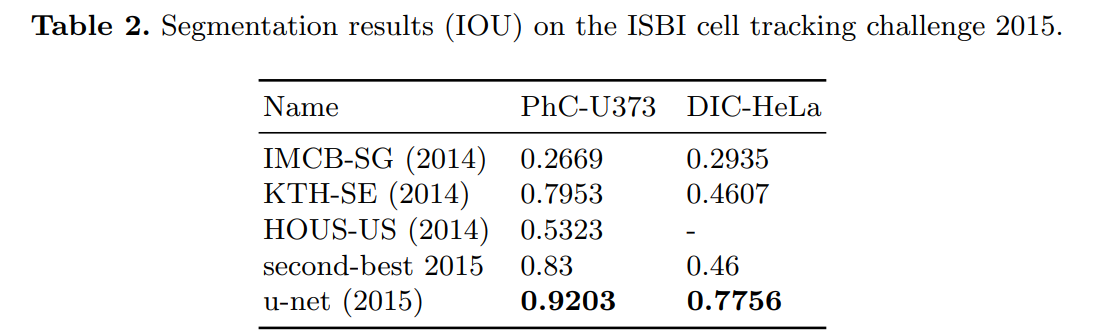
- 위 표는 ISBI cell tracking challenge에서의 IOU 결과를 보여준다.
PhC-U373 Dataset
- 35개의 부분적으로 annotating된 training image로 이뤄짐
DIC-HeLa Dataset
- 20개의 부분적으로 annotating된 training image로 이뤄짐
5. Conclusion
- 데이터셋이 워낙 작아서 NVidia Titan GPU(6GB)상에서 10시간동안 훈련했다고 한다.
여담
분명 간단하게 읽으려 했는데, 읽다보니 너무 꼼꼼히 읽느라 시간을 많이 써버린 느낌.
-
Elastic distortion 뭔지 공부하기!
https://www.kaggle.com/code/babbler/mnist-data-augmentation-with-elastic-distortion -
각 Error가 무엇인가
https://imagej.net/plugins/tws/topology-preserving-warping-error
