[GWAS] 1. GWAS, plink
GWAS
[GWAS 논문] https://www.nature.com/articles/s43586-021-00056-9
위의 논문 중 일부분 정리
GWAS
개개인의 Genetic varient 의 allel frequency 비교하여 표현형과 유전자형 사이의 관계를 알아냄.
일반적으로 개개인의 여러 SNP를 분석한다. (한사람당 몇만~ 의 SNP에 대한 정보를 가지고 있음)
전체의 서열을 분석하는 Whole genome sequencing과 구분.
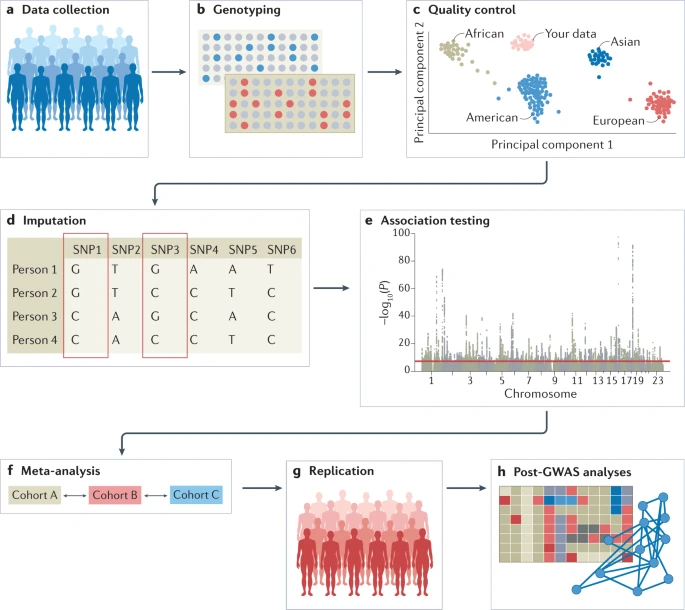
Overview of steps for conducting GWAS.
1. Data processing
input file
- 익명화된 개인 ID
- 가족관계
- 성별
- Phenotype 정보
- Covariate (공변량) : 잡음인자와 보고자 하는 독립변수가 공유하는 변량
- Genotype call : 각 개체의 유전자형 결정
Quality control
- rare, monomorphic 한 varient 제거
- Hardy - Weinberg equilibrium 안맞는 varient 제거
- missing SNP 제거
- Genotype 오류 확인, 제거
- Phenotype 확인
QC 까지 마치면 phasing 과 imputation 진행
Phasing
paternal or maternal 확인
Imputation
direct 하게 보지 못한 유전자형에 대한 통계적 추론
-
haplotype 이용 : 1000 Genome project 혹은 TopMed 이용
*연관에 의해 같이 유전되는 SNP 집합 패턴 -
GWAS consotia : 정기적으로 QC, Imputation 수행, 서버에 업로드됨
https://imputationserver.sph.umich.edu/index.html#!
https://imputation.biodatacatalyst.nhlbi.nih.gov/#!
2. Association 수행
Biometrical model기반
Phenotype : binary 또는 Continous
age, sex, ancestry 같은 공변량 확인
- Linear regression for GWAS
3. False Positive 확인
Gene - phenotype 연관성 분석
False positive : 관련 없는데 있다고 판단
공통적인 유전자 변이가 아닌 associated 변이만 찾기
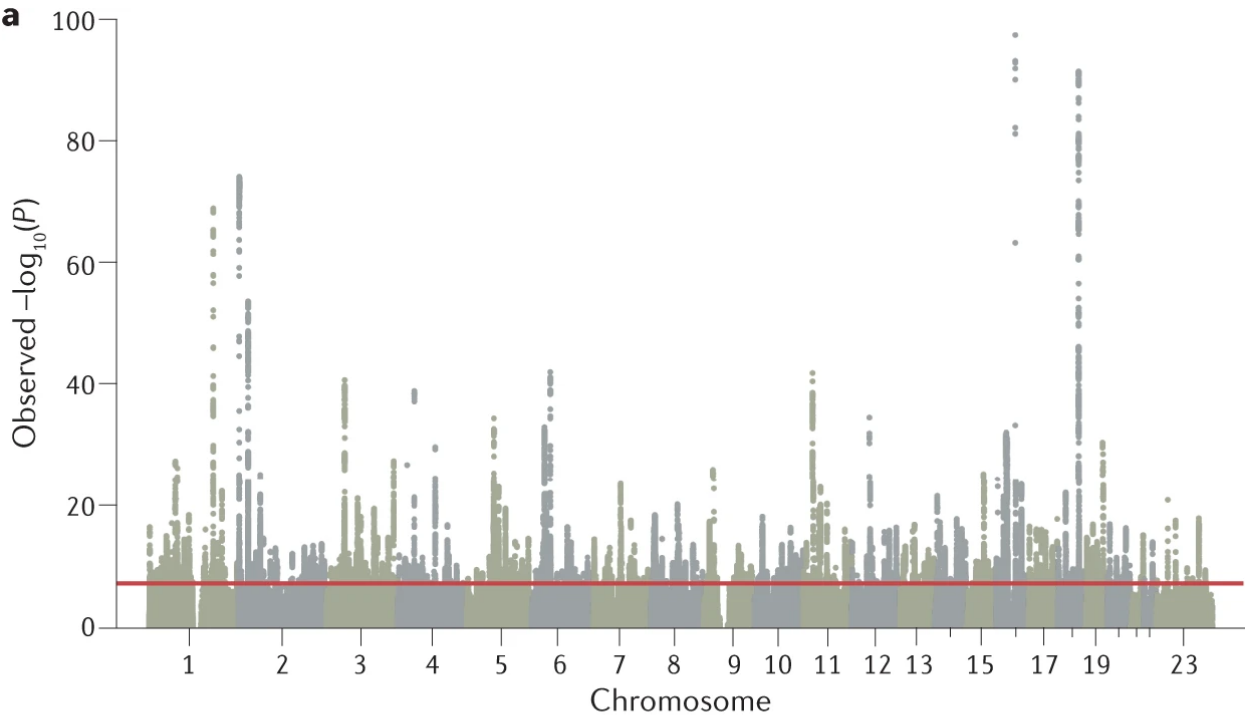
- Bonferroni testing threshold
P < 5 × 10–8 (representing a false discovery rate of 0.05/106)
population 따라 고려. 크기 클수록 엄격
highly polygenic 할 경우 associated gene 무시될 가능성 고려
4. Result
GWAS 결과 -> p values with phenotype
사용 전 추가 분석 필요
-
LD (Linkage Disequilibrium)
-
statistic fine-mapping
LD로 인해 non-causal varient 가 연관되어 있을 수 있음.- Fine mapping
GWAS로 확인한 유전자 위치 내에서 Phenotype과 관련있을 집합의 우선순위 결정하도록 설계
1. Conditional Association Analysis
2. Baysian Model
- Fine mapping
PLINK
-
QC, Association 수행하는 tool kit
.vcf 변환 등. -
ped , map 파일을 이용하며 유저가 많을 경우 binary 파일로 변환하여 사용
-> bim, fam, bed file이 생성됨 -
여러 유저의 GWAS 정보를 기반으로 연관 분석 등을 수행하는 tool일 뿐...
.bim
| Chromosome | Varient identifier (Marker ID) | Position in morgans or centimorgan | Position (bp) | Allel 1 (usually minor) | Allel 2 (usually Major) |
|---|---|---|---|---|---|
| 1 | rs2821050 | 3.7593 | 3497109 | T | C |
.fam
| Family ID | User ID | Paternal ID | Maternal ID | Sex code | Phenotype |
|---|---|---|---|---|---|
| 1 | 022_S_4320 | 0 | 0 | 1 : Male 2 : Female | 1: Control 2: Case -9/0/non : Missing |
.bed
SNP, 환자 정보 들어있는 binary 파일. 대량 정보 저장- 열 수 X
Method
.fam에 phenotype 넣어서 분석.
binary 혹은 continous 값 대입.