Process
- Create Tokenizer
- Make Dataset
- NSP(Next Sentence Prediction)
- Masking
Training
앞서 배웠던 내용이랑 조금 상반되는 내용이라 일단 적어본다.
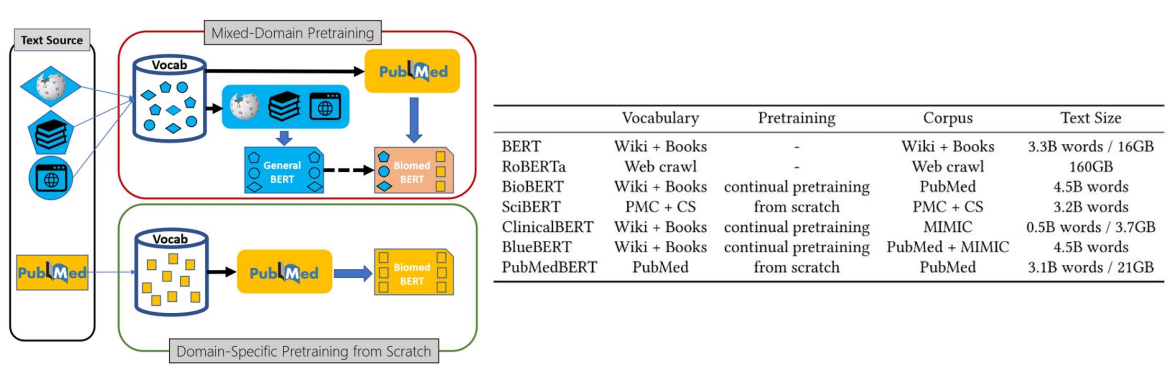
도메인 특화 task에서는 Pretrained model을 fine-tuning하는 것보다, 도메인 특화 데이터만 사용해 새롭게 학습하(scratch)는 것이 더 성능이 좋다.
ref: https://huggingface.co/microsoft/BiomedNLP-PubMedBERT-base-uncased-abstract
생리학 저널들을 아카이빙하는 곳들 중 가장 큰 PubMed의 데이터를 사용해 새롭게 BERT를 학습한 논문이다.
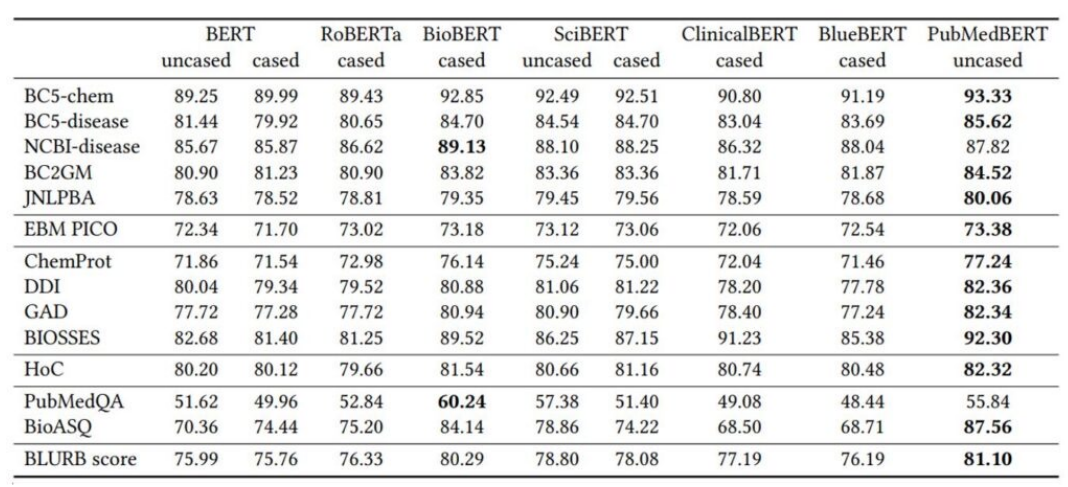
생리학 관련 task들에 대한 BERT들의 score다. 가령, BC5-chem은 화학 관련 개체명 인식 task다. 이러한 도메인 특화 task들은 fine-tuning보다 생리학 관련 데이터만 사용해 처음부터 BERT를 학습시키는 것이 성능이 좋다.
Data
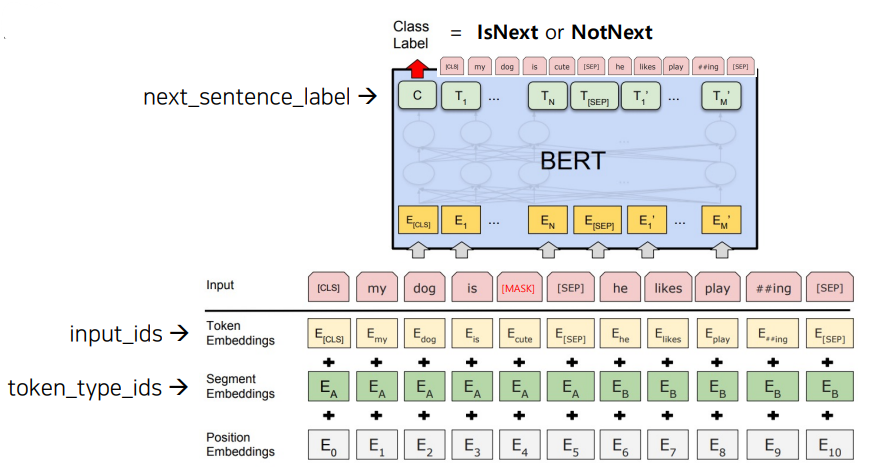
Dataset
model에 들어갈 수 있는 형태로 data를 바꿔준다.
BERT 입장에서는 다음과 같이 data를 새롭게 생성해 모델에 넣어줘야된다.
- input_ids: Token Embedding을 통해 생성된 Vocab Id.
- token_type_ids: Segment Embedding을 통해 생성된 Segment Id.
- Positional encoding 정보
target_seq_length
github에 있는 BERT 코드다. 여기서 BERT의 Embedding size를 다음과 같이 조절한다.
- max_num_tokens: BERT에 들어갈 수 있는 최대 token 개수
target_seq_length = max_num_tokens
if random.random() < self.short_seq_probability:
target_seq_length = random.randint(2, max_num_tokens)short_seq_probability에 의해서 확률적으로 target_seq_probability가 랜덤한 값을 가진다.
이렇게 조절해주는 이유는 모델의 범용성 때문이다. 만약 max_num_token대로 모든 데이터를 꽉꽉 채워서 학습하면 해당 모델은 max_num_token 이외의 token 수를 입력받으면 제대로 처리하지 못할 가능성이 높다.
따라서 확률적으로 최대 Embedding size를 조절해줘서 유연한 모델을 만들고자 한다.
Segment 조절
258번줄부터는 Segment를 조절하기 위한 코드다.
Dataset은 max embedding size를 꽉 채우는 데이터를 만들려고 노력할 것이다. 즉, '문장_1[SEP]문장_2'의 token size가 부족하다면 '문장_1+문장_2[SEP]문장_3+문장_4'과 같이도 만들겠다는 것이다. 물론 Segment는 여전히 2개다. '문장_1+문장_2'가 하나의 Segment가 되는 것이다.
이 때, 코드에서는 랜덤하게 Segment A(첫번째 segment)의 길이를 자른다. 말 그대로 랜덤한 정수 값을 얻고 해당 값까지만 Segment로 사용한다.
Truncation
'SegmentA[SEP]SegmentB'가 최대 Embedding size를 넘어설 수도 있다. truncation 작업이 필요한 조건이다.
truncation은 아래의 작업을 반복한다.
- 랜덤하게 Segment A와 B 중 한 가지를 선택한다.
- Segment의 가장 뒤에 오는 token을 제거한다.
- token 개수를 검사하고 다시 truncation이 필요하면 1번으로 회귀.
Dataloader
model에게 어떻게 data를 전달할 것인가를 결정.
BERT 입장에서는 Masking을 어떻게 할 것인가에 대한 문제가 된다.
