구강악 2D,3D 이미지(AIHub)
의료 영상 이미지이기 때문에 자세한 내용을 넣지는 못했습니다.
데이터셋 설명
구축년도 : 2020
설명 : 본 데이터셋은 구강에 대한 파노라마 영상(2D) 영상을 통한 치아 검출,CBCT(3D)영상을 통해 치아를 분류,분할 하치조 신경관을 분할
목적 : 치아 검출(Object Detection),치아 분할(Segmentation),하치조 신경관 분할(Segmentation)
추천 모델 : 치아 검출(Object Detection)용 추천 모델로는 YOLO V3 모델이 있으며 치아 분할과 하치조 신경관 분할(Segmentation)에는 3D UNet이 있다. Annotation 설명
입력 데이터 - jpg이미지로 3D CBCT의 경우 z축을 기준으로 단면 이미지들을 jpg로 저장
라벨 - json 파일 내부(AIHub 사이트 내의 사진)
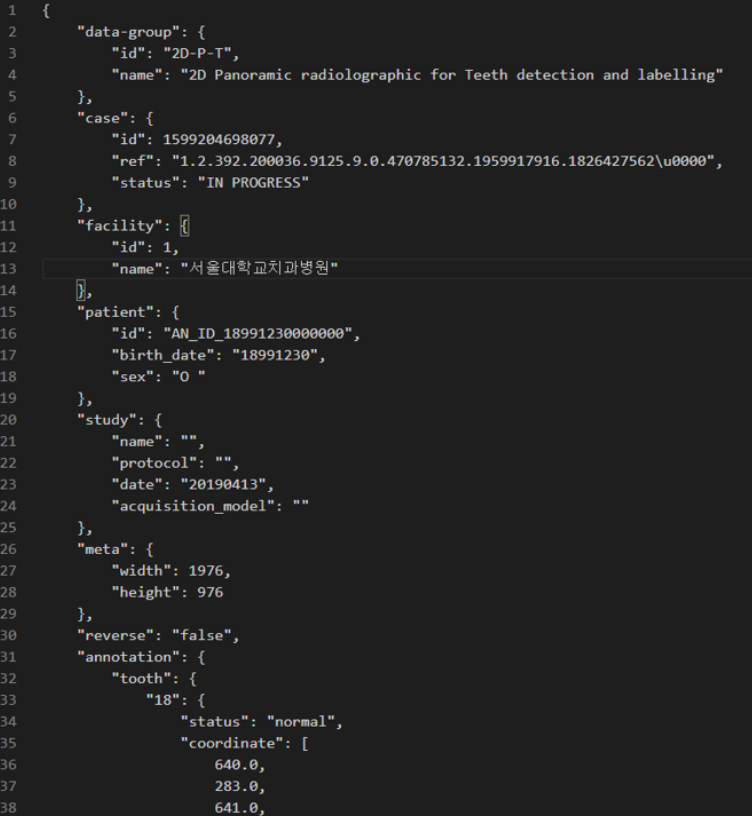
이 중 학습에 사용될 데이터는 다음과 같습니다.
data-group : 3D CBCT의 이미지임에도 불구하고 라벨은 2D 파노라마 이미지인 경우가 존재
id : 경우에 이 부분을 확인하면서 3D CBCT임을 확인해야함
meta : 해당 데이터셋의 입력의 너비,높이를 나타내며 3D CBCT의 경우 depth까지 추가 된다.
width : 각 이미지의 너비에 대한 정보
height : 각 이미지의 높이에 대한 정보
depth : 3D CBCT 이미지의 경우에만 존재, 각 3D CBCT의 높이에 대한 정보
annotation : 해당 데이터셋에 각각의 치아에 대한 좌표 정보가 나타나있다.
tooth : 치아 정보를 나타냄
num : 특정 치아에 해당하는 번호
status : 해당 치아가 있는지 없는지에 대한 정보
coordinate : list로 2D의 경우[x1,y1,x2,y2,...,xn,yn] 이고 3D의 경우[x1,y1,z1,...,xn,yn,zn] num이 18인 경우 mask[y][x][z] = 18로 처리됨Annotation 활용과정(3D CBCT)
3D CBCT 데이터를 활용하면서 Layer UNet의 2D convolution을 3D convolution 으로 사용하기에는 모델이 너무 커 GPU의 메모리가 부족하여 2D Slice로 사용하려고 하였습니다.
2D를 사용하기 위해 json 파일을 읽고 특정 slice의 annotation을 얻어내는 과정에서 개당 0.9초의 시간이 소요됨 이를 통해 전처리를 해주어 각 단면들을 따로 저장해 불러오도록 사용하였습니다.
데이터 셋 문제점
1. 3D CBCT의 경우 json 내의 ['meta']['depth']의 값이 실제 CBCT 이미지의 depth와 다른 경우가 발생 (이 경우 해당 데이터는 사용하지 않음)