
통계적 가설검정
모집단에 대해 어떤 가설을 설정하고, 그 모집단으로부터 추출된 표본을 분석함으로써,
그 가설이 틀리는지 맞는지 타당성 여부를 결정하는 통계적 기법이다.Statistical Hypothesis Test
- 검정통계량 (Test Statistics)
- 가설 검정 (Hypothesis Test)
- 귀무 가설 H₀ (Null Hypothesis)
- 대립 가설 H₁ (Alternative Hypothesis)
- 유의수준 α (Significance Level)
- 기각역 (Critical Region, Rejection Region)
- 임계치 (Critical Value)
- 검정통계량이 기각역에 속하면, 귀무가설을 기각
- p-value (유의확률)
p-value < α: 귀무가설 기각p-value > α: 귀무가설 채택
모평균에 대한 검정
모집단이 정규분포를 따르며, 모분산 σ₀² 이 알려진 경우
- Z ~ N(μ₀,σ₀²)
- σ₀²
scipy.stats.norm인스턴스 활용
| 가설의 종류 | 기각역에 의한 검정 | p-value에 의한 검정 |
|---|---|---|
| H₀: μ = μ₀ H₁: μ > μ₀ | if Z > Zα, H₀ rejected if Z > stats.norm.isf(α), H₀ rejected | p_value ≤ α, H₀ rejected |
| H₀: μ = μ₀ H₁: μ < μ₀ | if Z < -Zα, H₀ rejected if Z < stats.norm.ppf(α), H₀ rejected | p_value ≤ α, H₀ rejected |
| H₀: μ = μ₀ H₁: μ ≠ μ₀ | if |Z| > Zα/2, H₀ rejected if |Z| > stats.norm.isf(α/2), H₀ rejected | p_value ≤ α, H₀ rejected |
모집단이 정규분포를 따르며, 모분산 σ² 이 알려지지 않은 경우
- Z ~ N(μ₀,σ²)
- T ~ t(n-1)
- ddof = n - 1
scipy.stats.t인스턴스 활용
| 가설의 종류 | 기각역에 의한 검정 | p-value에 의한 검정 |
|---|---|---|
| H₀: μ = μ₀ H₁: μ > μ₀ | if T > tα,n-1, H₀ rejected if Z > stats.t.isf(α, ddof), H₀ rejected | p_value = stats.t.sf(T) p_value ≤ α, H₀ rejected |
| H₀: μ = μ₀ H₁: μ < μ₀ | if T < -tα, H₀ rejected if T < stats.t.ppf(α, ddof), H₀ rejected | p_value = stats.t.cdf(T) p_value ≤ α, H₀ rejected |
| H₀: μ = μ₀ H₁: μ ≠ μ₀ | if |T| > tα/2, H₀ rejected if |T| > stats.t.isf(α/2, ddof), H₀ rejected | p_value = 2 * min(stats.t.sf(T), stats.t.cdf(T)) p_value ≤ α, H₀ rejected |
scipy.stats.ttest_1samp 함수
scipy.stats.ttest_1samp(sample, popmean)함수 활용scipy.stats.ttest_1samp( a, popmean, axis=0, nan_policy='propagate', alternative='two-sided', *, keepdims=False, )
import numpy as np
import scipy.stats
sample = np.random.randn(100)
result = stats.ttest_1samp(sample, popmean=100)
print(result)
print(result.statistic)
print(result.pvalue)
print(result.df)
# 출력 결과
TtestResult(statistic=-901.7986066635818, pvalue=1.3444062839189925e-195, df=99)
-901.7986066635818
1.3444062839189925e-195
99두 독립 모집단의 모평균 차이에 대한 검정
- `scipy.stats.ttest_ind(sample1, sample2, equal_var=True)`
scipy.stats.ttest_ind(
a,
b,
axis=0,
equal_var=True,
nan_policy='propagate',
permutations=None,
random_state=None,
alternative='two-sided',
trim=0,
*,
keepdims=False,
) -> TtestResult두 모집단이 정규분포이고, 모분산이 알려지지 않았지만 동일한 경우
- `scipy.stats.ttest_ind(sample1, sample2)` 함수 활용
import numpy as np
import scipy.stats
sample1 = np.random.randn(100) * 1.5
sample2 = np.random.randn(100) * 1.5
stats.ttest_ind(sample1, sample2)
print(result)
print(result.statistic)
print(result.pvalue)
print(result.df)
# 출력 결과
TtestResult(statistic=0.7944197743329069, pvalue=0.42790223519011494, df=198.0)
0.7944197743329069
0.42790223519011494
198.0두 모집단이 정규분포이고, 모분산이 알려지지 않은 경우
- `scipy.stats.ttest_ind(sample1, sample2, equal_var=False)` 함수 활용
import numpy as np
import scipy.stats
sample1 = np.random.randn(100) * 1.5
sample2 = np.random.randn(100) * 1.7
result = stats.ttest_ind(sample1, sample2, equal_var=False)
print(result)
print(result.statistic)
print(result.pvalue)
print(result.df)
# 출력결과
TtestResult(statistic=-1.8363686911167, pvalue=0.06780564376279984, df=197.59327523235055)
-1.8363686911167
0.06780564376279984
197.59327523235055대응표본(Paired Sample)에 대한 모평균 차이에 대한 검정
- `scipy.stats.ttest_rel(sample1, sample2)` 함수
scipy.stats.ttest_rel(
a,
b,
axis=0,
nan_policy='propagate',
alternative='two-sided',
*,
keepdims=False,
) -> TtestResultimport numpy as np
import scipy.stats
sample1 = np.random.randn(100) * 1.5
sample2 = np.random.randn(100) * 1.7
result = stats.ttest_rel(sample1, sample2)
print(result)
print(result.statistic)
print(result.pvalue)
print(result.df)
# 출력 결과
TtestResult(statistic=-0.3963575170035945, pvalue=0.6926943211639452, df=99)
-0.3963575170035945
0.6926943211639452
99두 독립 모집단의 중앙값(Median) 차이에 대한 검정
- Mann-Whitney U 검정 활용
scipy.stats.mannwhitneyu(sample1, sample2)함수 활용
import numpy as np
import scipy.stats as stats
sample1 = [23, 25, 28, 30, 32]
sample2 = [19, 21, 24, 26, 29]
alpha = 0.05
# Mann-Whitney U 검정
statistic, p_value = stats.mannwhitneyu(sample1, sample2)
print(f"statistic = {statistic}")
print(f"p_value = {p_value}")
if p_value < alpha:
print("null hypothesis rejected.")
else:
print("null hypothesis accepted.")모분산에 대한 검정
- Z ~ N(μ₀,σ₀²)
- (n-1)S²/σ² ~ χ²(n-1)
- ddof = n - 1
scipy.stats.chi2인스턴스 활용
| 가설의 종류 | 기각역에 의한 검정 | p-value에 의한 검정 |
|---|---|---|
| H₀: σ² = σ₀² H₁: σ² > σ₀² | if χ² > χ²α,n-1, H₀ rejected if χ² > stats.chi2.isf(α, ddof), H₀ rejected | p_value = stats.chi2.sf(χ²) p_value ≤ α, H₀ rejected |
| H₀: μ = μ₀ H₁: μ < μ₀ | if χ² < χ²1-α,n-1, H₀ rejected if χ² < stats.chi2.ppf(α), H₀ rejected | p_value = stats.chi2.cdf(χ²) p_value ≤ α, H₀ rejected |
| H₀: μ = μ₀ H₁: μ ≠ μ₀ | if χ² > χ²α,n-1 or χ² < χ²1-α,n-1, H₀ rejected if χ² > stats.chi2.isf(α, ddof) or χ² < stats.chi2.ppf(α), H₀ rejected | p_value = 2 * min(stats.chi2.sf(χ²), stats.chi2.cdf(χ²)) p_value ≤ α, H₀ rejected |
분산분석 (ANOVA)
- 연속형 자료에 대해 2개 이상의 그룹 간의 평균차이를 검정한다.
- 집단간의 평균차이가 있는지 없는지 검정한다.
귀무가설- 집단 간의 평균차이가 없다.
대립가설- 집단 간의 평균차이가 있다.
1. scipy.stats.f_oneway 함수 활용
- `scipy.stats.f_oneway(*samples)`
scipy.stats.f_oneway(
*samples,
axis=0,
nan_policy='propagate',
keepdims=False
) -> F_onewayResultimport numpy as np
from scipy import stats
# 예제 데이터 생성
group1 = np.random.normal(20, 5, 100)
group2 = np.random.normal(22, 5, 100)
group3 = np.random.normal(19, 5, 100)
df = pd.DataFrame({"group1": group1, "group2": group2, "group3": group3})
statistic, p_value = stats.f_oneway(df["group1"], df["group2"], df["group3"])
print(f"statistic = {statistic}")
print(f"p_value = {p_value}")
# 출력 결과
statistic = 10.674059823981155
p_value = 3.3368970343403504e-052. statsmodels.api.stats.anova_lm 함수 활용
statsmodels.api.stats.anova_lm(model)statsmodels.formula.api.ols
import numpy as np
from scipy import stats
import statsmodels.api as sm
import statsmodels.formula.api as smf
# 예제 데이터 생성
# group1 = np.random.normal(20, 5, 100)
# group2 = np.random.normal(22, 5, 100)
# group3 = np.random.normal(19, 5, 100)
df = pd.DataFrame({"value": np.concatenate([group1, group2, group3]), "group": np.repeat(["group1", "group2", "group3"], [len(group1), len(group2), len(group3)])})
# ANOVA 수행
ols = smf.ols('value ~ C(group)', df).fit()
anova_table = sm.stats.anova_lm(ols)
print(anova_table) # DataFrame 리턴
# 출력 결과
df sum_sq mean_sq F PR(>F)
C(group) 2.0 497.143511 248.571755 10.67406 0.000033
Residual 297.0 6916.376013 23.287461 NaN NaN범주형 변수 적합도, 독립성 검정
카이제곱 적합도 검정
하나의 범주형 변수에 대해 관측 값들이 기대하는 비율 분포를 따르는지 검정
scipy.stats.chisquare(
f_obs,
f_exp=None,
ddof=0,
axis=0
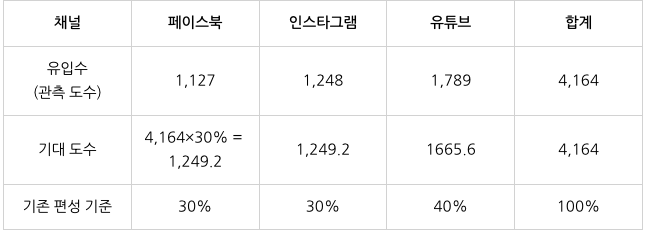
) -> Power_divergenceResultobserved = np.array([1127, 1248, 1789])
expected = np.array([0.3, 0.3, 0.4]) * observed.sum()
dof=len(observed) - 1
statistic, p_value = stats.chisquare(observed, expected)
print(f"statistic = {statistic}")
print(f"p_value = {p_value}")
# 출력 결과
statistic = 21.09746237592058
p_value = 2.6226736564792922e-05카이제곱 독립성 검정
서로 다른 두 범주형 변수 간에 연관성이 있는지를 검정
stats.chi2_contingency(
observed,
correction=True,
lambda_=None
) -> Chi2ContingencyResultimport pandas as pd
import scipy.stats as stats
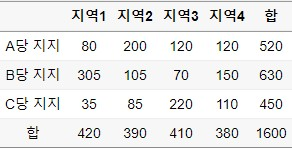
df = pd.DataFrame({"지역1": [80, 305, 35], "지역2": [200, 105, 85], "지역3": [120, 70, 220], "지역4": [120, 150, 110]}, index=["A당 지지", "B당 지지", "C당 지지"])
result = stats.chi2_contingency(df)
# statistic, p_value, dof, expected_freq = stats.chi2_contingency(df)
print(f"statistic = {result.statistic}")
print(f"statistic = {result.pvalue}")
print(f"statistic = {result.dof}")
print(f"statistic = {result.expected_freq}")
# 출력 결과
statistic = 411.3522846166838
p_value = 1.0128548246241254e-85
dof = 6
expected_freq = [[136.5 126.75 133.25 123.5 ]
[165.375 153.5625 161.4375 149.625 ]
[118.125 109.6875 115.3125 106.875 ]]pd.crosstab(index, columns)
pd.crosstab(
index,
columns,
values=None,
rownames=None,
colnames=None,
aggfunc=None,
margins: 'bool' = False,
margins_name: 'Hashable' = 'All',
dropna: 'bool' = True,
normalize: "bool | Literal[0, 1, 'all', 'index', 'columns']" = False,
) -> 'DataFrame'- 코드 예시
import pandas as pd
# 예제 데이터
data = {
'A': ['foo', 'foo', 'bar', 'bar', 'foo', 'bar'],
'B': ['one', 'one', 'two', 'two', 'one', 'one'],
'C': [1, 2, 3, 4, 5, 6]
}
df = pd.DataFrame(data)
# 기본 교차표
crosstab_basic = pd.crosstab(df['A'], df['B'])
print(crosstab_basic)
# 출력 결과
B one two
A
bar 1 2
foo 3 0
# aggfunc 사용 (합계)
crosstab_sum = pd.crosstab(df['A'], df['B'], values=df['C'], aggfunc='sum')
print(crosstab_sum)
# 출력 결과
B one two
A
bar 6.0 7.0
foo 8.0 NaN
# margins 사용 (합계 행과 열 추가)
crosstab_margins = pd.crosstab(df['A'], df['B'], margins=True)
print(crosstab_margins)
# 출력 결과
B one two All
A
bar 1 2 3
foo 3 0 3
All 4 2 6
# dropna 사용 (NaN 제거)
df_with_nan = df.copy()
df_with_nan.loc[0, 'B'] = None
crosstab_dropna = pd.crosstab(df_with_nan['A'], df_with_nan['B'], dropna=True)
print(crosstab_dropna)
# 출력 결과
B one two
A
bar 1 2
foo 2 0
# normalize 사용 (정규화)
crosstab_normalize = pd.crosstab(df['A'], df['B'], normalize='index')
print(crosstab_normalize)
# 출력 결과
B one two
A
bar 0.333333 0.666667
foo 1.000000 0.000000상관분석 (Correlation Analysis)
- 두 변수 사이의 연관성에 관한 분석
피어슨 상관계수
Pearson Correlation Coefficient
stats.pearsonr(x, y)- 변수들이 연속형이고 정규 분포를 따를 때 주로 사용됩니다.
스피어만 상관계수
Spearman's Rank Correlation Coefficient
stats.spearmanr(x, y)- 변수들이 서열형(ordinal) 자료이거나, 연속형이지만 정규 분포를 따르지 않을 때 사용됩니다.
켄달 타우 상관계수
Kendall's Tau Correlation Coefficient
stats.kendalltau(x, y)- 순위 데이터를 비교할 때 사용됩니다.
코드 예시
import numpy as np
import pandas as pd
from scipy import stats
# 예제 데이터 생성
np.random.seed(0)
x = np.random.rand(100)
y = 2 * x + np.random.normal(0, 0.1, 100)
# 데이터프레임으로 변환
df = pd.DataFrame({'x': x, 'y': y})
# 피어슨 상관계수
corr, p_value = stats.pearsonr(df['x'], df['y'])
print(f"피어슨 상관계수: {pearson_corr}, p-값: {p_value}")
# 스피어만 상관계수
corr, p_value = stats.spearmanr(df['x'], df['y'])
print(f"스피어만 상관계수: {corr}, p-값: {p_value}")
# 켄달 타우 상관계수
corr, p_value = stats.kendalltau(df['x'], df['y'])
print(f"켄달 타우 상관계수: {corr}, p-값: {p_value}")상관계수 행렬
# 피어슨 상관계수 행렬
pearson_corr_matrix = df.corr(method='pearson')
print("피어슨 상관계수 행렬:\n", pearson_corr_matrix)
# 스피어만 상관계수 행렬
spearman_corr_matrix = df.corr(method='spearman')
print("스피어만 상관계수 행렬:\n", spearman_corr_matrix)
# 켄달 타우 상관계수 행렬
kendall_corr_matrix = df.corr(method='kendall')
print("켄달 타우 상관계수 행렬:\n", kendall_corr_matrix)정규성 검정 (Nomality Test)
- 데이터가 정규분포를 따르는지 검정
Shapiro-Wilk Normality Test
샤피로-월크 정규성 검정
scipy.stats.shapiro(data)
Kolmogorov-Smirnov Test
콜모고로프-스미르노프 검정
scipy.stats.kstest(data, "norm")- ⚠️ data 파라미터에 StandardScaling 된 데이터를 넣어줘야 합니다.
D’Agostino-Pearson Test
다고스티노-피어슨 검정
scipy.stats.normaltest(data)
```python
import scipy.stats as stats
from sklearn.preprocessing import StandardScaler
data1 = [117, 108, 105, 89, 101, 93, 96, 108, 108, 94, 93, 112, 92, 91, 100, 96, 120, 86, 96, 95]
stdScaler = StandardScaler()
std_data1 = stdScaler.fit_transform(pd.DataFrame(data1)).flatten()
statistic, p_value = stats.kstest(std_data1, "norm")
```import scipy.stats as stats
import numpy as np
# data = stats.norm.rvs(size=100)
data = np.random.randn(100)
print(f"shpiro-wilk normality test = {stats.shapiro(data)}")
print(f"kolmogorov-Smirnov normality test = {stats.kstest(data, 'norm')}")
print(f"D'Agostino-Pearson normality test = {stats.normaltest(data)}")
# 출력 결과
shpiro-wilk normality test = ShapiroResult(statistic=0.9848038416985322, pvalue=0.3073893767936)
kolmogorov-Smirnov normality test = KstestResult(statistic=0.10383386500870145, pvalue=0.215652785442629, statistic_location=0.0346833095339652, statistic_sign=-1)
D'Agostino-Pearson normality test = NormaltestResult(statistic=1.364175080683408, pvalue=0.5055605120625232)