Preprocessing
ICV와 다른 요소와의 관계
import seaborn as sns
plt.figure(figsize = (4, 4), dpi = 100)
sns.scatterplot(data = df, x = "ICV", y = "Age");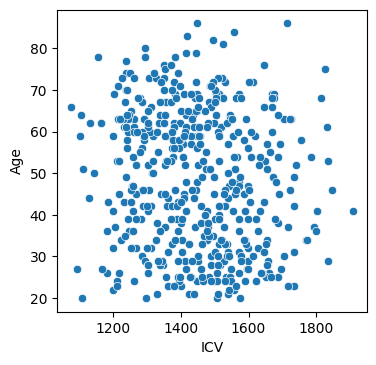
- 나이와 ICV간에 상관관계가 없음을 관찰할 수 있다.
plt.figure(figsize = (4, 4), dpi = 100)
sns.scatterplot(data = df, x = "ICV", y = "lbankssts")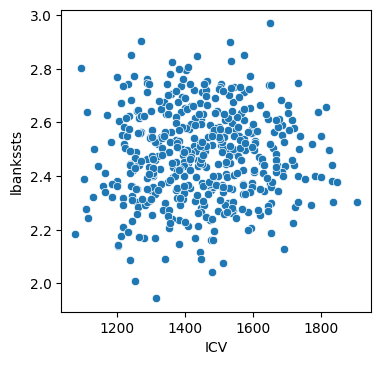
- ICV와 특정 부위의 피질 두께 간에 상관관계가 없음을 관찰할 수 있다.
Feature selection
- 위의 분포를 살펴보고 다음과 같은 case 실험을 진행하였다.
- ICV 제외
- 전체 데이터를 ICV로 나누어 동일한 부피로 가정한 후 학습을 진행
- ICV로 대뇌 피질 두께 데이터만 나눈 후 ICV도 가중치를 곱하여 학습을 진행
- 뇌를 완전한 구로 가정하여 역산 후 뇌의 반지름을 구한 후, 대뇌 피질 두께를 반지름으로 나눈 데이터로 학습을 진행
- 실험 결과 ICV를 제외하였을 때 정확도가 가장 높게 나왔기 때문에 ICV를 제외하였다.
- 또한 데이터를 성별에 따라 분류하였으며 학습에 필요없는 ID, ICV를 제외하고 True label이 Age값도 학습데이터에서 제외하였다.
man = df[df['Sex'].values == 1]
y_man = man.Age
X_man = man.drop(['ID', 'Age','Sex','ICV'], axis = 1)
woman = df[df['Sex'].values == 2]
y_woman = woman.Age
X_woman = woman.drop(['ID', 'Age','Sex','ICV'], axis = 1)정규화 및 데이터 분할
from sklearn.preprocessing import StandardScaler
std = StandardScaler()
x_man = std.fit_transform(X_man)
x_woman = std.fit_transform(X_woman)
from sklearn.model_selection import train_test_split, cross_val_score
x_man_train, x_man_valid, y_man_train, y_man_valid = \
train_test_split(x_man, y_man, test_size=0.1, shuffle=True, random_state=123)
x_woman_train, x_woman_valid, y_woman_train, y_woman_valid = \
train_test_split(x_woman, y_woman, test_size=0.1, shuffle=True, random_state=123)사용 방법
-
Age, ID, Sex를 제외한 모든 feature를 사용하는 방법
1.1 데이터를 변형하지 않고 사용하는 방법
1.2 ICV데이터 변형- The Effect of Age, Gender and Head Size on the Cortical Thickness of Brain 논문에 따르면 뇌의 표면적은 유전적 성향과 연관된 뇌의 접힙 양상 등에 따라 다양한 편차가 있어 피질 두께와 부피 사이의 관계가 항상 일정하지 않아 피질 두께 연구 결과들은 부피 연구들과 독립적인 양상들을 보인다.
- [뇌에게 나이를 묻다]의 내용에 따르면 나이가 들수록 뇌의 부피는 점점 줄어들기 시작하여 65세가 되면 20세와 비교 시 10%가량의 부피 감소가 관측된다고 한다.
- 뇌의 총 부피는 나이와 크게 상관없다는 내용을 토대로 ICV데이터의 변형 필요성을 느끼게 되었다.
-
ICV 데이터 제거
-
전체 데이터를 ICV로 나누는 방법
- 모든 사람의 뇌 부피를 1로 두고 데이터를 사용할 수 있다.
-
대뇌 피질 두께만을 ICV로 나누는 방법
- 대뇌 피질 두께를 같은 뇌부피로 가정했을 때의 데이터로 변형하며 뇌의 총 부피도 학습 결과에 영향을 주도록 변형하였다.
-
반지름을 구하여 데이터를 나누는 방법
- 뇌를 완전한 구로 가정하여 부피 공식을 역산하여 뇌의 반지름을 구한 후 대뇌 피질 두께 데이터를 반지름으로 나누어 ICV와 대뇌피질두께의 관계를 제거하고 학습을 진행하는 방법
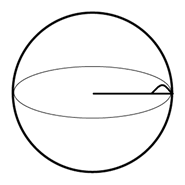
- 뇌를 완전한 구로 가정하여 부피 공식을 역산하여 뇌의 반지름을 구한 후 대뇌 피질 두께 데이터를 반지름으로 나누어 ICV와 대뇌피질두께의 관계를 제거하고 학습을 진행하는 방법
사용 회귀 모델
Lasso regression - man
from sklearn import linear_model
from sklearn.model_selection import GridSearchCV, RepeatedKFold
reg = linear_model.Lasso(max_iter = 10000, random_state = 1)
param_grid = {'alpha' : np.arange(0.01,1,0.01)}
grid = GridSearchCV(reg, param_grid = param_grid,
cv=RepeatedKFold(n_splits=5, n_repeats=5, random_state = 123),
return_train_score=True)
grid.fit(x_man_train,y_man_train)
print(f"best parameters: {grid.best_params_}")
print(f"valid-set score: {grid.score(x_man_valid, y_man_valid):.3f}")best parameters: {'alpha': 0.33}
valid-set score: 0.775
results = pd.DataFrame(grid.cv_results_)
results.plot('param_alpha', 'mean_train_score')
results.plot('param_alpha', 'mean_test_score', ax=plt.gca())
plt.fill_between(results.param_alpha.astype(np.float64),
results['mean_train_score'] + results['std_train_score'],
results['mean_train_score'] - results['std_train_score'], alpha=0.2)
plt.fill_between(results.param_alpha.astype(np.float64),
results['mean_test_score'] + results['std_test_score'],
results['mean_test_score'] - results['std_test_score'], alpha=0.2)
plt.legend()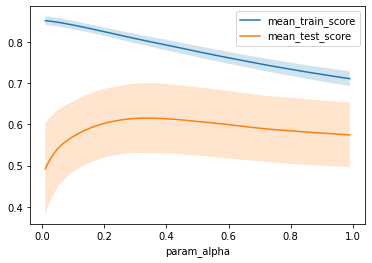
from sklearn.model_selection import cross_val_predict
reg = linear_model.Lasso(max_iter = 10000, random_state = 1, alpha = 0.33)
y_man_pred = cross_val_predict(reg, x_man_train, y_man_train, cv=5)
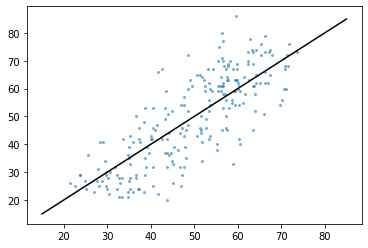
plt.plot([15, 85], [15, 85], color='k')
plt.scatter(y_man_pred, y_man_train, alpha=.5, s=4)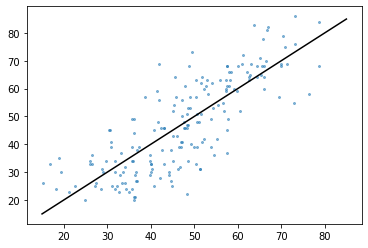
Lasso - Woman
reg = linear_model.Lasso(max_iter = 10000, random_state = 1)
param_grid = {'alpha' : np.arange(0.01,1,0.01)}
grid = GridSearchCV(reg, param_grid = param_grid,
cv=RepeatedKFold(n_splits=5, n_repeats=5, random_state = 123),
return_train_score=True)
grid.fit(x_woman_train,y_woman_train)
print(f"best parameters: {grid.best_params_}")
print(f"valid-set score: {grid.score(x_woman_valid, y_woman_valid):.3f}")best parameters: {'alpha': 0.26}
valid-set score: 0.573
results = pd.DataFrame(grid.cv_results_)
results.plot('param_alpha', 'mean_train_score')
results.plot('param_alpha', 'mean_test_score', ax=plt.gca())
plt.fill_between(results.param_alpha.astype(np.float64),
results['mean_train_score'] + results['std_train_score'],
results['mean_train_score'] - results['std_train_score'], alpha=0.2)
plt.fill_between(results.param_alpha.astype(np.float64),
results['mean_test_score'] + results['std_test_score'],
results['mean_test_score'] - results['std_test_score'], alpha=0.2)
plt.legend()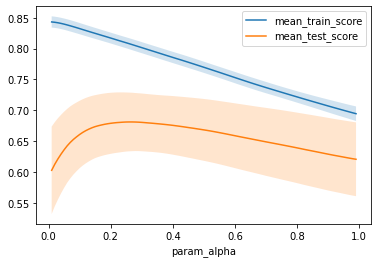
reg = linear_model.Lasso(max_iter = 10000, random_state = 1, alpha = 0.26)
y_woman_pred = cross_val_predict(reg, x_woman_train, y_woman_train, cv=5)
plt.plot([15, 85], [15, 85], color='k')
plt.scatter(y_woman_pred, y_woman_train, alpha=.5, s=4)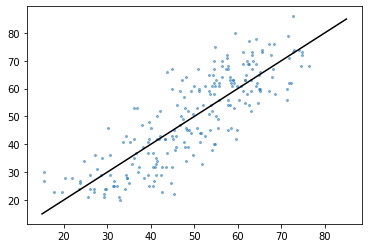
Ridge regression - man
reg = linear_model.Ridge(max_iter = 10000)
param_grid = {'alpha' : np.arange(50,60,0.1)}
grid2 = GridSearchCV(reg, param_grid = param_grid,
cv=RepeatedKFold(n_splits=5, n_repeats=5, random_state = 123),
return_train_score=True)
grid2.fit(x_man_train,y_man_train)
print(f"best parameters: {grid2.best_params_}")
print(f"valid-set score: {grid2.score(x_man_valid, y_man_valid):.3f}")best parameters: {'alpha': 56.30000000000009}
valid-set score: 0.772
results = pd.DataFrame(grid2.cv_results_)
results.plot('param_alpha', 'mean_train_score')
results.plot('param_alpha', 'mean_test_score', ax=plt.gca())
plt.fill_between(results.param_alpha.astype(np.float64),
results['mean_train_score'] + results['std_train_score'],
results['mean_train_score'] - results['std_train_score'], alpha=0.2)
plt.fill_between(results.param_alpha.astype(np.float64),
results['mean_test_score'] + results['std_test_score'],
results['mean_test_score'] - results['std_test_score'], alpha=0.2)
plt.legend()
reg = linear_model.Ridge(alpha = 56.3)
y_man_pred = cross_val_predict(reg, x_man_train, y_man_train, cv=5)
plt.plot([15, 85], [15, 85], color='k')
plt.scatter(y_man_pred, y_man_train, alpha=.5, s=4)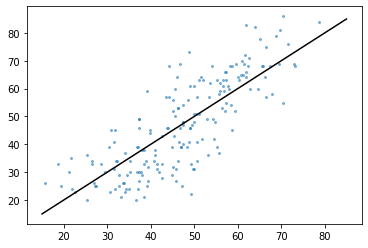
Ridge regression - woman
reg = linear_model.Ridge()
param_grid = {'alpha' : np.arange(40,50,0.01)}
grid = GridSearchCV(reg, param_grid = param_grid,
cv=RepeatedKFold(n_splits=5, n_repeats=5, random_state = 123),
return_train_score=True)
grid.fit(x_woman_train,y_woman_train)
print(f"best parameters: {grid.best_params_}")
print(f"valid-set score: {grid.score(x_woman_valid, y_woman_valid):.3f}")best parameters: {'alpha': 46.36999999999873}
valid-set score: 0.552
results = pd.DataFrame(grid.cv_results_)
results.plot('param_alpha', 'mean_train_score')
results.plot('param_alpha', 'mean_test_score', ax=plt.gca())
plt.fill_between(results.param_alpha.astype(np.float64),
results['mean_train_score'] + results['std_train_score'],
results['mean_train_score'] - results['std_train_score'], alpha=0.2)
plt.fill_between(results.param_alpha.astype(np.float64),
results['mean_test_score'] + results['std_test_score'],
results['mean_test_score'] - results['std_test_score'], alpha=0.2)
plt.legend()
reg = linear_model.Ridge(alpha = 46.3)
y_woman_pred = cross_val_predict(reg, x_woman_train, y_woman_train, cv=5)
plt.plot([15, 85], [15, 85], color='k')
plt.scatter(y_woman_pred, y_woman_train, alpha=.5, s=4)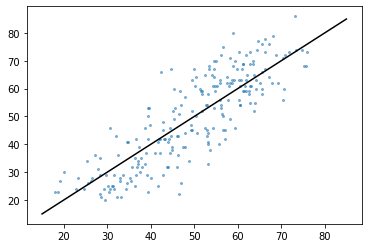
svm.SVR() - man
from sklearn import svm
reg = svm.SVR()
param_grid = { 'C': np.arange(10,20,0.1)}
grid3 = GridSearchCV(reg, param_grid = param_grid,
cv=RepeatedKFold(n_splits=5, n_repeats=5, random_state = 123),
return_train_score=True)
grid3.fit(x_man_train,y_man_train)
print(f"best parameters: {grid3.best_params_}")
print(f"valid-set score: {grid3.score(x_man_valid, y_man_valid):.3f}")best parameters: {'C': 15.999999999999979}
valid-set score: 0.587
results = pd.DataFrame(grid3.cv_results_)
results.plot('param_C', 'mean_train_score')
results.plot('param_C', 'mean_test_score', ax=plt.gca())
plt.fill_between(results.param_C.astype(np.float64),
results['mean_train_score'] + results['std_train_score'],
results['mean_train_score'] - results['std_train_score'], alpha=0.2)
plt.fill_between(results.param_C.astype(np.float64),
results['mean_test_score'] + results['std_test_score'],
results['mean_test_score'] - results['std_test_score'], alpha=0.2)
plt.legend()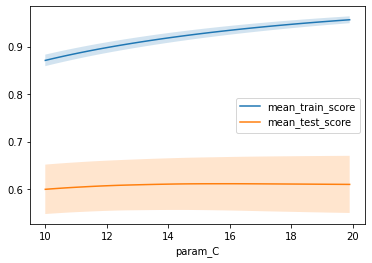
reg = svm.SVR(C = 16.0)
y_man_pred = cross_val_predict(reg, x_man_train, y_man_train, cv=5)
plt.plot([15, 85], [15, 85], color='k')
plt.scatter(y_man_pred, y_man_train, alpha=.5, s=4)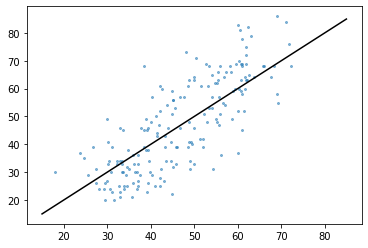
svm.SVR() - woman
reg = svm.SVR()
param_grid3 = { 'C': np.arange(20,25,0.1)}
grid3 = GridSearchCV(reg, param_grid = param_grid3,
cv=RepeatedKFold(n_splits=5, n_repeats=5, random_state = 123),
return_train_score=True)
grid3.fit(x_woman_train,y_woman_train)
print(f"best parameters: {grid3.best_params_}")
print(f"valid-set score: {grid3.score(x_woman_valid, y_woman_valid):.3f}")best parameters: {'C': 21.700000000000024}
valid-set score: 0.620
results = pd.DataFrame(grid3.cv_results_)
results.plot('param_C', 'mean_train_score')
results.plot('param_C', 'mean_test_score', ax=plt.gca())
plt.fill_between(results.param_C.astype(np.float64),
results['mean_train_score'] + results['std_train_score'],
results['mean_train_score'] - results['std_train_score'], alpha=0.2)
plt.fill_between(results.param_C.astype(np.float64),
results['mean_test_score'] + results['std_test_score'],
results['mean_test_score'] - results['std_test_score'], alpha=0.2)
plt.legend()
reg = svm.SVR(C = 21.7)
y_woman_pred = cross_val_predict(reg, x_woman_train, y_woman_train, cv=5)
plt.plot([15, 85], [15, 85], color='k')
plt.scatter(y_woman_pred, y_woman_train, alpha=.5, s=4)