DeepArrNet: An Efficient Deep CNN Architecture for Automatic Arrhythmia Detection and Classification From Denoised ECG Beats 논문 리뷰
Papers

Author : TANVIR MAHMUD, SHAIKH ANOWARUL FATTAH, MOHAMMAD SAQUIB
Keyword : Arrhythmia, Biosignal Analysis, CNN, Deep Learning, Disease detection, ECG
Abstract
저자들은 Denoised된 ECG신호로부터 자동적으로 부정맥을 탐지 및 분류하기 위한 Depthwise Temporal Convolution 기반 CNN 모델을 제안하였다. (명칭 : DeepArrNet)
wavelet Transform으로 noise를 감소시킨 ECG의 다양한 패턴을 고려하여 Class Imbalance를 해결하기위한 Data Augmentation을 진행했다고 한다.
PTP (Pointwise-Temporal-Pointwise)라고 불리는 Unit Block은 다양한 사이즈의 커널을 가진 Depthwise Temporal Convolution에 Pointwise Convolution을 앞뒤로 붙여진 구조로 구성되어있다고 한다.
다양한 사이즈의 커널을 multiple로 처리하는 방식은 Stride Convolution 방법보다 더 효율적으로 작업을 처리한다.
따라서 보다 최적으로 convolution layer 사이의 시간적 Feature를 탐색할 수 있다.
Introduction
부정맥 탐지는 일반적으로 전문가에 의해서 진단되어야 하지만 이거는 복잡하고 시간이 많이 소비되며, 사람이 직접 보고 진단하다보니 오류가 발생할 수 있음, 그리고 전문가가 부족함
대부분의 CNN 방식에서는 공통적인 문제를 겪고있음
- Noise가 너무 심하다
- Data Augmentation을 사용하더라도 ECG의 변화 패턴에 대해 제대로 포착하지 못함
- 매우 깊은 CNN을 사용해서 과적합, 계산복잡성을 증가시킴
Method
Pre-Processing
- Wavelet Based Denoising
여러 signal denoising 방법이 있지만, 그 중 wavelet Transformation 방식이 가장 denoising 효과가 좋음
- R-Peak Detection
R Peak Detection하는 방법 중에선 여러가지 방법이 있는데, 그 중 [1]번 방식을 채택함, 해당 방식이 가장 빠르고 정확하다고함
[1] M. Elgendi, ‘‘Fast QRS detection with an optimized knowledge-based method: Evaluation on 11 standard ECG databases,’’ PLoS ONE, vol. 8, no. 9, Sep. 2013, Art. no. e73557
[1]번 방식은 먼저 큰 값을 더 키우고, 고주파수 성분을 증폭하기 위해 noise가 제거된 신호가 제곱됨
그리고 two event-related moving average (TERMA) 방식으로 관심 블록을 생성해서 각각 QRS 특성을 추출하고 QRS Beat를 추출
각 생성된 블록에 threshold 적용 --> noise가 포함된 block으로부터 R-peak가 포함된 block을 분리하기하기위함
그리고 마지막으로 각 block의 절대값 중 max값이 R peak index라고 인식
- Beat Extraction
DL에 사용하기 위해선 심장박동 길이가 일정해야함
각 비트는 인접한 R-R interval의 중간지점에서 크롭하여 R peak를 중심으로 분할
경우에 따라 beat의 길이가 미리 지정한 길이보다 커지거나 작아질 수 있어서 R Peak를 중심으로 더 crop하거나 edge value를 사용해 padding이 수행됨
즉 인접한 R peak와의 중간 값
- n-1 index의 r peak와의 중간값
- n+1 index의 r peak와의 중간값
이 두 점의 길이가 Beat Length라고 할 수 있음
--> 그러나 이 길이가 지정한 길이보다 작거나 클 수 있는데 가장자리를 자르거나, padding을 씌워서 길이를 맞춤
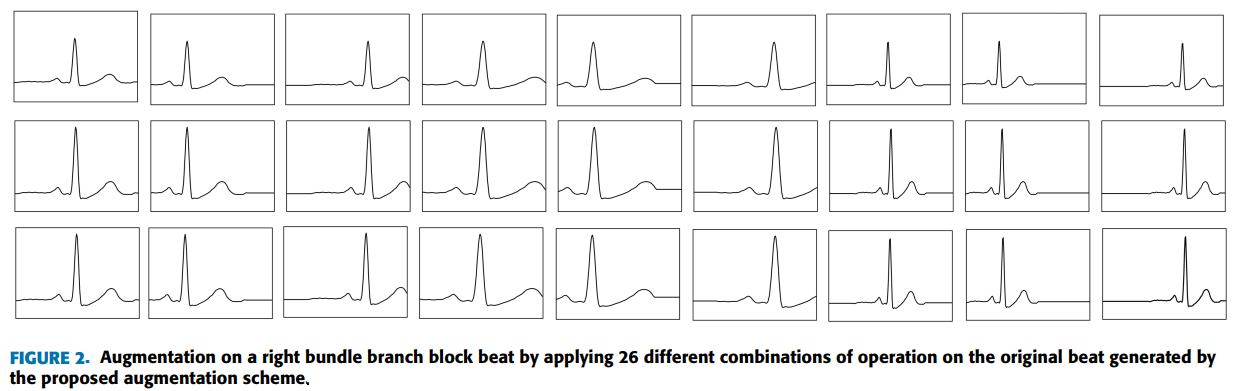
- Beat Augmentation
보다 현실적인 augmentation을 수행
Amplitude Scaling -> Time Scaling -> Shifting 을 거쳐서 Augmentation을 수행함
Amplitude Scaling에서는 데이터를 증폭, 감쇠 혹은 아무것도 하지 않음 --> 임의로 결정한 범위에서 랜덤으로 scaling factor를 결정
이렇게 Amplitude Scaling 거쳐 변화한 데이터는 좋은 augmented Beat를 제공함
Time Scaling은 amplitude scaling을 거친 데이터에서 팽창, 수축을 하는데
팽창시에는 oversampling을, 수축시에는 under-sampling을 수행함
마찬가지로 factor는 임의로 선택한 범위에서 랜덤으로 선택
특정 한도 내에서 널리 보이고(?), Beat의 원래 형태도 해치지않음
Shifting은 left, right shifting을 수행함
미리 정의된 범위에서 랜덤하게 샘플데이터의 개수가 선택됨
이 작업은 분류 작업에 어려움을 주지만.. global feature를 일반화 하기 위한 우선순위를 요구함? 이게뭔소리
- Data Normlization
[0,1] 범위로 min-max Normalization을 수행함
Proposed Model
- Depthwise Separable Convolution for 1D signal
일반적인 2D convolution을 사용하면 연산량이 기하급수적으로 늘어나고 Overfitting이 될 수 있음
Depthwise Separable Convolution (DSC) 기법은 공간적 convolution, 채널간 convolution을 개별적으로 진행함
우선적으로 각 채널별로 spatial Convolution을 각 채널별로 수행하고, conv를 수행한 채널을 다 합쳐서
1x1 convolution (pointwise convolution) 을 수행
이 convolution은 기존 conv보다 적은 수의 model parameter를 전달함
: input 데이터의 길이
: kernel의 사이즈
: Channel의 개수
Temporal Convolution은 의 데이터 길이와 만큼의 네트워크 파라미터를 가져서
Computational Cost는 를 가지게된다.
Pointwise Convolution은 만큼의 네트워크 파라미터를 가져서
Computational Cost는 를 가지게되어 Total Computational Cost는
커널이 클수록 채널 수가 증가함에 따라 1D 신호의 경우 네트워크 매개 변수 수를 줄이면서 Computational Cost를 크게 절감
- Structural Unit
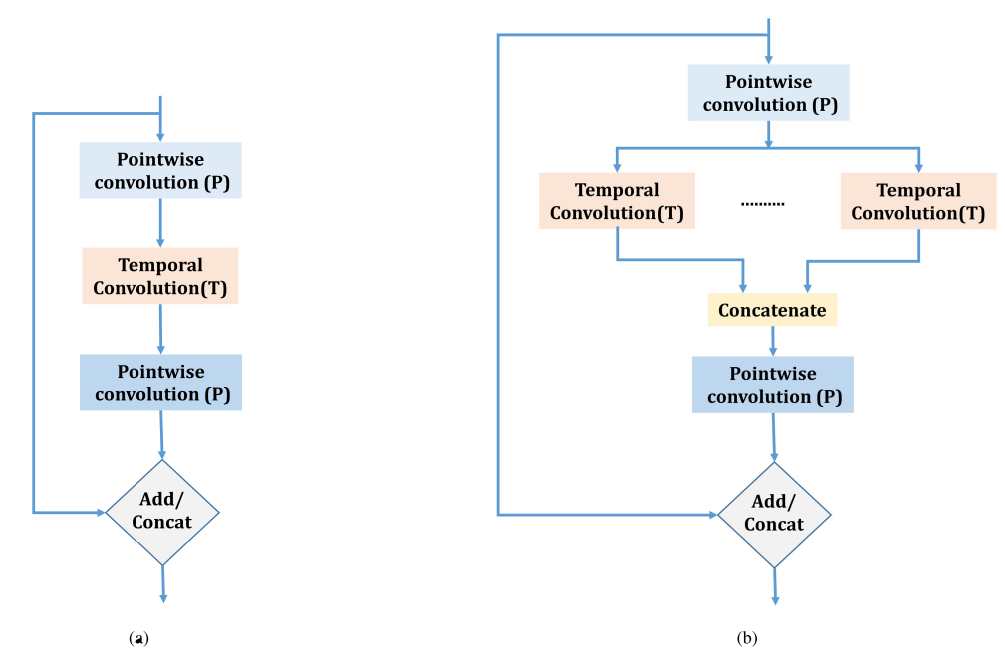
Structural Unit의 간단한 구조
PTP 구조
- Pointwise Convolution 1
채널 간 입력 데이터 정보를 결합, 채널 수가 더 증가한 더 큰 공간에 정보를 projection(?)
즉 더 넓은 공간으로 데이터를 맵핑시켜서 명확하게 분류를 하기위해 사용함
Pointwise가 Channel Reduction을 수행하는데, 여기선 Channel을 증가시킨다고한다.
이해가 안되긴함
- Temporal Convolution
각 채널로부터 시간적 정보를 capture하는 파트, Pointwise Convolution 이후 더 깊은 feature map에서 temporal Convolution을 수행함 --> Kernel 차원은 변경될 수 있음
Depthwise Separable Convolution 구조와 비슷함
--> Dpethwise Convolution에서 Feature map을 최대한 적게 뽑아내고, Pointiwse 단계에서 Feature map을 필요한 만큼 뽑아냄
- Pointwise Convolution 2
위 Temporal Convolution에서 수행한 다양한 채널들의 시간적 정보를 결합하고 추출된 Feature들을 다시 Pointwise Convolution을 하여 차원축소함
- Residual Path
Input Feature와 Extracted Feature와 결합 (Skip Conncection)
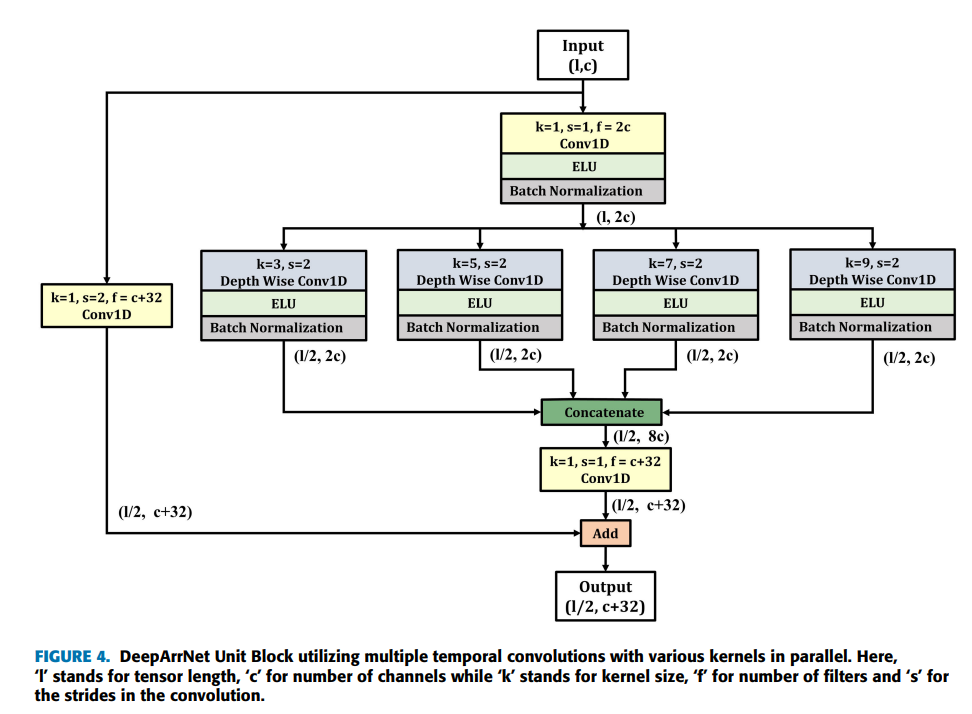
DeepArrNet Unit Block의 상세한 구조
input : (length, Channel)
위 구조를 보면 Pointwise Convolution (Conv 1D)를 거친다. 필터의 개수가 2c개 이므로 최종적으로 만들어지는 채널의 개수는 2c 개이다.
Temporal Convolution에서 각기 다른 Kernel Size로 Depthwise Convolution를 병렬로 수행한다. 그리고 stride를 2로 사용해서 계산복잡도를 줄인다.
그리고 병렬로 수행된 Depthwise Convolution의 결과들을 concat한다 (합친다). 이때 4개의 Convolution 결과를 합친 것이므로 채널은 4배가 된다 --> 8c, 그리고 stride가 2이므로 로 줄어든다.
concat 후, 다시 Pointwise Convolution (Conv 1D)를 거쳐서 c+32 개의 채널을 생성하는데, 32개인 이유는 뒤에서 설명
마지막으로 Residual Path , Skip connection으로 pointwise를 통과한 input Feature map을 더해서 최종 output (Length/2 , Channel+32) 를 output으로 결과를 산출
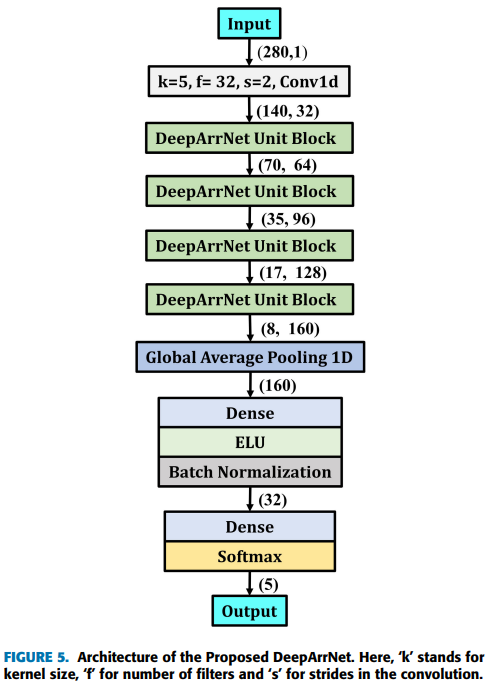
해당 구조에서 input은 (length=280, channel=1)로 입력 값을 주고
Kernel=5, Filter=32 , stride=2 인 Standard Convolution 1D를 거쳐 (length=140, channel=32)인 feature map을 산출한다
그리고 4개의 DeeArrNet Unit Block을 거쳐서 length는 절반씩, Channel의 수는 32씩 증가시켜 최종적으로 8,160이 되고 이를 GAP(Global Average Pooling)을 거쳐서 Feature들을 1차원 벡터로 만들고 Dense Layer를 지나 softmax를 사용해서 총 5가지의 상태의 확률을 출력을 낸다.
이러한 multiple parallel Temporal Convolution을 사용하여 보다 다양한 특징 추출을 위한 최적의 성능 제공
그리고 데이터에서 미세한 class들간의 차이들을 효과적으로 구별할 수 있다고한다.
- Optimization and Learning Technique
Activation Function
ELU & Softmax
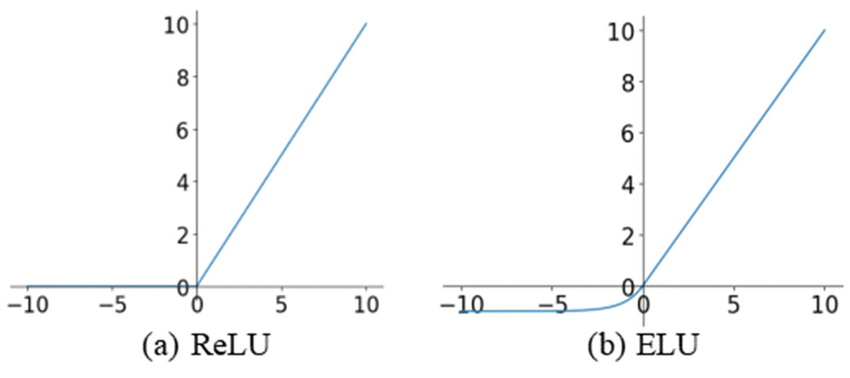
이 논문에서는 로 설정하였으며 더 빨리 수렴하도록 함
그리고 Softmax 함수를 사용해서 0~1 사이값으로 가장 근접한 클래스는 1에 가까운 값으로 출력한다.
Kernel Initialization
Xavier Initialization
He-Normal initialization
이 두 초기화기법은 최근 딥러닝 네트워크에서 광범위하게 사용됨
ReLU함수와 비슷한 ELU 활성화함수를 사용하므로 He-Normal initialization 기법을 사용함
Xavier initialization은 ReLU같은 활성화함수를 사용할 때 비효율적인 결과를 보이기도함
그래서 He-Normal initialization을 사용함
Learning Methodology
흔히 분류 모델에서는 Cross-Entropy cost function을 널리 사용함, 해당 논문에서도 Cross-entropy를 사용하였다
optimizer : Adam optimizer (lr = 0.001), Batch size = 64
Batch Normalization은 각 배치별로 평균과 분산을 이용해서 데이터를 정규화시킵니다.
그래서 각 unit(block)마다 Batch Norm이 붙어있습니다.
Result and Discussion
MIT BIH Dataset은 N,S,V,F,Q..그 외 디테일하게 class를 나눌 수 있는데, 본 실험에서는 5가지의 카테고리를 사용했다고 한다.
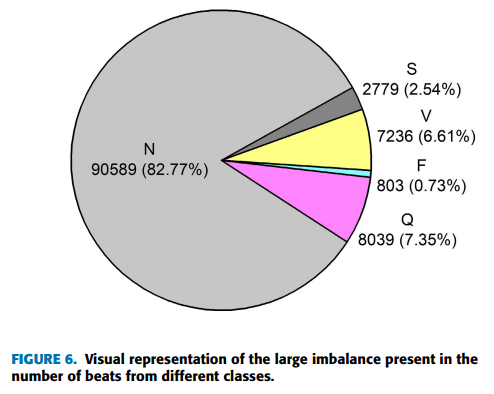
Ectopic Beat : 이전의 정상 cardiac Cycle보다 먼저 발생하는 박동을 의미함
N : Normal
S : Supraventricular Ectopic Beat - 심실 위쪽에서 발생하는 비정상 심장박동
V : Ventricular Ectopic Beat - 심실에서 발생하는 비정상 심장박동
F : Fusion Beat - 이소성(비정상) 박동과 정상박동이 융합되어 나타나는 심장박동
Q : Unknown Beat - 알 수 없는 심장박동
Experimentation
10-fold-cross-validation을 사용하였고, 평가수치는 일반적으로 사용하는
Accuracy, Sensitivity, Positive Predictive Value, F1 Score를 사용하였다고 한다.
Result
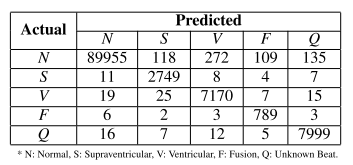
Confusion matrix는 다음과 같이 나타낸다.
그에 따른 결과를 정리하면
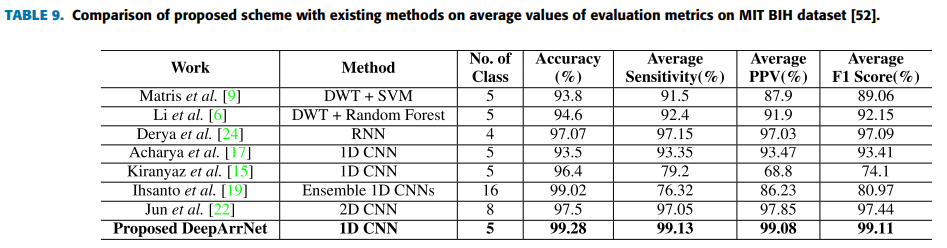
다음과 같이 나타낼 수 있다.
5개 class의 분류에 대해서 Accuracy는 99.28%, 평균 Sensitivity는 99.13%, 평균 PPV 99.08%, 평균 F1 Score는 99.11%로 나타낼 수 있다.
해당 논문에서 제시한 방법은 R Peak 감지에 민감하다. robustness를 증가시키기 위해선 보다 까다로운 경우에서 해당 모델 성능을 제시해야한다고 한다.
Conclusion
1D 영역에서 Depthwise Separable Convolution을 활용한 PTP Convolution을 수행하는 Unit Block을 기반으로하는 아키텍쳐이다.
Wavelet Decomposition 기술을 사용하여 Denoising, 그리고 Data Augmentation을 사용하여 보다 현실적인 데이터를 증강시켜 데이터의 불균형을 최소화 시켰다고한다.
다양한 Kernel size를 가진 PTP Block을 사용하였고 Residual Path를 사용해 Feature들을 결합시켰다.
또한 다양한 Temporal window를 병렬로 활용하는 동안에 Strided Convolution을 사용하여 Feature map을 감소시켰다.
SOTA급의 성능을 제공하고, 경량모델이라고 한다.
