위키북스의 파이썬 머신러닝 완벽 가이드 책을 토대로 공부한 내용입니다.
1. GMM(Gaussian Mixture Model) 소개
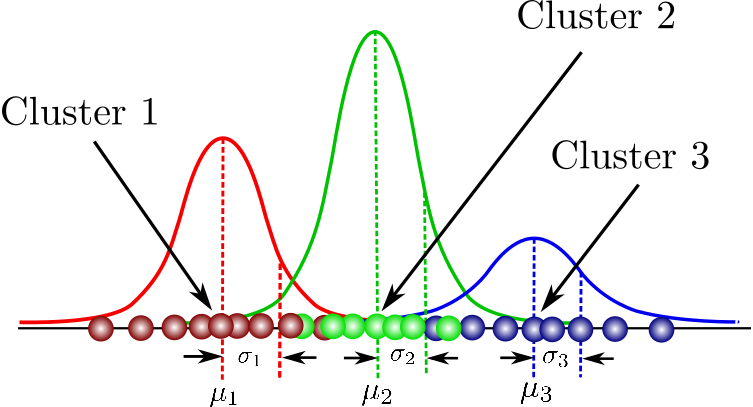
GMM 군집화는 군집화를 적용하고자 하는 데이터가 여러 개의 가우시안 분포를 가진 데이터 집합들이 섞여서 생성된 것이라는 가정하에 군집화를 수행하는 방식이다. 정규 분포(Normal distribution)로도 알려진 가우시안 분포는 좌우 대칭형의 종(Bell) 형태를 가진다. 정규 분포는 평균 를 중심으로 높은 데이터 분포도를 가지고 있으며, 좌우로 표준편자만큼의 범위안에 전체 데이터의 68.27%를, 좌우로 표준편차의 2배만큼의 범위안에 전체 데이터의 95.45%를, 3배 만큼에는 99.37%를 가지고 있다. 그리고 평균이 0이고, 표준 편차가 1인 정규 분포를 표준 정규 분포라고 한다.
GMM은 데이터를 여러 개의 가우시안 분포가 섞인 것으로 간주하여 섞인 데이터 분포에서 개별 유형의 가우시안 분포를 추출한다. 따라서 전체 dataset은 서로 다른 정규 분포 형태를 가진 여러가지 확률 분포 곡선으로 구성될 수 있으며, 이러한 서로 다른 정규 분포에 기반해 군집화를 수행하는 것이 GMM 군집화 방식이며, 각각의 개별 데이터가 어떤 정규 분포에 속하는지 결정하는 방식이다. 이와 같은 방식은 GMM에서는 모수 추정이라고 하는데, 모수 추정은 대표적으로 개별 정규 분포의 평균과 분산, 각 데이터가 어떤 정규 분포에 해당되는지의 확률을 추정하는 것이다. 이러한 모수 추정을 위해 GMM은 EM(Expectation and Maximization) 방법을 적용한다. 사이킷런은 GMM의 EM 방식을 통한 모수 추정 군집화를 지원하기 위해 GaussianMixture 클래스를 지원한다.
2. GMM을 이용한 iris dataset 군집화
GMM은 확률 기반 군집화이고 K-평균은 거리 기반 군집화이다. iris dataset으로 두가지 방식의 군집화를 수행한 뒤 비교해보겠다. GaussianMixture 객체의 가장 중요한 초기화 파라미터는 n_component로 gaussian mixture 모델의 총 개수이다.
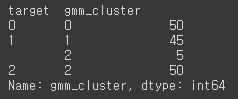
from sklearn.datasets import load_iris from sklearn.cluster import KMeans from sklearn.mixture import GaussianMixture import matplotlib.pyplot as plt import numpy as np import pandas as pd %matplotlib inline iris = load_iris() feature_names = ['sepal_length','sepal_width','petal_length','petal_width'] # 보다 편리한 데이타 Handling을 위해 DataFrame으로 변환 irisDF = pd.DataFrame(data=iris.data, columns=feature_names) irisDF['target'] = iris.target gmm = GaussianMixture(n_components=3, random_state=0).fit(iris.data) gmm_cluster_labels = gmm.predict(iris.data) # 클러스터링 결과를 irisDF 의 'gmm_cluster' 컬럼명으로 저장 irisDF['gmm_cluster'] = gmm_cluster_labels irisDF['target'] = iris.target # target 값에 따라서 gmm_cluster 값이 어떻게 매핑되었는지 확인. iris_result = irisDF.groupby(['target'])['gmm_cluster'].value_counts() print(iris_result) kmeans = KMeans(n_clusters=3, init='k-means++', max_iter=300,random_state=0).fit(iris.data) kmeans_cluster_labels = kmeans.predict(iris.data) irisDF['kmeans_cluster'] = kmeans_cluster_labels iris_result = irisDF.groupby(['target'])['kmeans_cluster'].value_counts() print(iris_result)[output]
Target 0과 2는 모두 잘 매핑되었지만, Target 1은 cluster 1과 2로 약간 분산되어 매핑되었고, iris dataset의 K-평균 군집화 결과보다 더 효과적인 분류 결과를 도출하였다. 하지만 어떤 알고지름이 더 뛰어나다는 의미를 가지지는 못하고 iris dataset이 GMM 군집화에 더 효과적이라는 의미를 가지게 된다. K-평균은 평균 거리 중심으로 중심을 이동하면서 군집화를 수행하는 방식이므로 개별 군집 내의 데이터가 원형으로 흩어져 있는 경우에 매우 효과적으로 군집화가 수행될 수 있다.
3. GMM과 K-평균의 비교
KMeans는 원형의 범위에서 군집화를 수행하기 때문에 dataset이 원형의 범위를 가질수록 군집화 효율은 더욱 높아진다. 따라서 make_blobs()을 이용하여 3개의 군집으로 군집 내의 데이터가 뭉쳐있도록 생성한 dataset에 K-평균을 적용해보겠다.
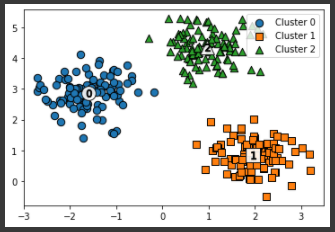
### 클러스터 결과를 담은 DataFrame과 사이킷런의 Cluster 객체등을 인자로 받아 클러스터링 결과를 시각화하는 함수 def visualize_cluster_plot(clusterobj, dataframe, label_name, iscenter=True): if iscenter : centers = clusterobj.cluster_centers_ unique_labels = np.unique(dataframe[label_name].values) markers=['o', 's', '^', 'x', '*'] isNoise=False for label in unique_labels: label_cluster = dataframe[dataframe[label_name]==label] if label == -1: cluster_legend = 'Noise' isNoise=True else : cluster_legend = 'Cluster '+str(label) plt.scatter(x=label_cluster['ftr1'], y=label_cluster['ftr2'], s=70,\ edgecolor='k', marker=markers[label], label=cluster_legend) if iscenter: center_x_y = centers[label] plt.scatter(x=center_x_y[0], y=center_x_y[1], s=250, color='white', alpha=0.9, edgecolor='k', marker=markers[label]) plt.scatter(x=center_x_y[0], y=center_x_y[1], s=70, color='k',\ edgecolor='k', marker='$%d$' % label) if isNoise: legend_loc='upper center' else: legend_loc='upper right' plt.legend(loc=legend_loc) plt.show() from sklearn.datasets import make_blobs # make_blobs() 로 300개의 데이터 셋, 3개의 cluster 셋, cluster_std=0.5 을 만듬. X, y = make_blobs(n_samples=300, n_features=2, centers=3, cluster_std=0.5, random_state=0) # feature 데이터 셋과 make_blobs( ) 의 y 결과 값을 DataFrame으로 저장 clusterDF = pd.DataFrame(data=X, columns=['ftr1', 'ftr2']) # 3개의 Cluster 기반 Kmeans 를 X_aniso 데이터 셋에 적용 kmeans = KMeans(3, random_state=0) kmeans_label = kmeans.fit_predict(X) clusterDF['kmeans_label'] = kmeans_label visualize_cluster_plot(kmeans, clusterDF, 'kmeans_label',iscenter=True)[output]
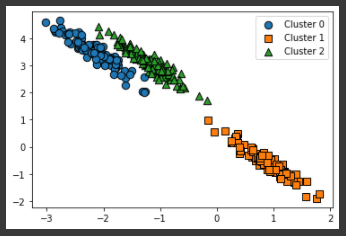
위 이미지와 같이 KMeans 군집화는 원형의 범위로 퍼져있는 데이터에 대해 군집화를 잘 수행해낸다. 하지만 데이터가 원형의 범위로 퍼져 있지 않고 길쭉한 타원형으로 늘어선 경우에는 군집화를 잘 수행하지 못한다.# 길게 늘어난 타원형의 데이터 셋을 생성하기 위해 변환함. transformation = [[0.60834549, -0.63667341], [-0.40887718, 0.85253229]] X_aniso = np.dot(X, transformation) clusterDF = pd.DataFrame(data=X_aniso, columns=['ftr1', 'ftr2']) # 3개의 Cluster 기반 Kmeans 를 X_aniso 데이터 셋에 적용 kmeans = KMeans(3, random_state=0) kmeans_label = kmeans.fit_predict(X_aniso) clusterDF['kmeans_label'] = kmeans_label visualize_cluster_plot(kmeans, clusterDF, 'kmeans_label',iscenter=True)[output]
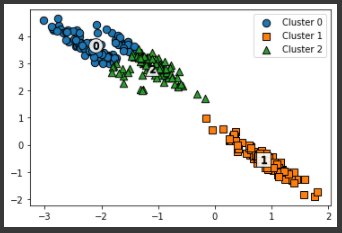
KMeans로 군집화를 수행할 경우, 주로 원형 영역 위치로 개별 군집화가 되면서 원하는 방향으로 구성되지 않는 것을 알 수 있다. KMeans가 평균 거리 기반으로 군집화를 수행하므로 같은 거리상 원형으로 군집을 구성하면서 위와 같이 길쭉한 방향으로 데이터가 밀집해 있을 경우에는 최적의 군집화가 어렵다.# 3개의 n_components기반 GMM을 X_aniso 데이터 셋에 적용 gmm = GaussianMixture(n_components=3, random_state=0) gmm_label = gmm.fit(X_aniso).predict(X_aniso) clusterDF['gmm_label'] = gmm_label # GaussianMixture는 cluster_centers_ 속성이 없으므로 iscenter를 False로 설정. visualize_cluster_plot(gmm, clusterDF, 'gmm_label',iscenter=False)[output]
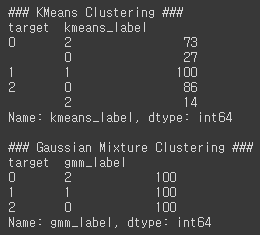
GMM으로 군집화를 수행해보면 데이터가 분포된 방향에 따라 정확하게 군집화가 된 것을 알 수 있다.print('### KMeans Clustering ###') print(clusterDF.groupby('target')['kmeans_label'].value_counts()) print('\n### Gaussian Mixture Clustering ###') print(clusterDF.groupby('target')['gmm_label'].value_counts())[output]
위 결과에서 알 수 있듯이 GMM의 경우는 KMeans보다 유연하게 다양한 dataset에 잘 적용될 수 있다는 장점이 있다. 하지만 군집화를 위한 수행 시간이 오래 걸린다는 단점이 있다.