이상치 처리 방법론
- 이상치를 제거하는 방법은 여러가지가 존재한다.
- 다양한 방법중에서 아래 진행하는 과정은 데이터를 분석함수로써 모델링하는 방법이다.
- 예를 들면 정규분포(=가우스 분포)로 특정 확률보다 작은 모든 값들을 찾고 제거하는 과정을 확신이 들때 까지 반복하는 과정이다.
직접 구현
import numpy as np
import matplotlib.pyplot as plt
from scipy.stats import norm
from scipy.stats import multivariate_normal as mn
from sklearn.neighbors import LocalOutlierFactor
d1 = np.loadtxt("outlier_1d.txt")
d2 = np.loadtxt("outlier_2d.txt")
d3 = np.loadtxt("outlier_curve.txt")
print(d1.shape, d2.shape, d3.shape)
# 결과
(1010,) (1010, 2) (1000, 2)d1의 경우 1차원, d2, d3는 2차원 데이터
plt.scatter(d1, np.random.normal(7,0.2,size=d1.size),s=1, alpha=0.5)
plt.show()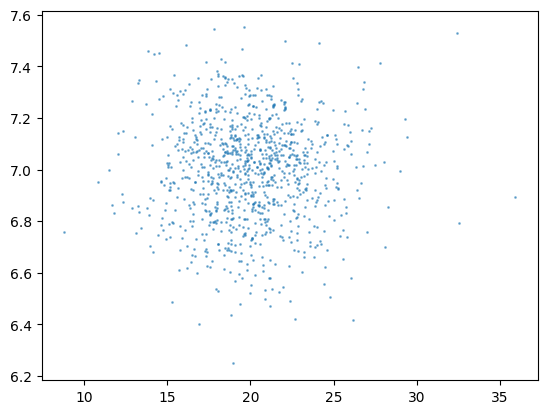
plt.scatter(d2[:,0],d2[:,1])
plt.show()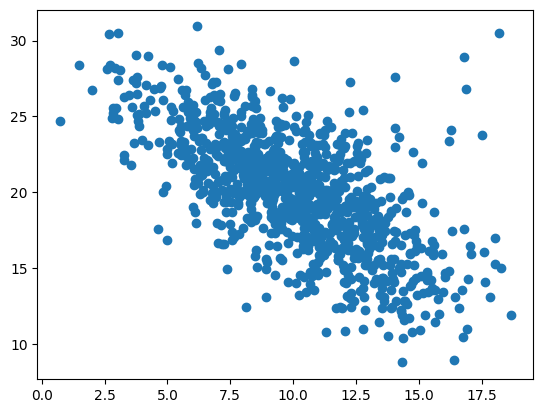
plt.plot(d3[:,0],d3[:,1])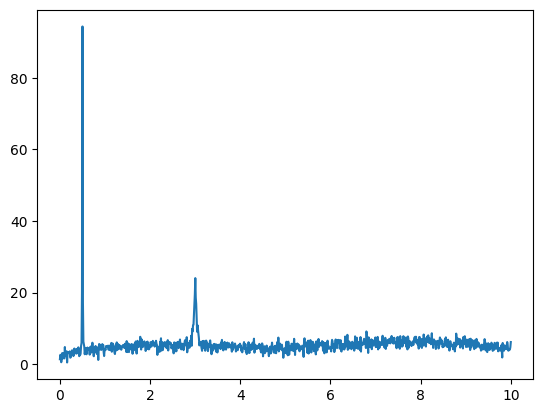
위의 3개의 값 d1, d2, d3 이상치 확인 방법을 아래에서 실행예정
# 기본적으로 데이터가 정규분포를 따른다고 가정
mean, std = np.mean(d1), np.std(d1)
z_score = np.abs((d1-mean)/std)
threshold = 3 # z-score가 3 이상이면 모두 제거하도록 임계값 설정(변경하면서 진행)
good = z_score < thresholdZ-Score : 각 데이터 값이 평균으로부터 얼마나 떨어져 있는지를 나타내는 표준편차 숫자이다. Z-Score가 0이면 정확히 평균에 해당한다.
print(f"Rejection {(~good).sum()} points")
print(f"z-score of 3 corresponds to a prob of {100 * 2 * norm.sf(threshold):0.2f}%")
visual_scatter = np.random.normal(size=d1.size)
plt.scatter(d1[good], visual_scatter[good], s=2, label="Good", color = "#4CAF50")
plt.scatter(d1[~good], visual_scatter[~good], s=2, label="Bad", color = "#F44336")
plt.legend()# result
Rejection 5 points
z-score of 3 corresponds to a prob of 0.27%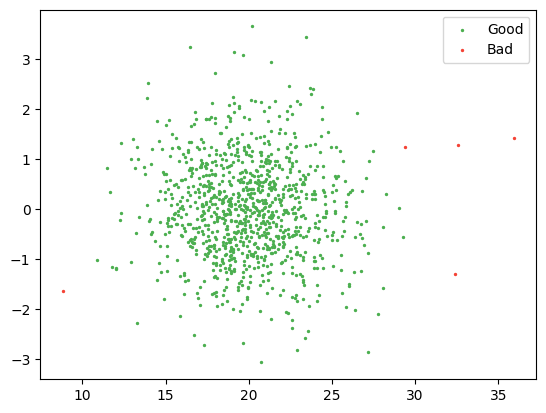
여기서 5개의 점이 z-score로 설정한 3보다 크기 때문에 Bad로 표현된다.
# d2의 분포를 2차원 가우스로 정량화
mean, cov = np.mean(d2, axis=0), np.cov(d2.T)
good = mn(mean, cov).pdf(d2) > 0.01 / 100
- scipy.stats에서 다변수정규분포를 위한 multivariate_normal() 함수 제공
- pdf는 특정 구간에 속할 확률을 계산하기 위한 함수
plt.scatter(d2[good,0], d2[good,1], s=2, label="Good", color = "#4CAF50")
plt.scatter(d2[~good, 0], d2[~good,1], s=2, label="Bad", color = "#F44336")
plt.legend()
plt.show()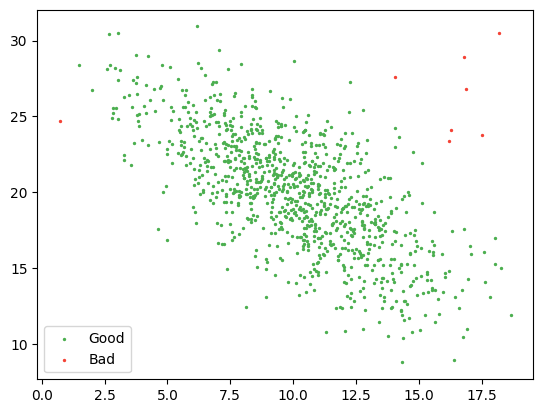
여기서도 몇 개의 데이터들이 good으로 설정한 값보다 크므로 Bad로 표현 된다.
xs, ys = d3.T
p = np.polyfit(xs, ys,deg=5)
ps = np.polyval(p, xs)
- numpy.polyfit 함수는 주어진 데이터에 대해 최소 제곱을 갖는 다항식 피팅을 반환한다. -> 위에서는 5차원 다항식을 출력
- numpy.polyval 함수는 입력 배열을 다항식으로 간주하고, 입력값을 넣어 계산된 값을 반환한다.
plt.plot(xs, ys, ".", label="Data")
plt.plot(xs, ps, label="poly fit")
plt.legend()
plt.show()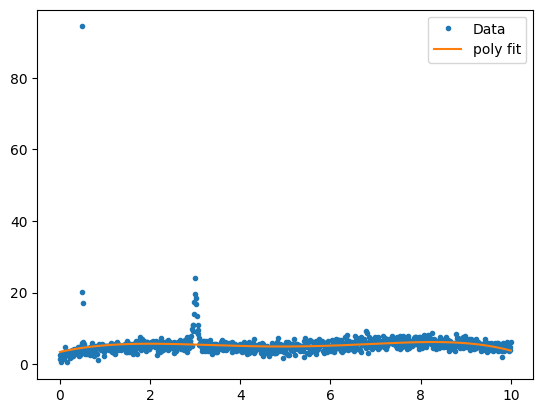
주황색 선이 데이터로 예측한 5차 다항식으로 표현이 된다.
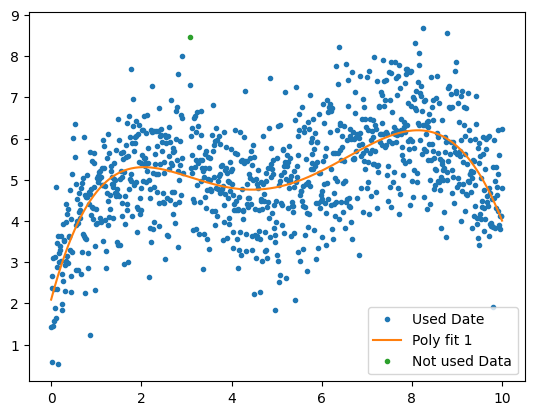
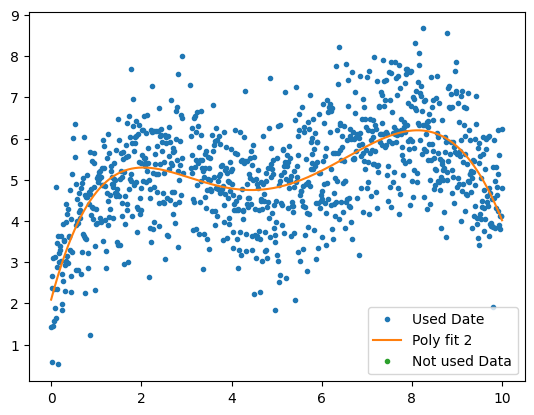
- 처음에 언급한 부분처럼 이제 위에서 했던 방식을 for문을 통해 반복이 가능하다.
x, y = xs.copy(), ys.copy()
for i in range(5):
p = np.polyfit(x,y,deg=5)
ps = np.polyval(p,x)
good = y-ps < 3 # only remove positive outliers
x_bad, y_bad = x[~good], y[~good]
x,y = x[good], y[good]
plt.plot(x,y,".", label = "Used Date")
plt.plot(x,np.polyval(p,x), label=f"Poly fit {i}")
plt.plot(x_bad, y_bad, ".", label="Not used Data")
plt.legend()
plt.show()
if (~good).sum() == 0:
break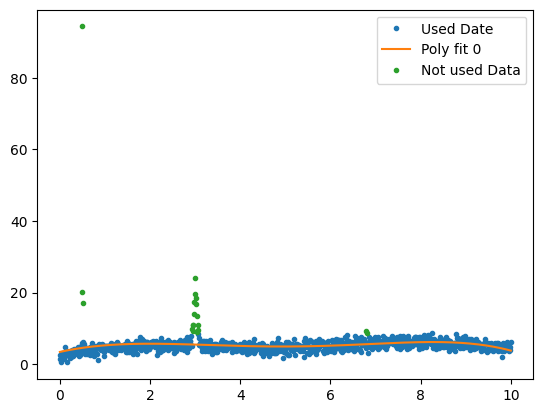
라이브러리 활용
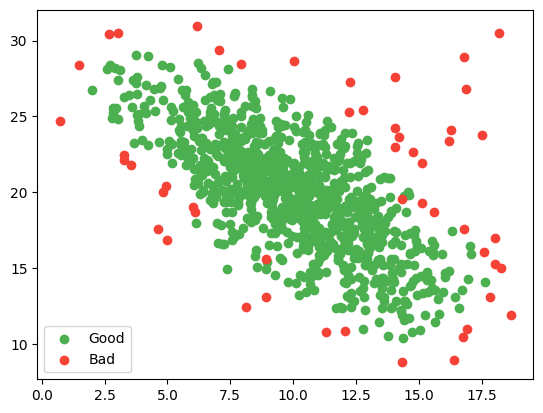
- 직접구현 하는 방법도 존재하지만, 실제로는 sklearn에 구현이 되어 있다.(링크)
lof = LocalOutlierFactor(n_neighbors=20, contamination=0.05)
good = lof.fit_predict(d2) == 1
plt.scatter(d2[good, 0], d2[good ,1], label="Good", color="#4CAF50")
plt.scatter(d2[~good, 0], d2[~good ,1], label="Bad", color="#F44336")
plt.legend()LocalOutlierFactor 내부의 contamination의 경우에는 위에서 설정한 임계값을 설정해주는 변수이다.
- 참고 : 유데미 - 【한글자막】 Python : 통계 분석을 위한 파이썬
