[Review] Convolutional Neural Networks for Classification of Alzheimer’s Disease: Overview and Reproducible Evaluation
Review

❗️혼자 읽고 공부한 것을 정리한 글로, 내용상 오류가 있을 수 있습니다❗️
1. Introduction
- AD 진단의 목적
Alzheimer's disease(AD) 환자를 돌보는 데 사용되는 비용이 막대하기 때문에 AD를 초기에 정확하게 진단하는 컴퓨터를 활용한 개인별 시스템이 필요함
- 기존의 ML 방식 vs. DL(CNN)
기존의 ML 방식은 SVM이나 Random Forest 같이 다른 종류의 분류기가 결합된 이미지 전처리 파이프라인으로부터 특징을 미리 정의하는 방법을 제안했지만,
CNN과 같은 DL 방식으로 low-to-high 수준까지 잠재 특징 표현을 자동으로 추상화할 수 있게 되면서 이미지 전처리나 특징 추출과 같은 이전 단계들이 비교적 간단해졌음SVM
결정 경계(Decision Boundary)를 위한 기준 선을 정의하는 모델
결정 경계를 정의하는 것이 서포트 벡터이기 때문에 여러 데이터 포인트 중 서포트벡터만 잘 골라내면 나머지 데이터들은 무시할 수 있어 매우 빠름
⇒ 데이터 군으로부터 최대한 멀리 떨어지는 게 좋음 (= margin이 최대)- Margin : 결정 경계와 서포트 벡터 사이의 거리
- Support Vectors : 결정 경계와 가까이 있는 데이터 포인트들
- 이전 연구
전통적인 ML 방식을 사용한 AD 분류를 평가하는 오픈 소스 프레임워크를 제안함
이 프레임워크는 ADNI, AIBL, OASIS 데이터를 BIDS 형태로 자동으로 변환해줌
+) PET, T1-weighted(T1w) MRI, diffusion MRI와 같은 형태의 데이터에도 이 프레임워크 사용 가능
-
이 연구가 기여한 점
-
AD 분류를 위해 CNN과 해부학적 MRI를 사용한 각각 다른 연구들을 요약함
(검증 단계와 data leakage 존재 가능성 위주) -
오픈소스 프레임워크를 DL을 사용한 AD 분류까지 확장함
- DL 기반 이미지 전처리, 분류 모델, 평가 단계에 대한 modular set을 보충하는 방식
⇒ 다른 구조를 가진 CNN 모델 간 성능을 엄밀하게 평가 가능
- DL 기반 이미지 전처리, 분류 모델, 평가 단계에 대한 modular set을 보충하는 방식
-
분류 정확도에 영향을 주는 주요 구성 요소 찾기 & CNN의 일반화 능력 평가
1) CNN과 기존 ML(선형 SVM) 간 비교하는 방식
2) ADNI로 학습하고 테스트한 모델, ADNI로 학습하고 AIBL이나 OASIS로 테스트한 모델
-
2.State of the art
이 연구는 여러 연구 중 이미지 데이터에 대해 end-to-end CNN을 적용한 32개의 연구들을 리뷰함
end-to-end Network
모델의 모든 매개변수가 하나의 손실함수에 대해 동시에 훈련되는 경로가 가능한 네트워크
신경망의 한 쪽 끝에서 입력을 받아들이고 다른 쪽 끝에서 출력을 생성하는데, 입력 및 출력을 직접 고려하여 네트워크 가중치를 최적화하는 학습 방식
입력에서 출력까지 파이프라인 네트워크 없이 신경망으로 한 번에 처리함
충분히 라벨링된 데이터가 있으면 신경망 모델로 해결 가능함
⇒ 사람이 직접 특징 추출 과정을 수행하지 않아도 됨
- 파이프라인 네트워크 : 전체 네트워크를 이루는 부분적인 네트워크
2.1 Main classification taks
AD 진단은 아래와 같이 여러 개의 분류 기준이 존재함
- AD vs. CN
- CN vs. MCI
- AD vs. MCI
- sMCI vs. pMCI
- 다중 분류
- MCI : 치매 진단 전 단계로, 가벼운 인지 장애를 가진 사람
- sMCI : MCI 단계를 유지하는 사람
- pMCI : AD가 진행될 사람
- MCI : 치매 진단 전 단계로, 가벼운 인지 장애를 가진 사람
2.2 Main causes of data leakage
Data leakage
: test data가 traing 과정 중 사용된 경우
DL은 모델이 복잡하고 유연하기 때문에 이러한 data leakage를 발견하기 어려움
- 원인
- Wrong data split
subject-level에서 데이터셋이 분류되지 않은 경우로,
같은 subject(환자)의 데이터가 각각 train/validation/test data로 구분되어 있음을 의미함
→ 3D 이미지로부터 patch나 slice를 추출할 때 발생할 수 있음
- Late split
Data augmentation, feature selection, AutoEncorder와 같은 과정은 test 데이터를 절대 사용하지 않도록 train/test data 분류 후 실행되어야 하는데 그렇지 않은 경우
- Biased transfer learning
source와 target의 도메인(범위)가 겹치는 경우로, 전이학습 시 사용한 train data가 이후 target 모델의 test data로 쓰일 수 있음
ex) AD / CN 분류(source task)를 위해 사전 학습된 모델이 MCI / CN 분류(target task) 문제에 사용되는 경우
- Absence of an independent test set
test data는 최종 성능을 평가할 때만 사용되어야 하는데 그렇지 않은 경우
→ 하이퍼 파라미터 최적화와 같은 과정은 train/validation data를 사용해야 함
이 연구에서는 32개의 연구에 대해 data leakage 여부에 따라 3가지 카테고리로 분류함
- Claer : data leakage가 확실하게 발견된 경우
- Unclear : data leakage가 발생했다는 충분한 증거가 없는 경우
- None detected : data leakage가 발생하지 않은 경우
2.3 Classification of AD with end-to-end CNNs
이 연구는 유클리드 공간에 대해 end-to-end CNN을 적용한 연구들을 다룸
2.3.1 2D slice-level CNN
⇒ 3D MRI로부터 2D 슬라이스를 추출하여 입력으로 사용함
- 장점
- ResNet, VGGNet과 같이 이미 좋은 성능이 보장된 모델을 사용해 전이학습이 가능함
- 3D MRI로부터 수많은 2D 슬라이스를 추출할 수 있어 traing data의 수가 늘어남
- 문제점
- MRI는 3차원인 반면, 2D conv filter는 한 환자에 대한 slice를 모두 독립적으로 분석함
- 입력으로 사용할 슬라이스를 선택하는 방법이 매우 다양하기 때문에 slice-level 정확도와 subject-level 정확도가 혼동될 수 있음
- (Valliani and Soni, 2017)
- data leakage가 없고, ResNet과 기존 CNN(conv layer 1개, FC layer 2개) 간 비교를 진행함
- ImageNet 기반 사전학습 모델 사용
- affine 변환을 통한 data augmentation
사전학습과 data augmentation이 ResNet 정확도 향상에 도움됨
- 나머지 연구들
- data leakage 발생 & 불균형 데이터로 인해 불균형 지표가 계산되어 평가 단계에서 오류가 발생함
- 연구 간 슬라이스 선택에 대한 차이도 존재함
- 주어진 평면에 대한 모든 슬라이스를 사용하는 경우
- 자동으로 일부 슬라이스만 선택하여 사용하는 경우
- 한 환자에 대해 오직 한 슬라이스만 사용하는 경우
2.3.2 3D patch-level CNN
⇒ 입력으로 2D 데이터를 사용하는 경우의 한계를 보완하기 위해 구상됨
- 장점
- train data의 수가 증가함
- 메모리 사용량이 적음
- (모든 patch에 대해 같은 모델을 사용할 경우) 학습시킬 파라미터의 수가 적음
- 문제점
- patch에 대한 크기와 간격을 필수로 선택해야 함
- 모델이 복잡함
( 각 patch마다 하나의 모델을 학습시키고 이들을 결합해 subject-level로 재학습시킴)
- (Cheng et al., 2017; Manhua Liu et al., 2018)
- 100 X 81 X 80 voxels의 전체 MRI로부터 50 X 41 X 50 voxels의 overlapping patch를 27개 추출함
- 하나의 patch마다 하나의 CNN(conv layer 4개, FC layer 2개) 학습시킴
- 이 CNN은 subject-level 분류에도 사용되었음
- (Li et al., 2018)
-
32 X 32 X 32 크기의 patch 사용함
(patch 간 가능한 불일치를 고려하여 patch의 크기 줄임) -
subject(환자) 간 불일치를 피하기 위해 patch를 k-means로 군집화함
K-means
K개의 클러스터로 데이터를 분류하는 알고리즘- Means : 각 데이터로부터 그 데이터가 속한 클러스터의 중심까지의 평균 거리
⇒ 이 값을 최소화하는 것이 K-means의 목표
- K개의 임의의 중심점(centroid)를 배치한다
- 각 데이터들을 가장 가까운 중심점으로 할당하여 군집을 생성한다
- 군집으로 지정된 데이터들을 기반으로 해당 군집의 중심점을 업데이트한다
- 더이상 중심점이 업데이트되지 않을 때까지 2, 3번 과정을 반복한다
- Means : 각 데이터로부터 그 데이터가 속한 클러스터의 중심까지의 평균 거리
-
각 patch 군집마다 하나의 CNN을 학습시키고 얻은 cluster-level의 특징을 결합함
-
- (Lian et al., 2018; Mingxia Liu et al., 2018a, 2018c)
- 19 X 19 X 19 크기의 해부학적 지표를 가진 patch들만 선정하여 사용함
(AD/CN 그룹 비교를 통해 이미 알려진 지표 기준으로 선정함)- Mingxia Liu et al., 2018c
각 patch에 대해 사전 학습된 CNN 사용하였고, 결과는 subject 진단을 위해 결합됨 - Mingxia Liu et al., 2018a
patch는 진단 라벨을 붙일 수 없다 생각함
→ 각 patch마다 하나의 CNN을 사용하는 대신 ensemble 모델을 사용함 - (Lian et al., 2018)
약한 수준의 지도학습 방식을 제시함
→ patch-level 모델의 loss가 subject-level에서의 최종 분류 점수에 기반함
- Mingxia Liu et al., 2018c
- 19 X 19 X 19 크기의 해부학적 지표를 가진 patch들만 선정하여 사용함
2.3.3 ROI-based CNN
3D patch가 AD와 관련 없는 뇌의 영역까지 포함하기 때문에 대부분 유익하지 않음
⇒ Hippocampus(해마)와 같이 AD와 관련된 뇌의 영역에만 집중하는 방식
-
장점
- 모델을 학습시킬 때 사용되는 input이 줄어 모델 복잡도가 줄어듦
-
단점
- AD의 변화는 뇌의 여러 영역에 걸쳐있는데 ROI는 해마만 이용함
⇒ overfit 위험을 줄일 수 있음
( 입력 patch의 수가 비교적 적음)
- AD의 변화는 뇌의 여러 영역에 걸쳐있는데 ROI는 해마만 이용함
- (Aderghal et al., 2018, 2017a, 2017b)
- 해마 주변 3개의 2D-slice를 결합하여 만든 3D-patch 이용함
- 사구체 관점에서 하나의 patch를 분류하여 CNN(conv layer 2개, FC layer 1개) 학습시킴
- 사구체/관상평면/축삭평면 관점에서 3개의 patch 분류하여 각각 학습시키고 결합함
- 해마 주변 3개의 2D-slice를 결합하여 만든 3D-patch 이용함
- (Lin et al., 2018)
- 비선형 방식으로 subject(환자) 간 voxel 일치성을 파악함
2.3.4 3D subject-level CNN
- 장점
- 공간 정보가 완전히 통합됨
- 단점
- 데이터의 수가 다른 방법에 비해 적기 때문에 overfit 위험
( 하나의 환자 = 하나의 데이터)
- 데이터의 수가 다른 방법에 비해 적기 때문에 overfit 위험
- 기존 CNN을 재조정한 연구
- ResNet, VGGNet과 같은 기존 CNN을 MRI data에 적용하기 위한 연구 결과, 다른 3D subject-level CNN보다 최고 정확도가 낮은 것을 확인할 수 있었음
- ResNet, DenseNet의 일부만 사용함
- 기존 CNN을 그대로 사용한 연구
- data leakage 발견함
2.3.5 Conclusion
사용된 데이터셋, 선별된 표본, 전처리 방식이 달라 대부분의 연구 간 비교가 불가함
2.4 Other deep learning approached for AD classification
-
CNN 기반
- CNN을 특징 추출기로만 사용하고 분류기로는 다른 모델을 사용한 경우
- CNN을 특징 추출기와 분류기에 다 사용한 경우
-
CNN이 아닌 다른 모델로 MRI 분석
-
FC layer들로 구성된 Multilayer perceptron
-
확률적 신경망
-
비지도학습, AE, 지도학습 방식으로 특징을 추출 & SVM 분류
-
RNN
MLP
퍼셉트론을 여러 층 쌓아만든 인공신경망- 퍼셉트론 : 다수의 입력으로부터 하나의 결과를 내보내는 알고리즘
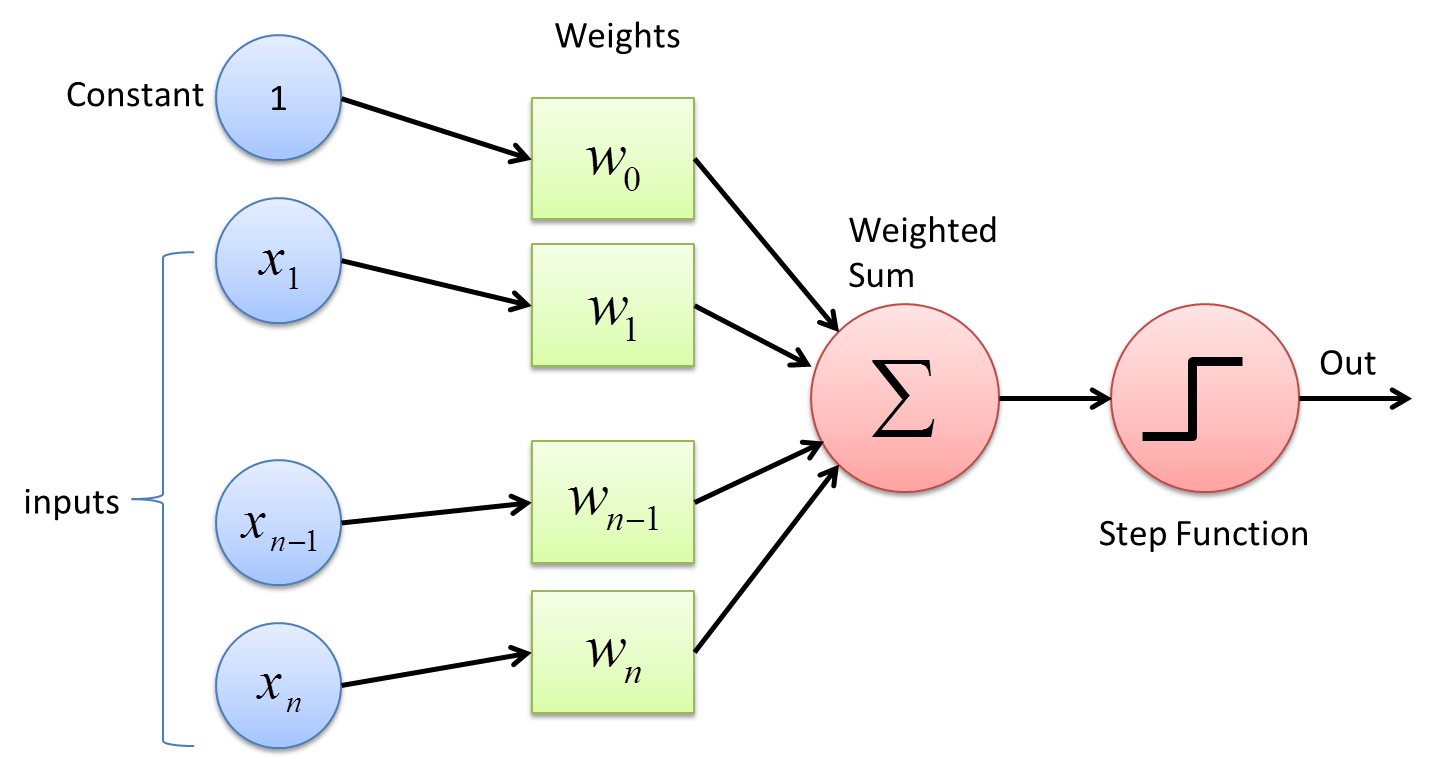
RNN (순환신경망)
한 층의 출력이 순환하며 다시 입력되는 재귀 결합을 가진 모델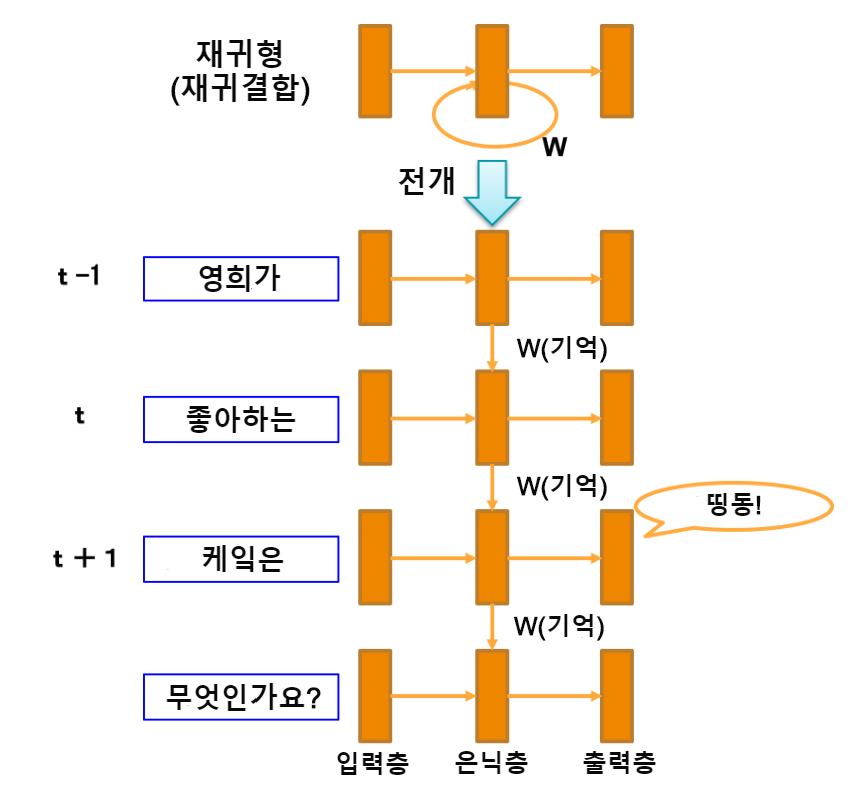 ⇒ CNN은 고정 길이의 이미지를 간직해서 처리하는 반면, RNN은 시간의 흐름과 가변 길이를 고려함
⇒ CNN은 고정 길이의 이미지를 간직해서 처리하는 반면, RNN은 시간의 흐름과 가변 길이를 고려함 - 퍼셉트론 : 다수의 입력으로부터 하나의 결과를 내보내는 알고리즘
3. Materials
ADNI, AIBL, OASIS 데이터셋을 사용함
- ADNI : AD/CN/MCI/sMCI/pMCI
- AIBL : AD/CN/MCI/sMCI/pMCI
- OASIS : AD/CN
4. Methods
4.1 Converting datasets to a standardized data structure
4.2 Preprocessing of T1w MRI
4.3 Classification models
처음에는 data leakage가 없는 연구에서 사용한 모델을 사용했으나,
모델 간 비교가 불가능하기 때문에 경험적 방식으로 각 approach별 최적 모델을 찾음
-
3D subject-level
4개의 conv blocks와 5개의 FC layer를 가진 overfit된 모델에서 출발하여 아래 규칙을 따라 최적 모델을 탐색함
1) 검증 데이터셋의 정확도가 유의미하게 감소할 때까지 FC layer의 수를 줄임
2) 하나의 conv block을 추가함conv block
= 1개의 conv layer + 1개의 batch normalization + 1개의 ReLU + 1개의 max pooling layer⇒ 결과
- 4 conv block + 2 FC layers (3D patch-level과 ROI based에도 최적)
- 4 conv block + 1 FC layers
- 7 conv block + 2 FC layers
- 7 conv block + 1 FC layers
-
2D slice-level
⇒ ResNet 끝부분에 FC-layer 1개 추가한 모델
-
4.3.1 3D subject-level CNN
: 5 conv blocks + 3 FC layers
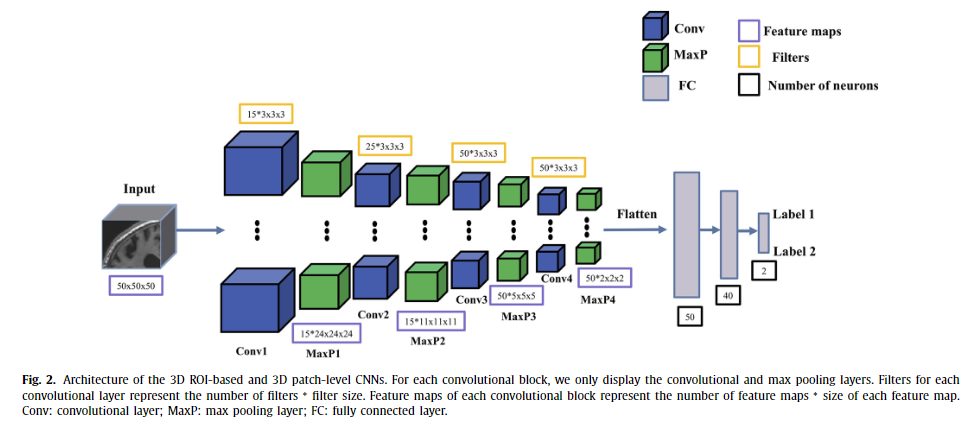
4.3.2 3D ROI-based and 3D patch-level CNN
: 4 conv blocks + 3 FC layers
: 50 X 50 X 50 크기의 patch 사용함
: 각 이미지마다 36개의 patch 추출함
- 3D patch-level
- single-CNN : 모든 추출된 patch들을 하나의 CNN으로 학습하는 방식
- multi-CNN : 각각의 patch마다 하나의 CNN을 사용 (⇒ 36개의 CNN 모델)
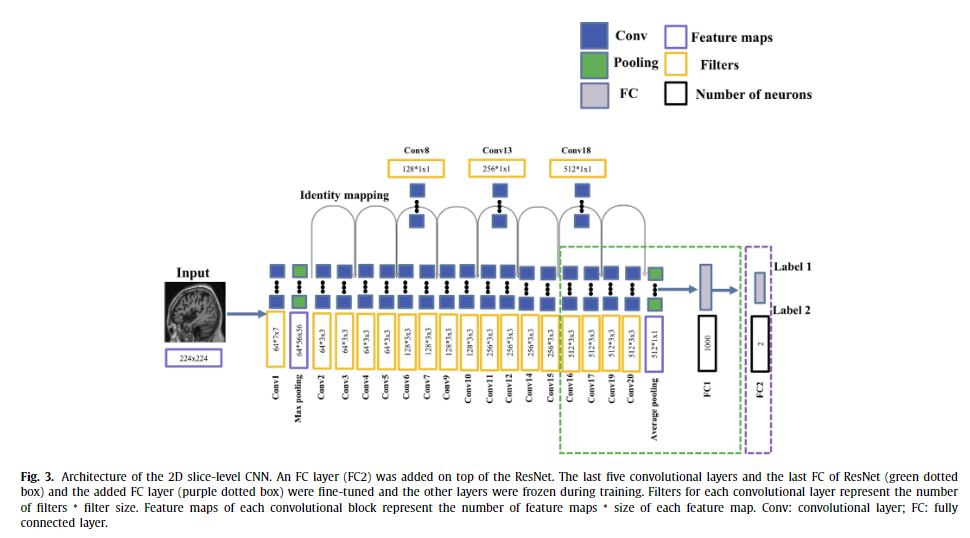
4.3.3 2D slice-level CNN
: ImageNet으로 사전학습된 ResNet + 1 FC layer (at the top of ResNet)
: ResNet의 마지막 5개 conv layers는 미세조정됨
: 사구체 관점으로 slicing → RGB 3개의 채널로 복제 → 첫번째랑 마지막 슬라이스는 제외함
⇒ 각 이미지당 원본 45개의 이미지로부터 129개의 RGB 슬라이스 추출
4.3.4 Majority voting system
3D ROI-based, 3D path-level, 2D slice-level 후
subject-level decision을 위해 soft voting system을 선택함
: 같은 환자로부터 모든 slice/patch/image를 softmax normalization한 결과를 예측한 확률
: j번째 slice/patch/image의 normalized된 정확도에 따라 부여된 가중치
4.3.5 Comparison to a linear SVM on voxel-based features
- 선형 SVM을 사용한 이유
4.4 Transfer learning
4.4.1 AE pre-training
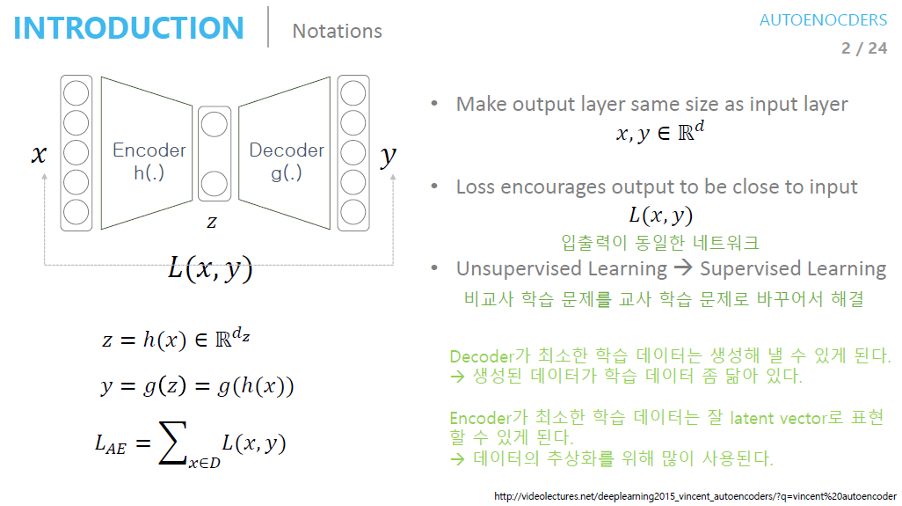
AutoEncoder
입력 데이터를 압축시키고 다시 확장해 결과 데이터가 입력 데이터와 동일할 확률을 최대로 만드는(MLE) 최적의 파라미터를 학습시킴
입력 데이터를 압축함으로써 latent vector를 얻어 입력 데이터의 대표적인 특성을 추출함
고차원 데이터를 잘 표현하는 latent vector를 모델이 자동으로 추출해줌→ 활용 분야 : 차원 축소, 특징 추출, DAE
- encoder : 여러 개의 (conv layer + batch norm layer + ReLU + max pooling layer)
- decoder : 여러 개의 (ReLU + batch norm layer + conv layer + max pooling layer)
data leakage를 피하기 위해 train/validation 데이터셋을 이용해 AE를 사전학습했고
AD/CN 분류를 위해 MCI,AD,CN에 대한 모든 가능한 훈련 데이터셋을 사용함
4.4.2 ImageNet pre-training
2D slice 모델을 위해 ImageNet으로 사전학습된 ResNet을 사용함
4.5 Classification task
- AD vs. CN
- sMCI vs. pMCI
⇒ 1에서 성능이 좋은 모델을 source로 2에 대해 미세조정함
4.6 Evaluation strategy
4.6.1 Validation procedure
test data와 train/validation data가 반드시 독립적으로 존재해야 함
- ADNI에 대해 학습된 모델을 ADNI/AIBL/OASIS로 test함
cross validation (교차검증)
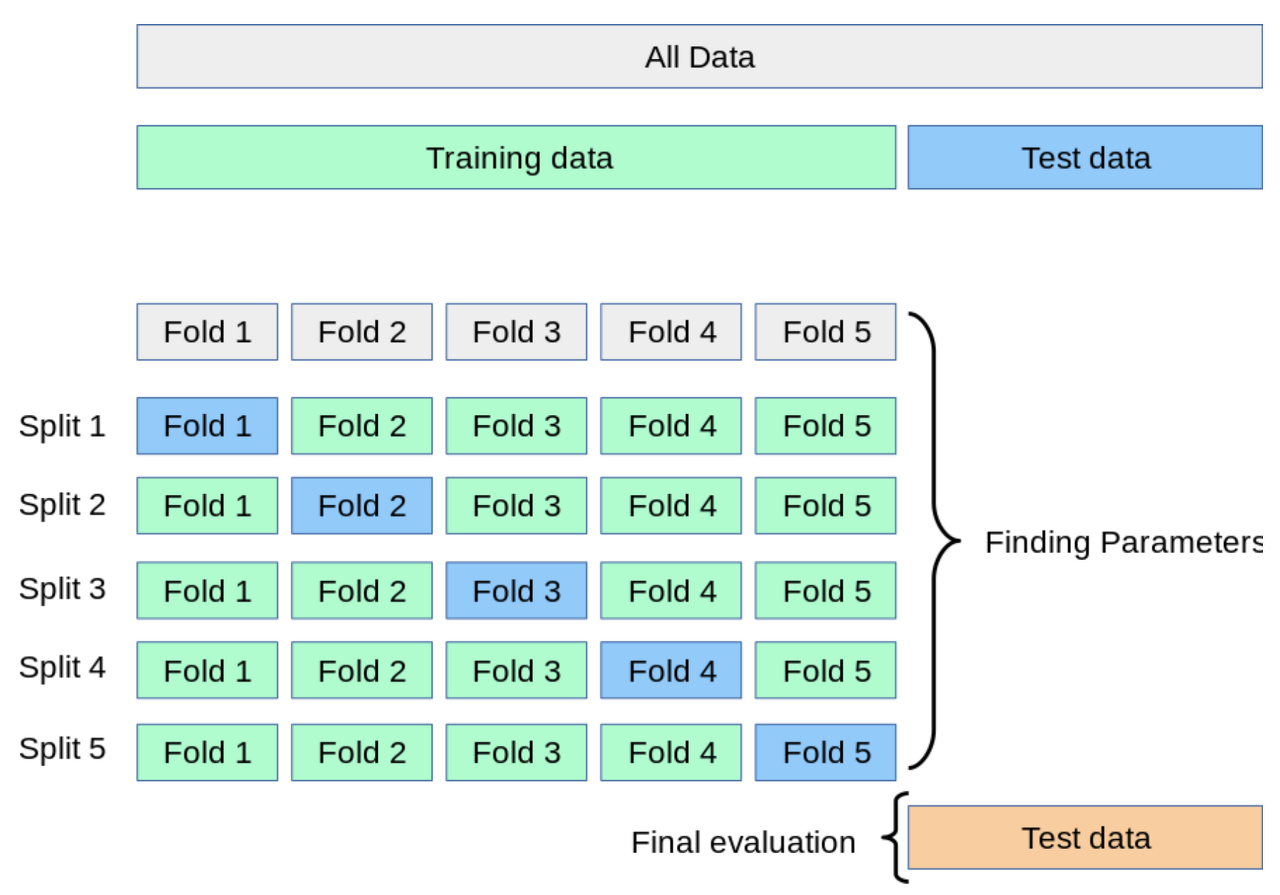 ⇒ Training data를 다시 train / validation으로 나눠 test data를 이용한 최종 평가 전 검증 단계를 수행함
⇒ Training data를 다시 train / validation으로 나눠 test data를 이용한 최종 평가 전 검증 단계를 수행함
4.6.2 Metrics
- BA
- ROC Curve
- Accuracy
- Specificity
5. Experiments and results
5.1 Results on traing/validation set
5.1.1 3D subject-level
- Influence of intensity rescaling
→ intensity를 rescaling하지 않으면 찍어서 맞히는 것(BA = 0.5)보다 CNN의 성능이 좋지 않음 - Influence of transfer learning(AE pretraining)
→ AE pretraining한 것이 그렇지 않은 것보다 성능이 약간 좋음 - Influence of the training data size
→ longitudinal data가 약간 성능이 더 좋음 - Influence of preprocessing
→ "Extensive"와 "Minimal" 둘 다 비슷한 성능을 보임 - Classification of sMCI vs. pMCI
→ longitudinal data와 baseline의 성능 같음
5.1.2 3D ROI-based
3D subject-level보다 전반적으로 약간 더 높은 성능을 보임
5.1.3 3D patch-level
ROI > Multi CNN patch-level > Single CNN patch-level
( Multi CNN은 최악의 patch를 제외하는 임계값 시스템이 존재함)
5.1.4 2D Slice-level
ROI, 3D subject-level, 3D multi CNN > 2D slice-level > 3D single CNN
5.1.5 Linear SVM
AD vs. CN : SVM > DL
sMCI vs. pMCI : SVM < DL
5.2 Results on the test sets
5.2.1 3D subject-level
AD vs. CN : ADNI와 AIBL은 비슷한 성능, OASIS는 성능 좋지 않음
sMCI vs. pMCI : ADNI는 성능 괜찮지만 AIBL은 아님
5.2.2 3D ROI-based
AD vs. CN : ADNI > AIBL > OASIS
sMCI vs. pMCI : ADNI는 3D subject-level와 성능 비슷하고 AIBL은 여전히 성능 안좋음
5.2.3 3D patch-level
AD vs. CN : ADNI와 AIBL은 비슷한 성능, OASIS는 성능 좋지 않음
sMCI vs. pMCI : ADNI는 성능 괜찮지만 AIBL은 아님
5.2.4 2D slice-level
AD vs. CN : ADNI와 AIBL은 비슷한 성능, OASIS는 성능 좋지 않음
+) data leakage model 포함
5.2.5 Linear SVM
AD vs. CN : ADNI와 AIBL은 비슷한 성능, OASIS는 성능 좋지 않음
sMCI vs. pMCI : ADNI는 성능 괜찮지만 AIBL은 아님
6. Discussion
- 이 연구의 결과
- 다른 연구 분석
- CNN과 T1w MRI로 AD 분류하는 오픈소스 프레임워크 제안
- 오픈소스 프레임워크 적용하여 성능의 주요 구성요소 파악
- 성능의 주요 구성 요소
- AE pre-training
- baseline data 대신 longitudinal data로 훈련시키기
- 이미지 전처리
- intensity rescale
- 이 연구의 한계
- 모델과 학습 파라미터를 선택할 때 수많은 옵션들이 존재함
- Cross-validation 과정이 딱 한 번만 진행됨
- overfitting 존재
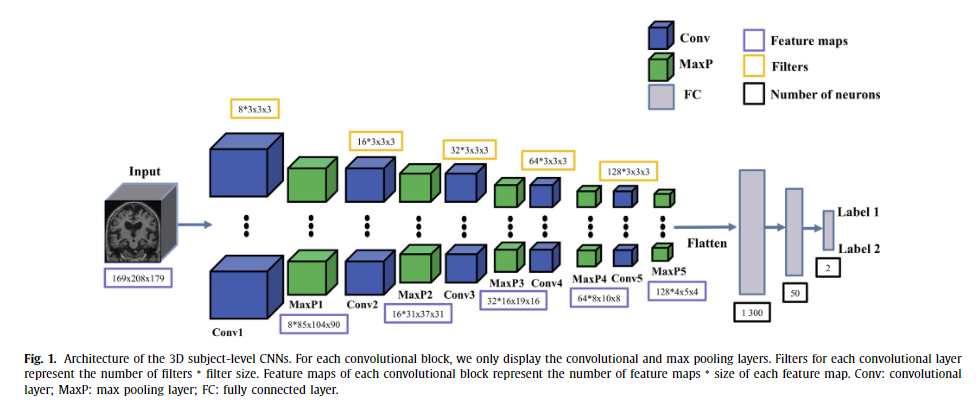
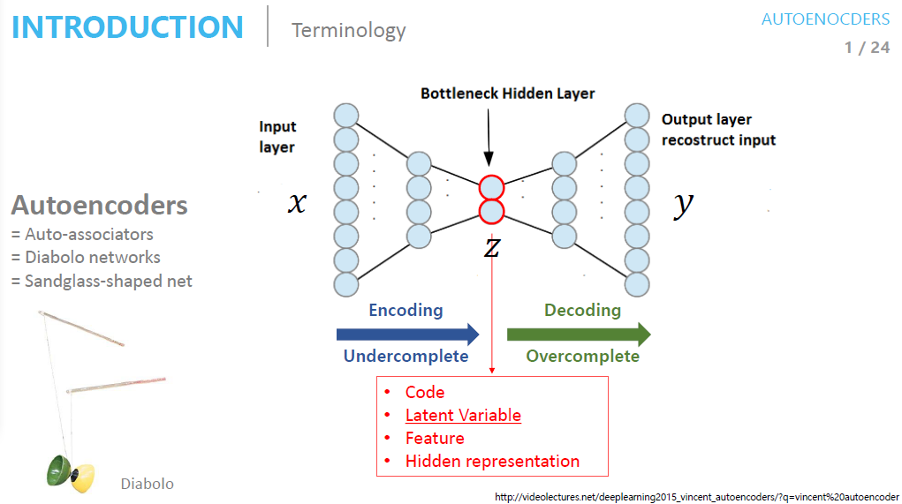
 : 입력과 출력이 같은 구조
: 입력과 출력이 같은 구조