EDA
데이터 불러오기
df_train = pd.read_csv('C:/workspace/py_dacon/data/penguin/train.csv')
df_train.info()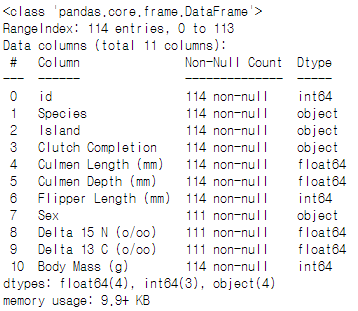
df_train.head()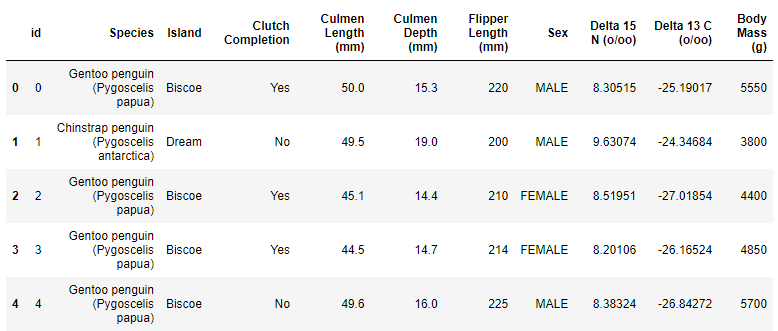
id는 필요 없어서 바로 drop
df_train.drop(['id'], axis=1, inplace=True)범주형, 연속형 변수 컬럼명 지정
discrete_names = ['Species', 'Island', 'Clutch Completion', 'Sex']
continuous_names = ['Culmen Length (mm)', 'Culmen Depth (mm)', 'Flipper Length (mm)', 'Delta 15 N (o/oo)', 'Delta 13 C (o/oo)']결측치 체크
연속형 변수는 각 컬럼의 평균 값으로 넣어주고, 범주형 변수는 어차피 one-hot 인코딩 할 예정이라 그냥 둠
df_train.isna().sum()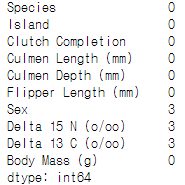
df_train['Delta 15 N (o/oo)'].fillna(value=df_train['Delta 15 N (o/oo)'].mean(), inplace=True)
df_train['Delta 13 C (o/oo)'].fillna(value=df_train['Delta 13 C (o/oo)'].mean(), inplace=True)아래의 각 그래프들의 소스는 dacon의 운영자분의 코드를 참조하였음
boxplot으로 이상치 확인
feature = continuous_names
plt.figure(figsize=(20,15))
plt.suptitle("Boxplots", fontsize=40)
for i in range(len(feature)):
plt.subplot(2,3,i+1)
plt.title(feature[i])
plt.boxplot(df_train[feature[i]])
plt.show()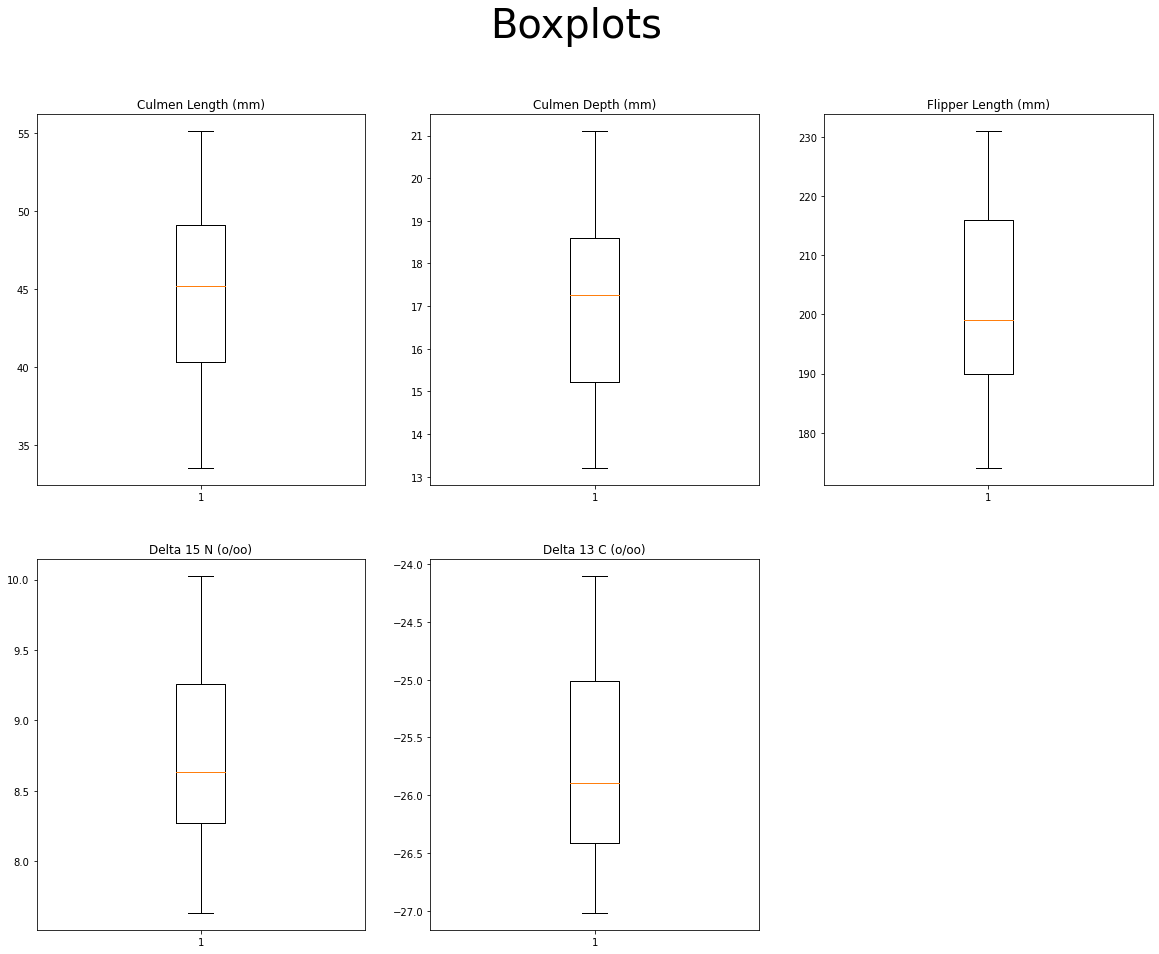
특이한 이상치는 보이지 않는 것으로 판단
산점도
target = "Body Mass (g)"
plt.figure(figsize=(20,15))
plt.suptitle("Scatter Plot", fontsize=40)
for i in range(len(continuous_names)):
plt.subplot(2,3,i+1)
plt.xlabel(continuous_names[i])
plt.ylabel(target)
corr_score = df_train[[continuous_names[i], target]].corr().iloc[0,1].round(2)
c = 'red' if corr_score > 0 else 'blue'
plt.scatter(df_train[continuous_names[i]], df_train[target], color=c, label=f"corr = {corr_score}")
plt.legend(fontsize=15)
plt.tight_layout(rect=[0, 0.03, 1, 0.95])
plt.show()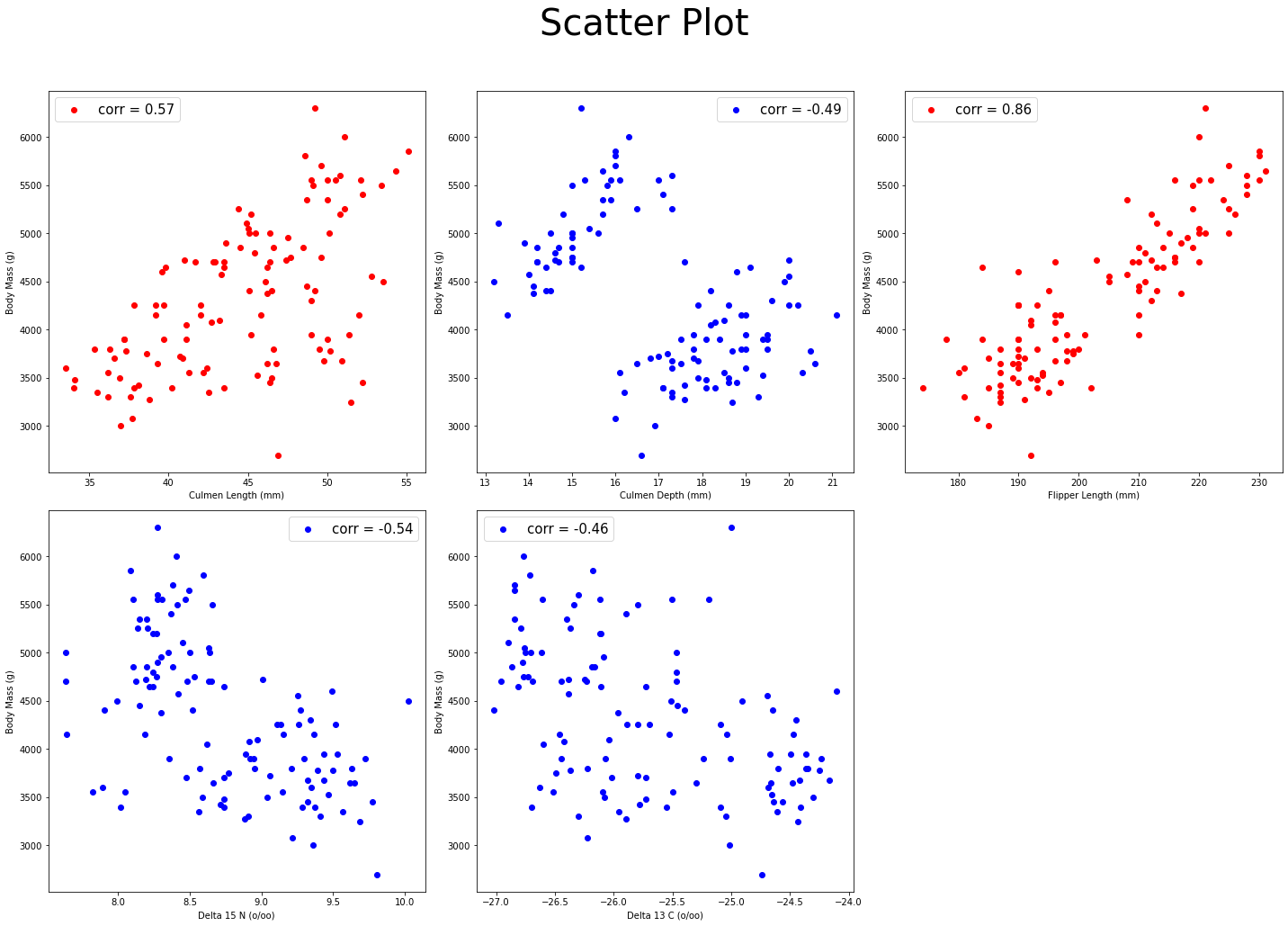
0 미만의 상관값을 가진 변수는 파란색으로 나타냈는데, 'Culmen Depth (mm)' 변수의 경우 양쪽으로 나뉘어져 있긴 하나 다른 변수와의 상관관계가 있을 것 같아 놔두고, 'Delta 15 N (o/oo)', 'Delta 13 C (o/oo)' 두 변수는 너무 퍼져있어 drop할 예정
bar plot
# 히스토그램 을 사용해서 데이터의 분포를 살펴봅니다.
feature = discrete_names
plt.figure(figsize=(20,15))
plt.suptitle("Bar Plot", fontsize=40)
for i in range(len(feature)):
plt.subplot(2,2,i+1)
plt.title(feature[i], fontsize=20)
temp = df_train[feature[i]].value_counts()
plt.bar(temp.keys(), temp.values, width=0.5, color='b', alpha=0.5)
plt.xticks(temp.keys(), fontsize=12)
plt.tight_layout(rect=[0, 0.03, 1, 0.95])
plt.show()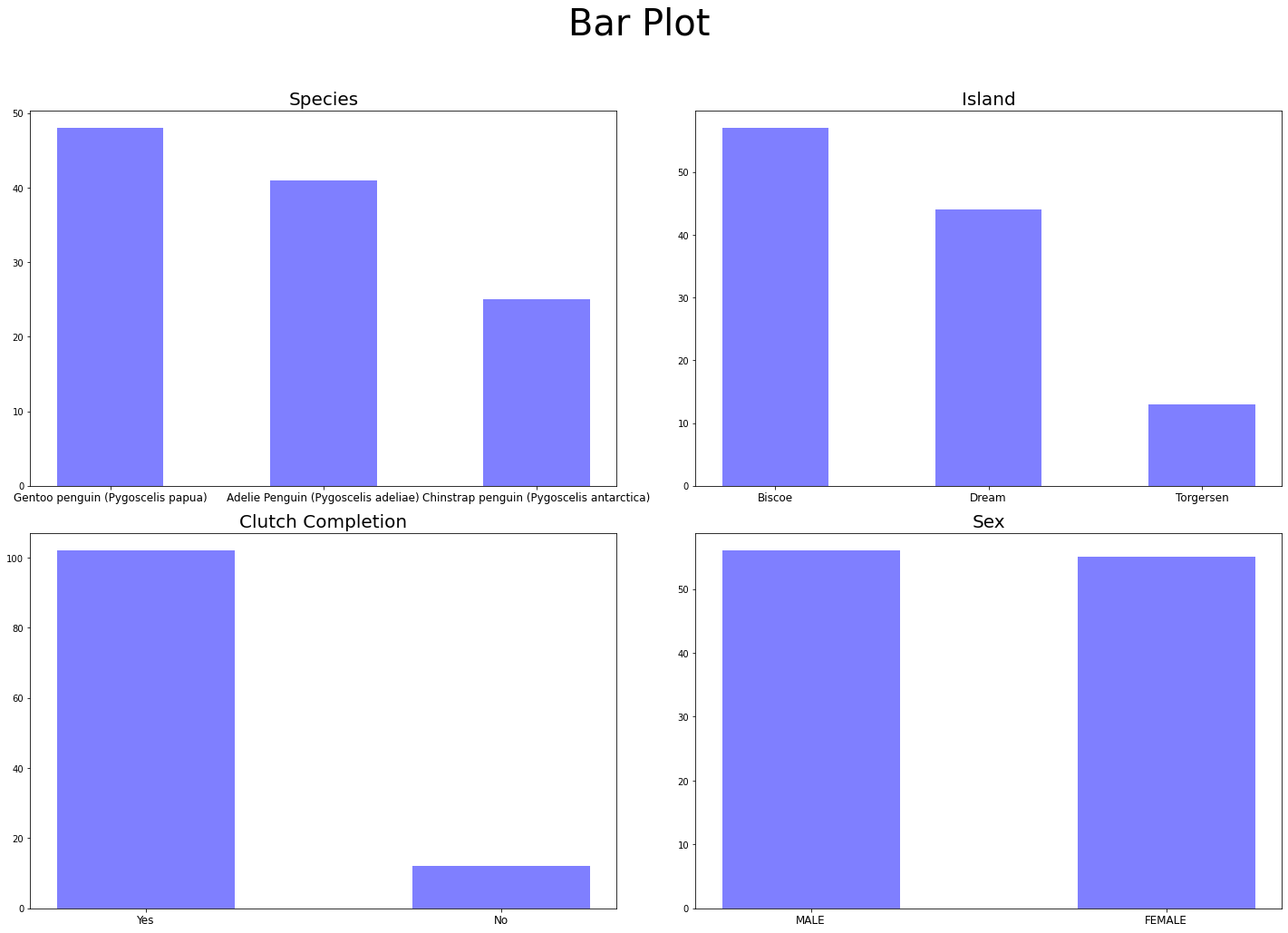
'Clutch Completion' 변수는 값의 분포가 너무 극단적이여서 drop할 예정
heatmap
from sklearn.preprocessing import LabelEncoder
corr_df = df_train.copy()
corr_df[corr_df.columns[corr_df.dtypes=='O']] = corr_df[corr_df.columns[corr_df.dtypes=='O']].astype(str).apply(LabelEncoder().fit_transform)
plt.figure(figsize=(15,10))
heat_table = corr_df.corr()
mask = np.zeros_like(heat_table)
mask[np.triu_indices_from(mask)] = True
heatmap_ax = sns.heatmap(heat_table, annot=True, mask = mask, cmap='coolwarm')
heatmap_ax.set_xticklabels(heatmap_ax.get_xticklabels(), fontsize=15, rotation=45)
heatmap_ax.set_yticklabels(heatmap_ax.get_yticklabels(), fontsize=15)
plt.title('correlation between features', fontsize=40)
plt.show()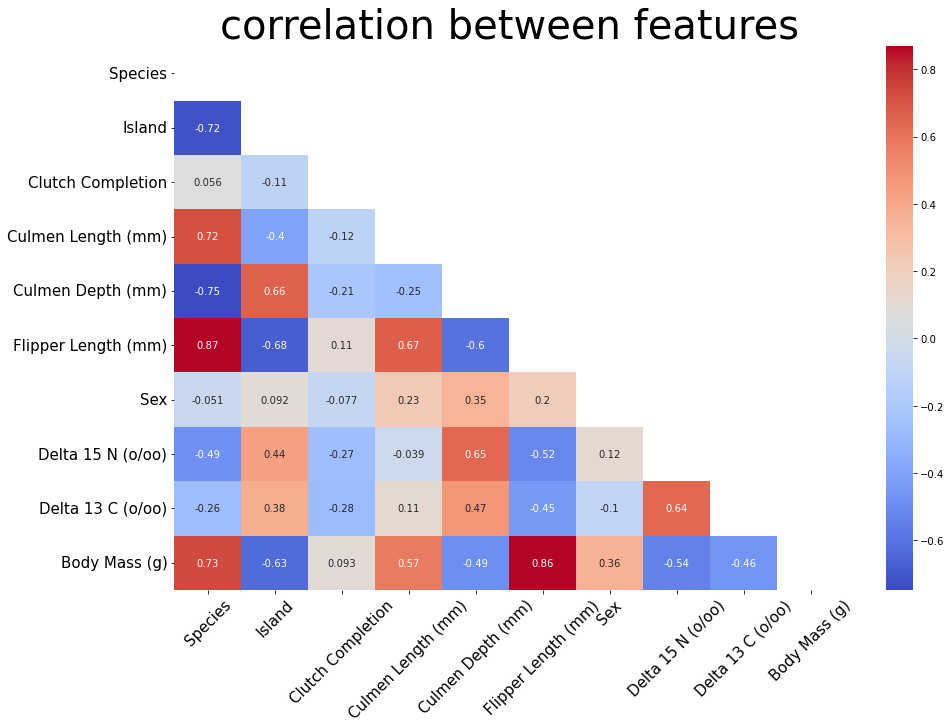
수치 값으로만 그릴 수 있어 범주형 변수를 라벨인코더를 통해 가공하고 확인해보면 제일 높은 상관관계를 보이는 변수는 'Flipper Length (mm)'임
EDA 정리
- 각 수치형 변수들의 이상치는 딱히 없어보임
- 범주형 변수는 라벨인코딩 보다는 one-hot으로 간단하게 정리하는 것이 편할 듯
- 'Delta 15 N (o/oo)', 'Delta 13 C (o/oo)'는 산점도 확인 시, 분포가 너무 퍼져있어 drop
- 'Culmen Depth (mm)'는 양쪽으로 나뉘어있긴 하나, x로 활용할 수 있는 변수가 너무 적기도 하여 일단 유지
- 'Clutch Completion'는 값의 분포가 너무 극단적이어서 drop