1. 프로젝트 개요
Iris의 품종 분류는 총 세 가지이다.
- Versicolor 2. Virginica 3. Setosa
이 중, 꽃잎(petal), 꽃받침(sepal)의 길이/너비 정보를 이용해서 이 3종의 품종을 구분할 수 있을까?
2. 데이터 관찰
from sklearn.datasets import load_iris
iris = load_iris()
iris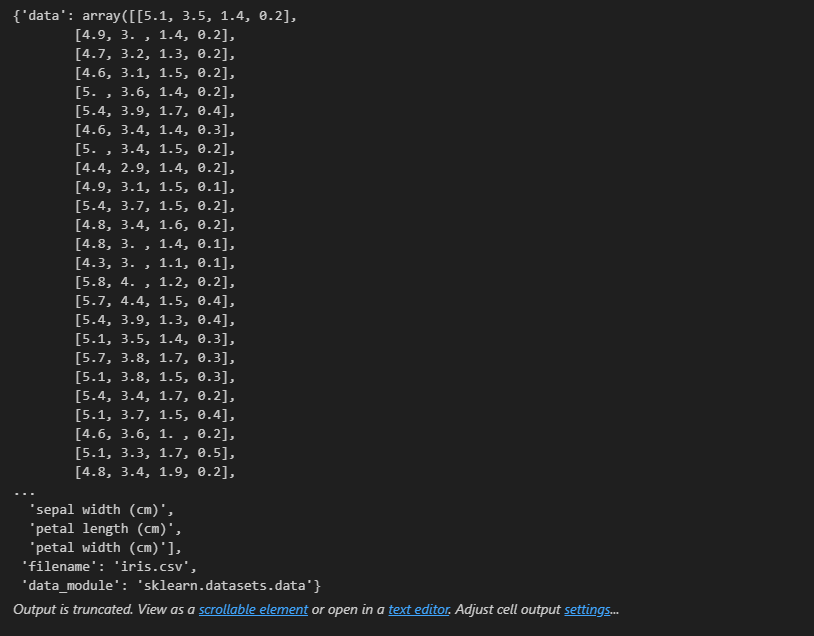
딕셔너리 형태이다. key 값들을 확인해보자 !
iris.keys()
이 데이터의 설명이 자세히 입력되어있는 'DESCR'을 확인해보자 !
print(iris['DESCR'])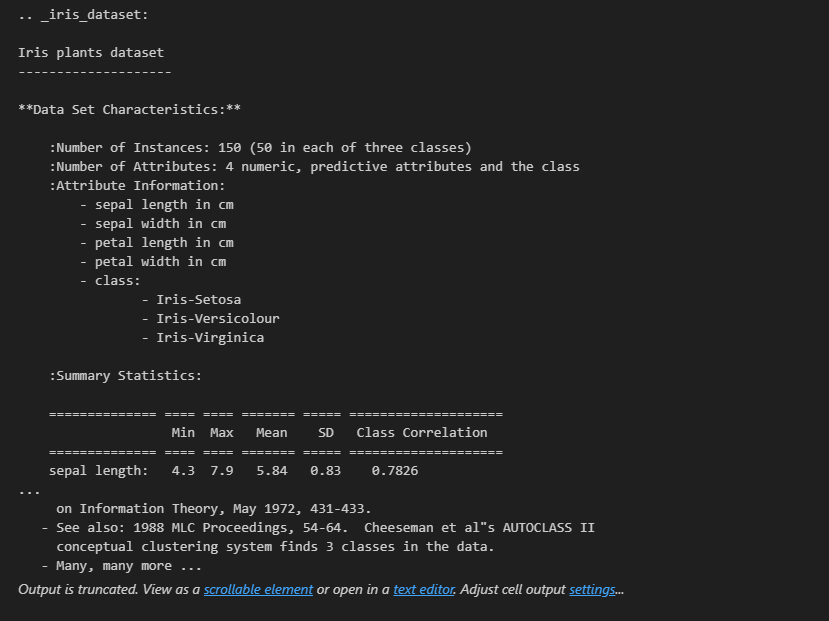
데이터를 dataframe 으로 설정
import pandas as pd
iris_pd = pd.DataFrame(data=iris.data, columns=iris.feature_names)
iris_pd.head()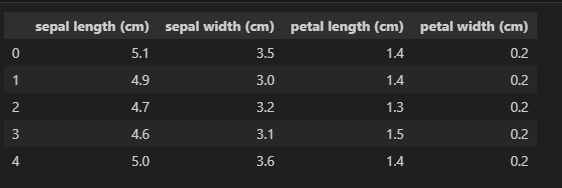
품종 정보도 column에 포함하자 !
iris_pd['species'] = iris.target
iris_pd.head()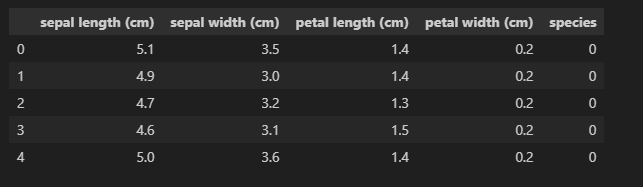
3. 데이터 시각화
3-1. sepal length 와 species 와의 관계
import matplotlib.pyplot as plt
import seaborn as sns
plt.figure(figsize=(12,6))
sns.boxplot(x='sepal length (cm)', y='species' , data=iris_pd, orient='h');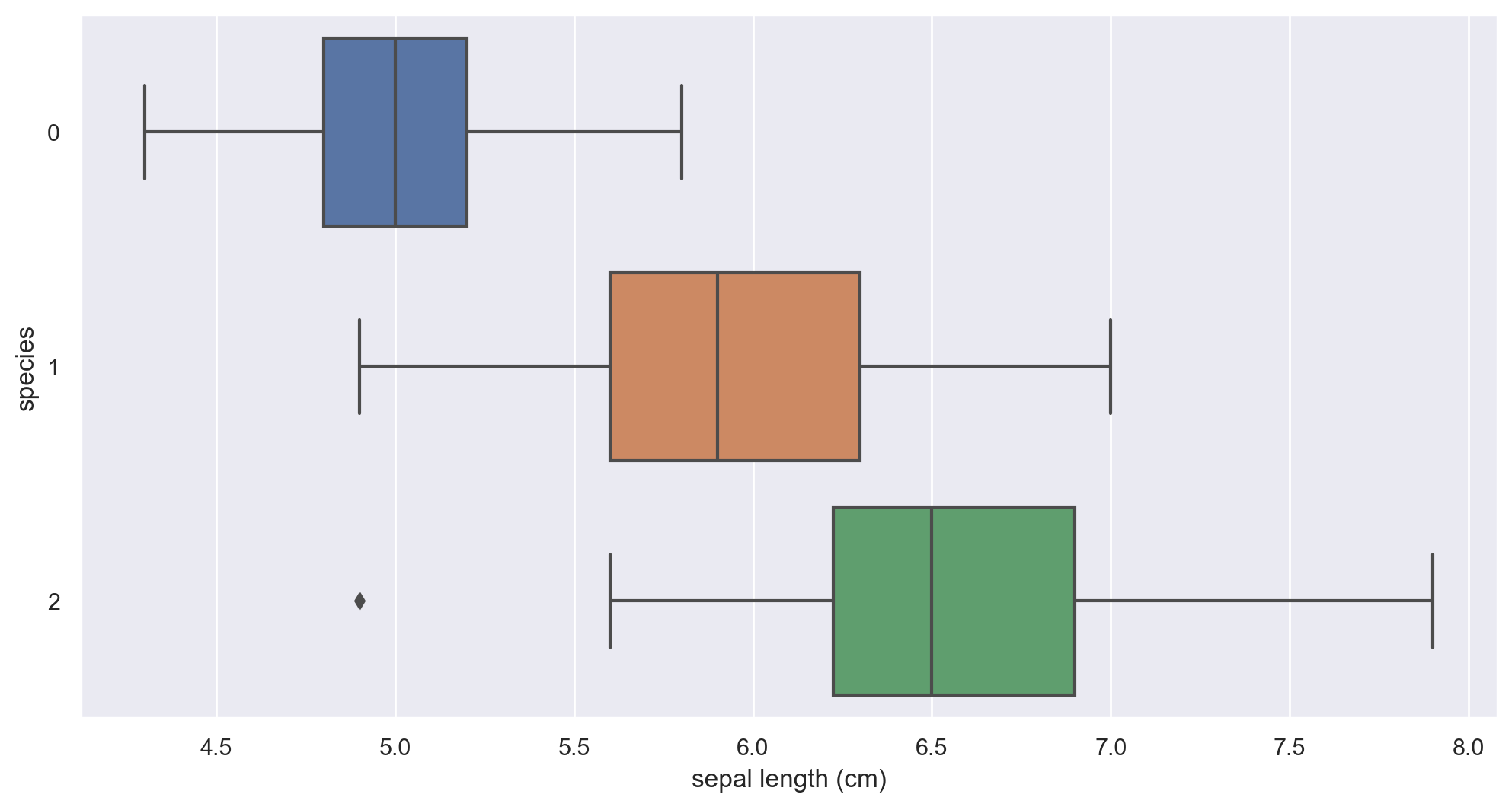
1번과 2번이 살짝 겹쳐지는 부분이 존재
3-2. sepal width 와 species 와의 관계
plt.figure(figsize=(12,6))
sns.boxplot(x='sepal width (cm)', y='species' , data=iris_pd, orient='h');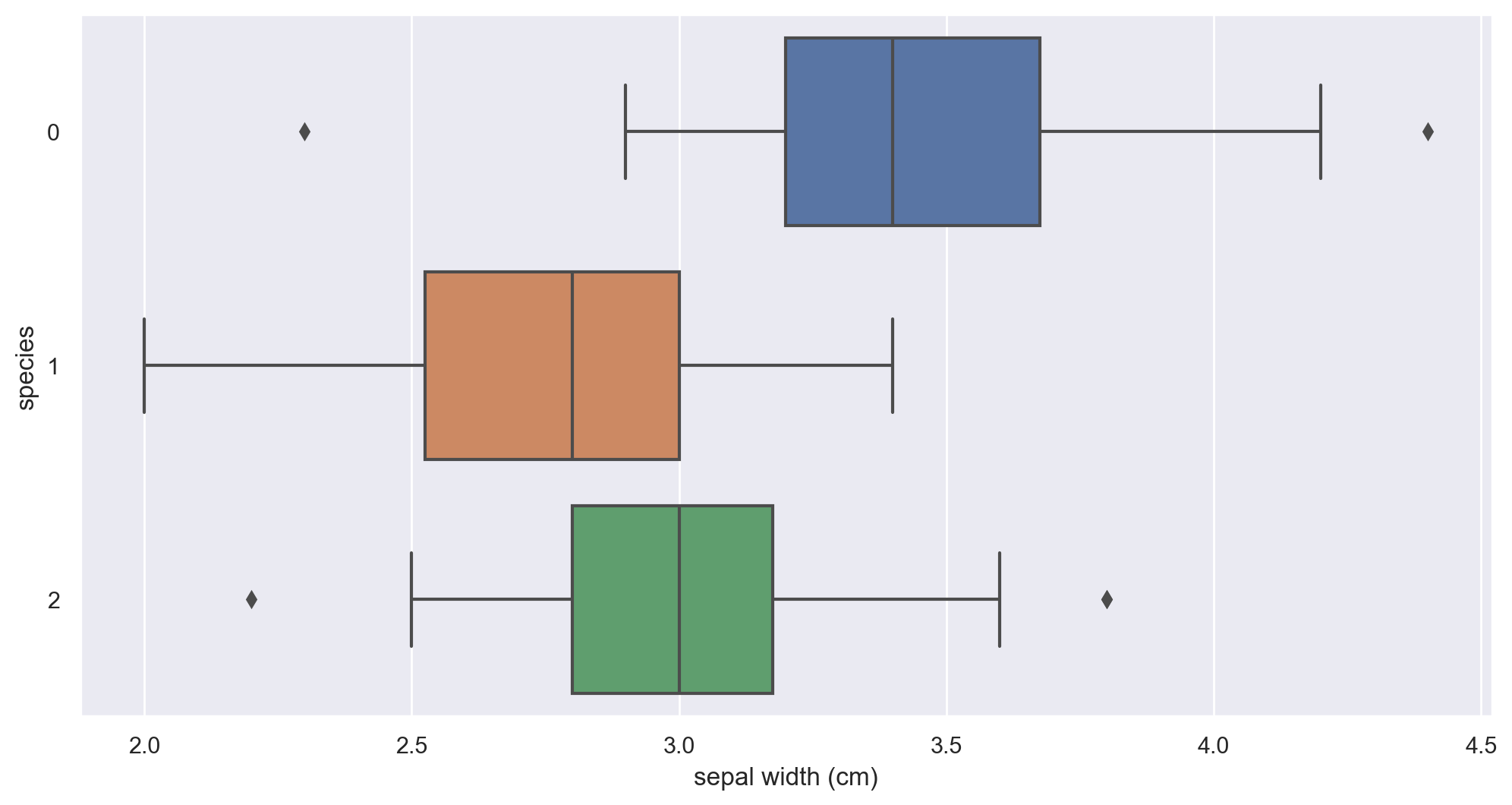
겹쳐지는 부분이 더욱 많이 존재한다.
3-3. petal length 와 species 와의 관계
plt.figure(figsize=(12,6))
sns.boxplot(x='petal length (cm)', y='species' , data=iris_pd, orient='h');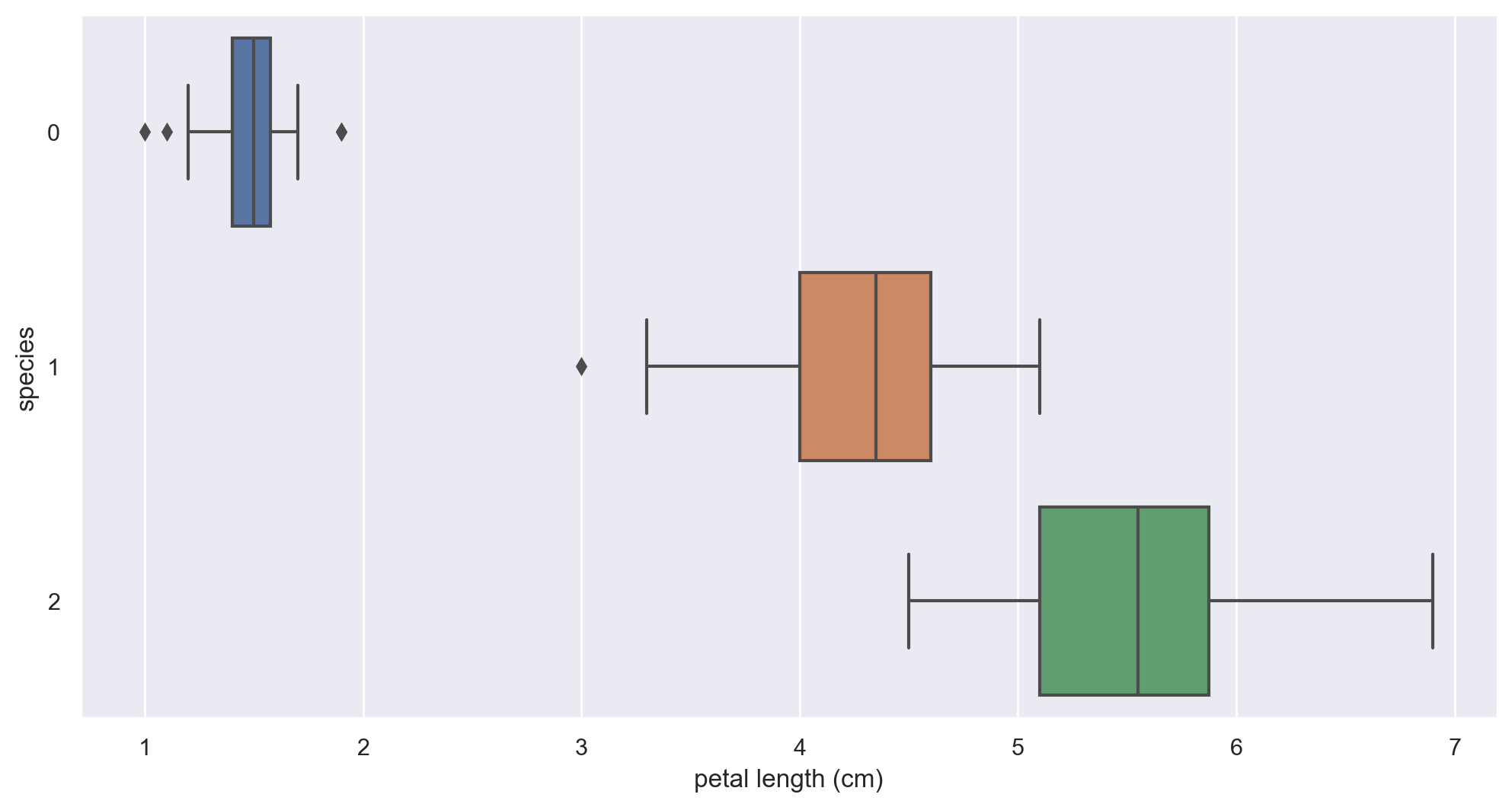
0번이 확실히 분리된다.
3-4. petal width 와 species 와의 관계
plt.figure(figsize=(12,6))
sns.boxplot(x='petal width (cm)', y='species' , data=iris_pd, orient='h');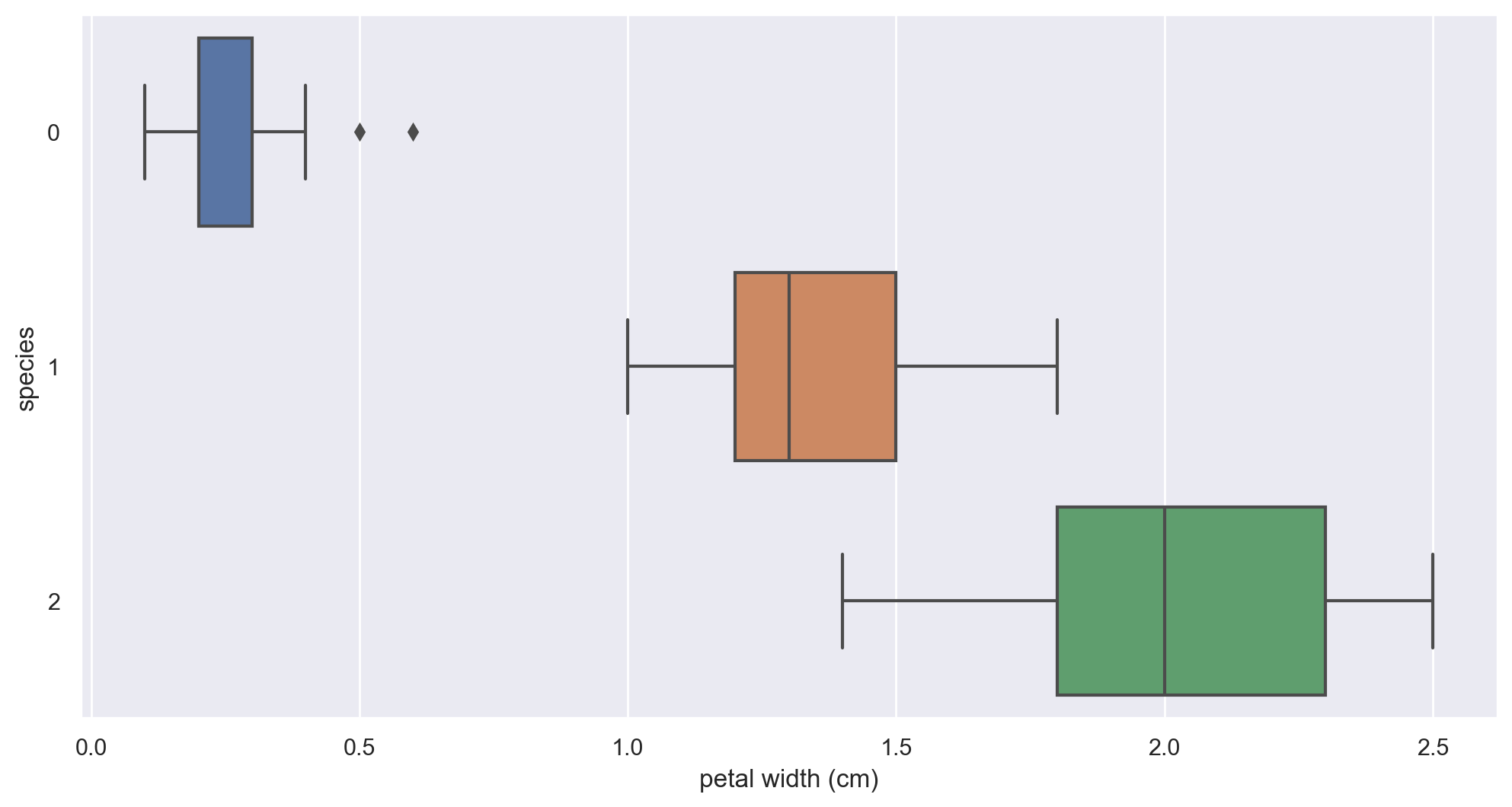
마찬가지이다. 이제 pairplot으로 확인해보자 !
sns.pairplot(iris_pd, hue='species');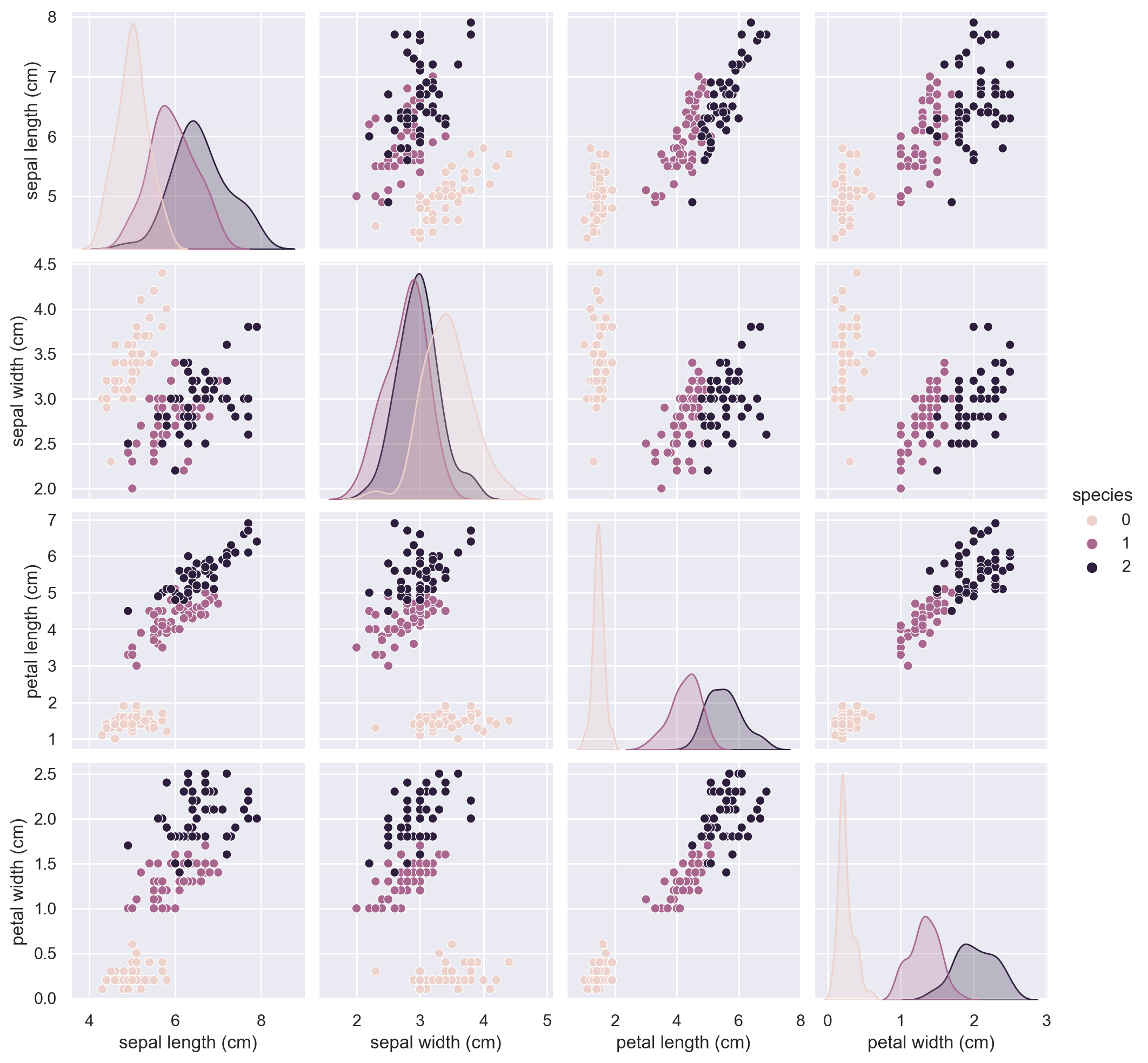
petal length 와 petal width 로 species를 잘 분류할 수 있을 것 같다
0번은 확실히 분류할 수 있지만, 1번과 2번을 분류하는 기준을 고민해볼 필요가 있다.
4. 데이터 모델링
의사결정나무를 이용하여 모델링을 시행해보자 !
from sklearn.tree import DecisionTreeClassifier
iris_tree = DecisionTreeClassifier()
iris_tree.fit(iris.data[:,2:], iris.target)from sklearn.metrics import accuracy_score
y_pred_tr = iris_tree.predict(iris.data[:,2:])
y_pred_tr
accuracy_score(iris.target, y_pred_tr)
99퍼센트의 성능을 보이므로 좋은 것 같아 보이지만, 사실 과적합 문제를 고려해야 한다 !
from sklearn.tree import plot_tree
plt.figure(figsize=(12,8))
plot_tree(iris_tree)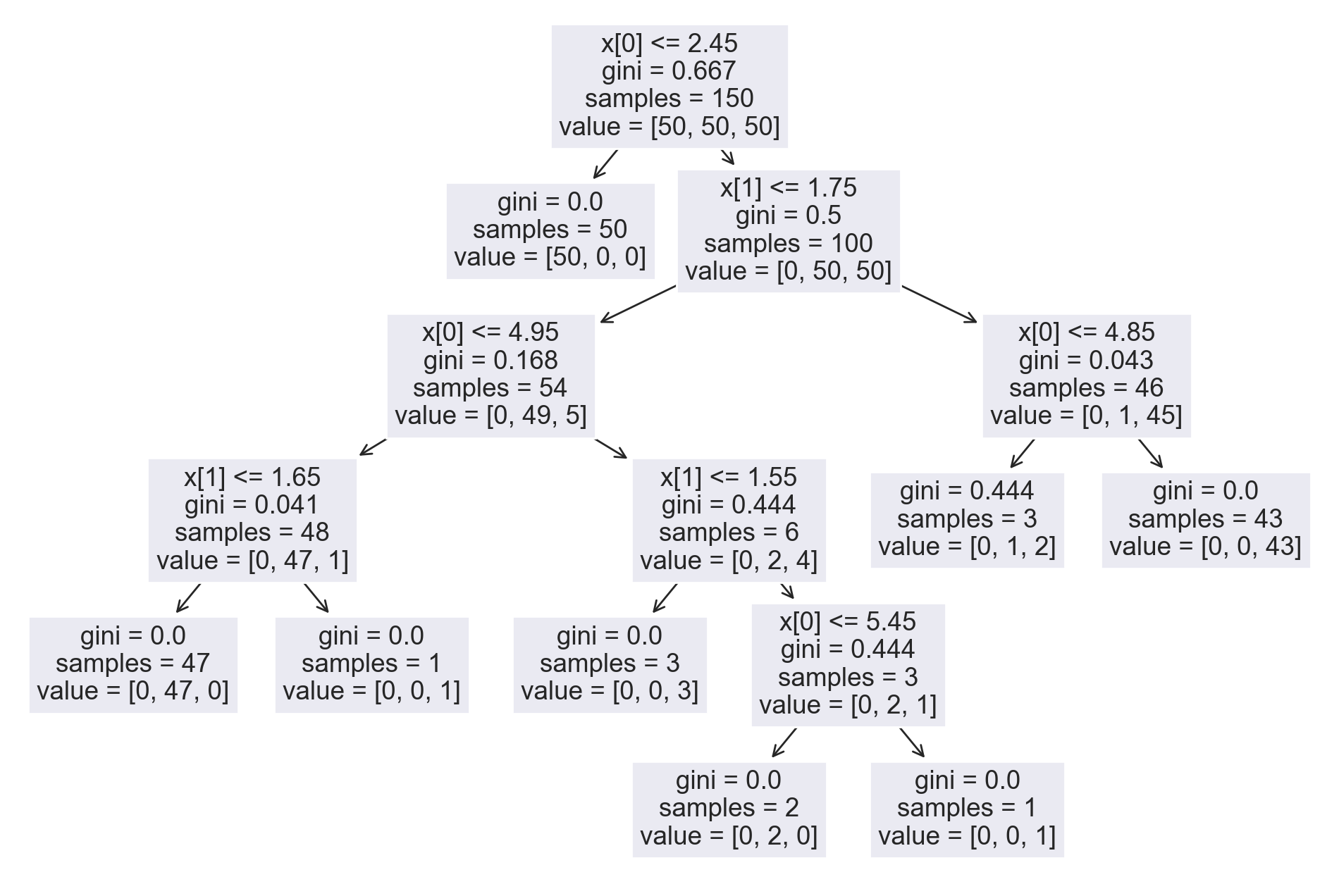
from mlxtend.plotting import plot_decision_regions
plt.figure(figsize=(14,8))
plot_decision_regions(X=iris.data[:, 2:], y=iris.target, clf=iris_tree, legend=2)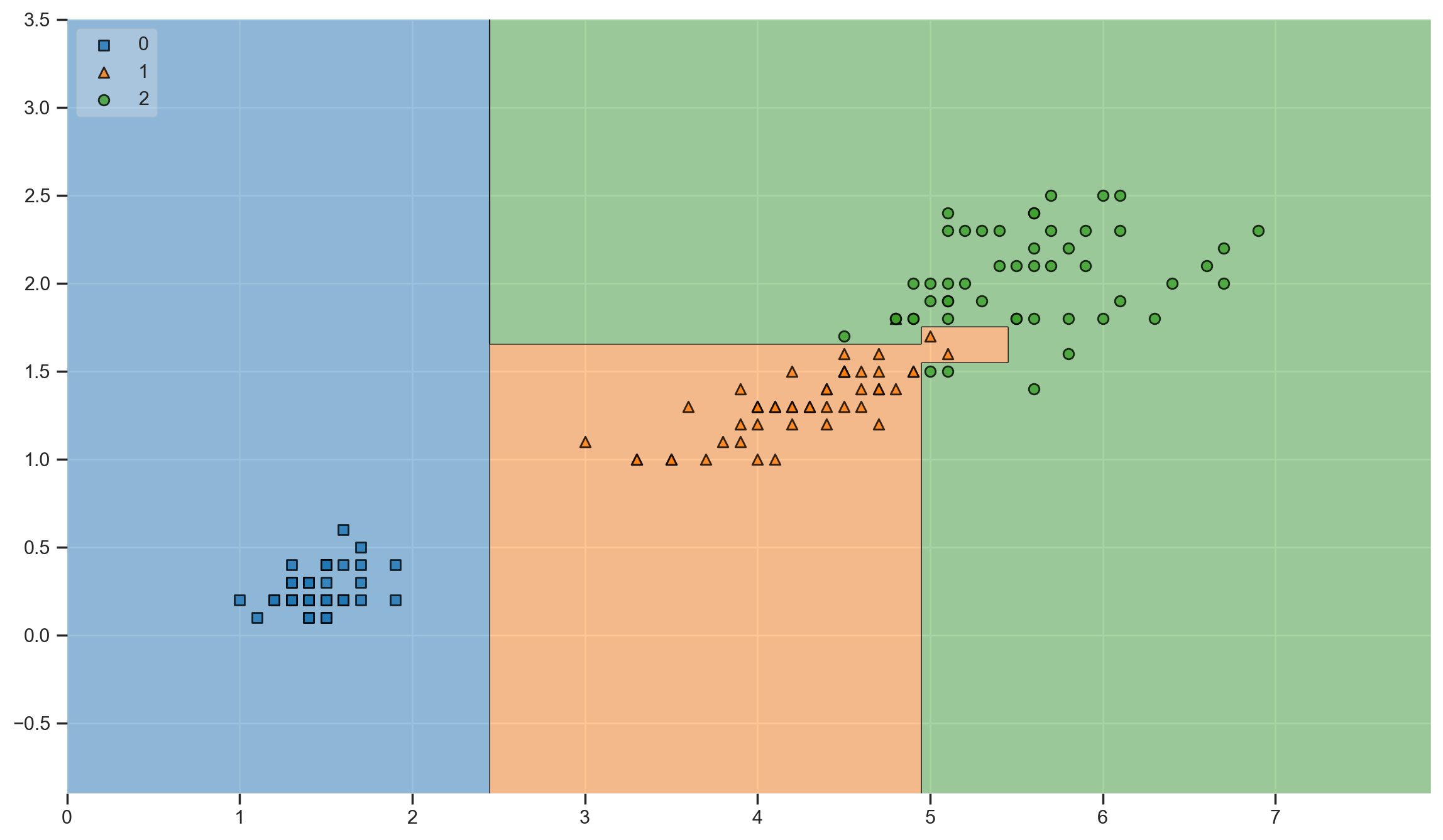
결정 경계를 확인해보니 매우 세밀하게 형성되어 있는 것을 확인할 수 있다.
-
150개의 data가 이 세상 모든 iris를 표현할 수 있을까 ?
-
이 결과로 과연 일반화할 수 있을 것일까 ?
결국, 복잡한 경계면은 모델의 성능을 나쁘게 만들고 내가 가진 데이터에만 너무 fit해지게 되면, 과적합이 발생하게 된다.
4-1. 데이터 분리
따라서, 전체 데이터를 분리해야 한다.
- 훈련 데이터 2. 테스트 데이터
확보한 데이터 중에서 모델 학습에 사용하지 않고 빼둔 데이터를 가지고 모델을 테스트한다.
iris = load_iris()
from sklearn.model_selection import train_test_split
features = iris.data[:, 2:]
labels = iris.target
X_train, X_test, y_train, y_test = train_test_split(features, labels, test_size=0.2, random_state=13)X_train.shape, X_test.shape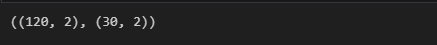
np.unique(y_test, return_counts=True)
각 클래스별로 동일 비율이 아니다. 따라서 stratify 옵션 이용 !
from sklearn.model_selection import train_test_split
features = iris.data[:, 2:]
labels = iris.target
X_train, X_test, y_train, y_test = train_test_split(features, labels, test_size=0.2, random_state=13, stratify=labels)np.unique(y_test, return_counts=True)
4-2. 데이터 모델링
다시 train 데이터만 대상으로 결정나무 모델을 만들어 보자 !
from sklearn.tree import DecisionTreeClassifier
iris_tree = DecisionTreeClassifier(max_depth=2, random_state=13)
iris_tree.fit(X_train, y_train)from sklearn.tree import plot_tree
plt.figure(figsize=(12,8))
plot_tree(iris_tree)
4-3. Accuracy 및 결정 경계 확인
train 데이터에 대한 accuracy 확인
from sklearn.metrics import accuracy_score
y_pred_tr = iris_tree.predict(iris.data[:,2:])
accuracy_score(iris.target, y_pred_tr)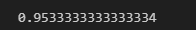
train 데이터에 대한 결졍 경계 확인
from mlxtend.plotting import plot_decision_regions
plt.figure(figsize=(14,8))
plot_decision_regions(X=X_train, y=y_train, clf=iris_tree, legend=2)
plt.show()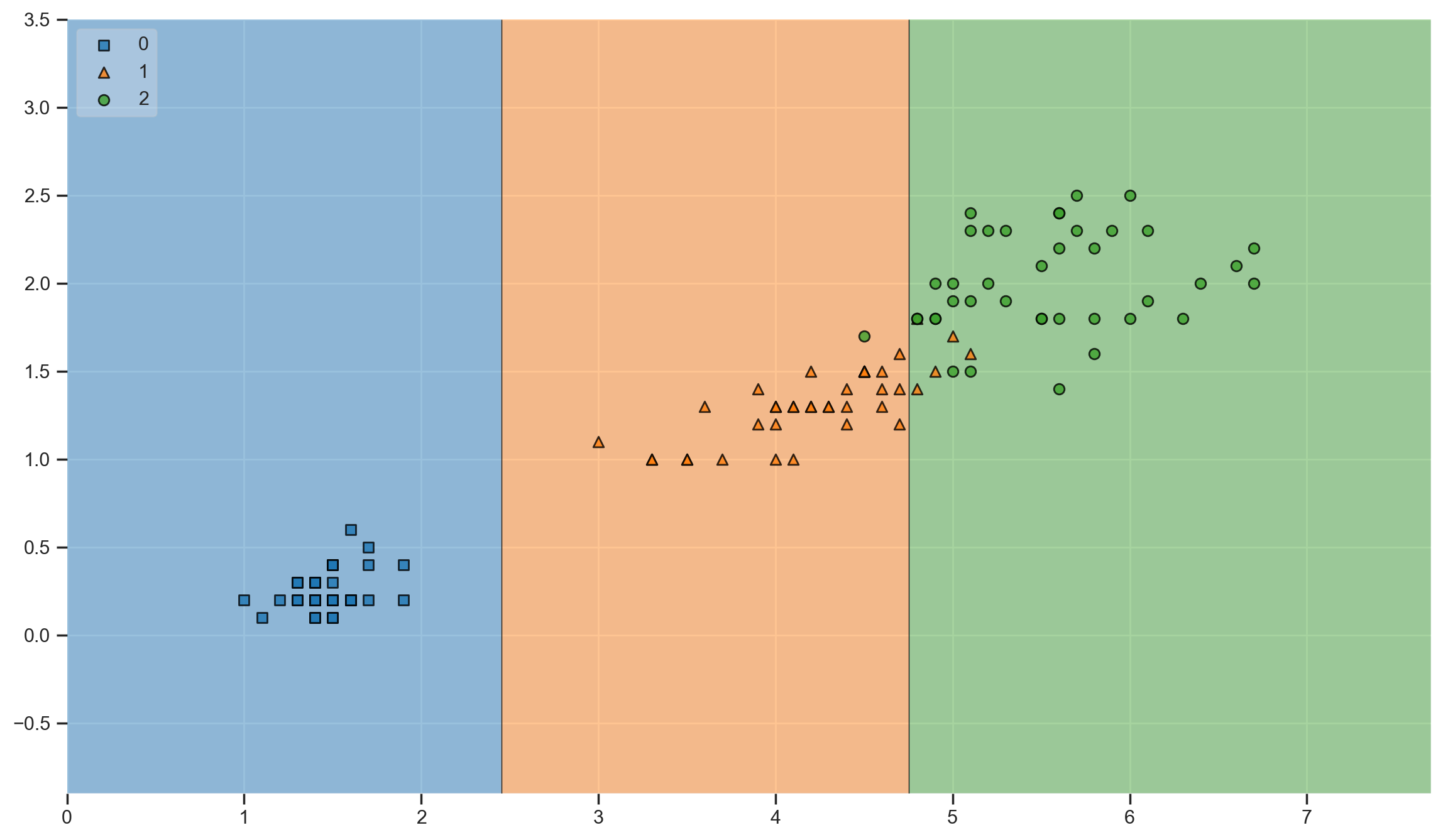
test 데이터에 대한 accuracy 확인
y_pred_test = iris_tree.predict(X_test)
accuracy_score(y_test, y_pred_test)
train 데이터에서는 95프로 정도 였는데 test 데이터에서는 그 근처가 나왔으니
적어도 이 데이터에서는 과적합이 발생하지 않았다.
test 데이터에 대한 결정 경계 확인
from mlxtend.plotting import plot_decision_regions
plt.figure(figsize=(14,8))
plot_decision_regions(X=X_test, y=y_test, clf=iris_tree, legend=2)
plt.show()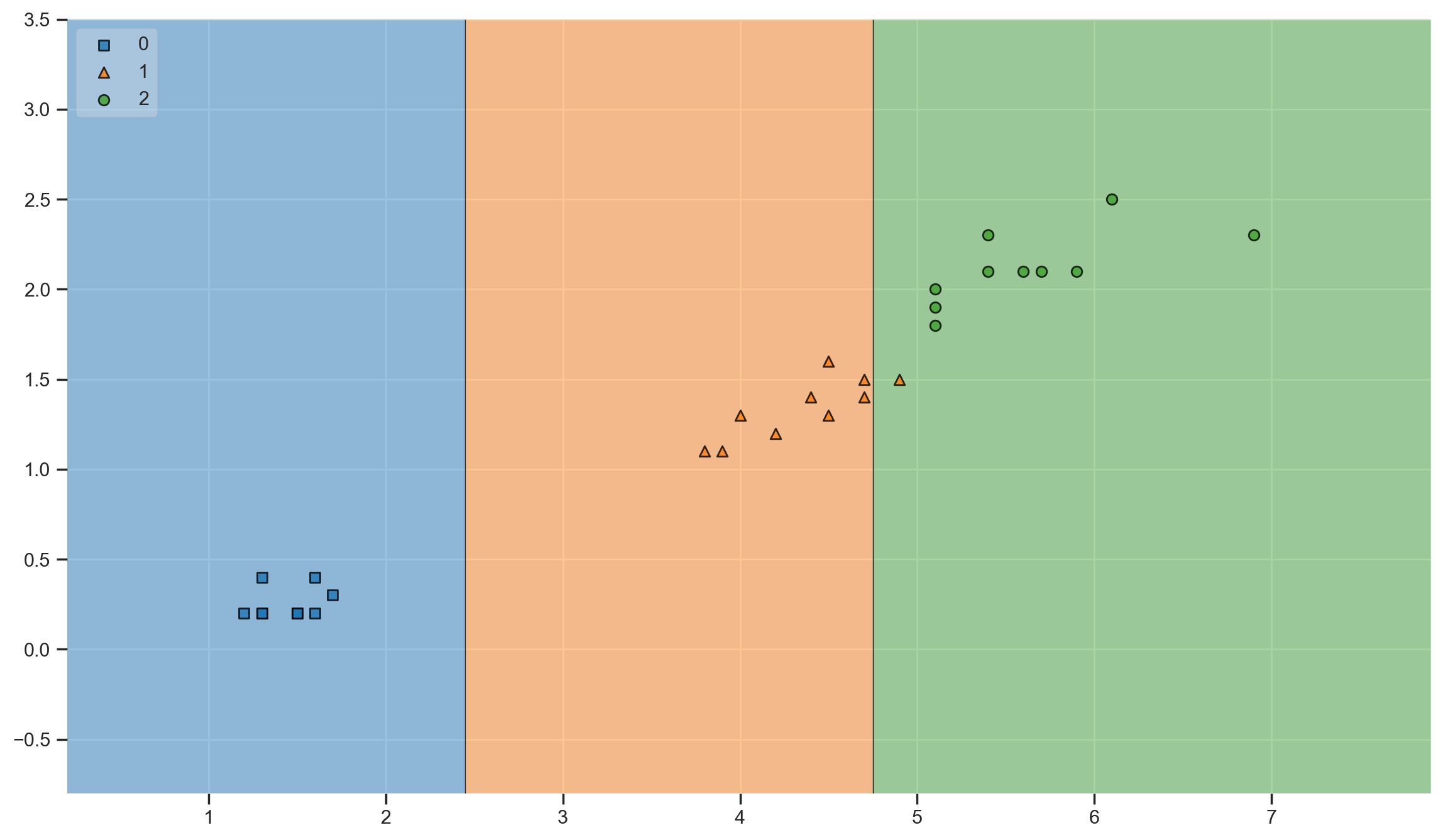
경계면이 훨씬 단순해졌다 !
# feature 를 네 개로 설정해보자
features = iris.data
labels = iris.target
X_train, X_test, y_train, y_test = train_test_split(features, labels, test_size=0.2, random_state=13, stratify=labels)
iris_tree = DecisionTreeClassifier(max_depth=2, random_state=13)
iris_tree.fit(X_train, y_train)plt.figure(figsize=(12,8))
plot_tree(iris_tree)
모델 사용 방법
# 새로운 데이터
test_data = np.array([[4.3, 2., 1.2, 1.]])
iris_tree.predict(test_data)
iris_tree.predict_proba(test_data)
iris.target_names[iris_tree.predict(test_data)]
4-4. 그 외 tip
- train 와 test 데이터의 결정 경계를 한번에 확인하고 싶을 때
scatter_highlight_kwargs = {'s' : 150, 'label' : 'Test data', 'alpha' : 0.9}
scatter_kwargs = {'s' : 120, 'edgecolor' : None , 'alpha' : 0.9}plt.figure(figsize=(12,8))
plot_decision_regions(X=features, y=labels,
X_highlight = X_test, clf=iris_tree, legend=2,
scatter_highlight_kwargs=scatter_highlight_kwargs,
scatter_kwargs=scatter_kwargs,
contourf_kwargs={'alpha':0.2});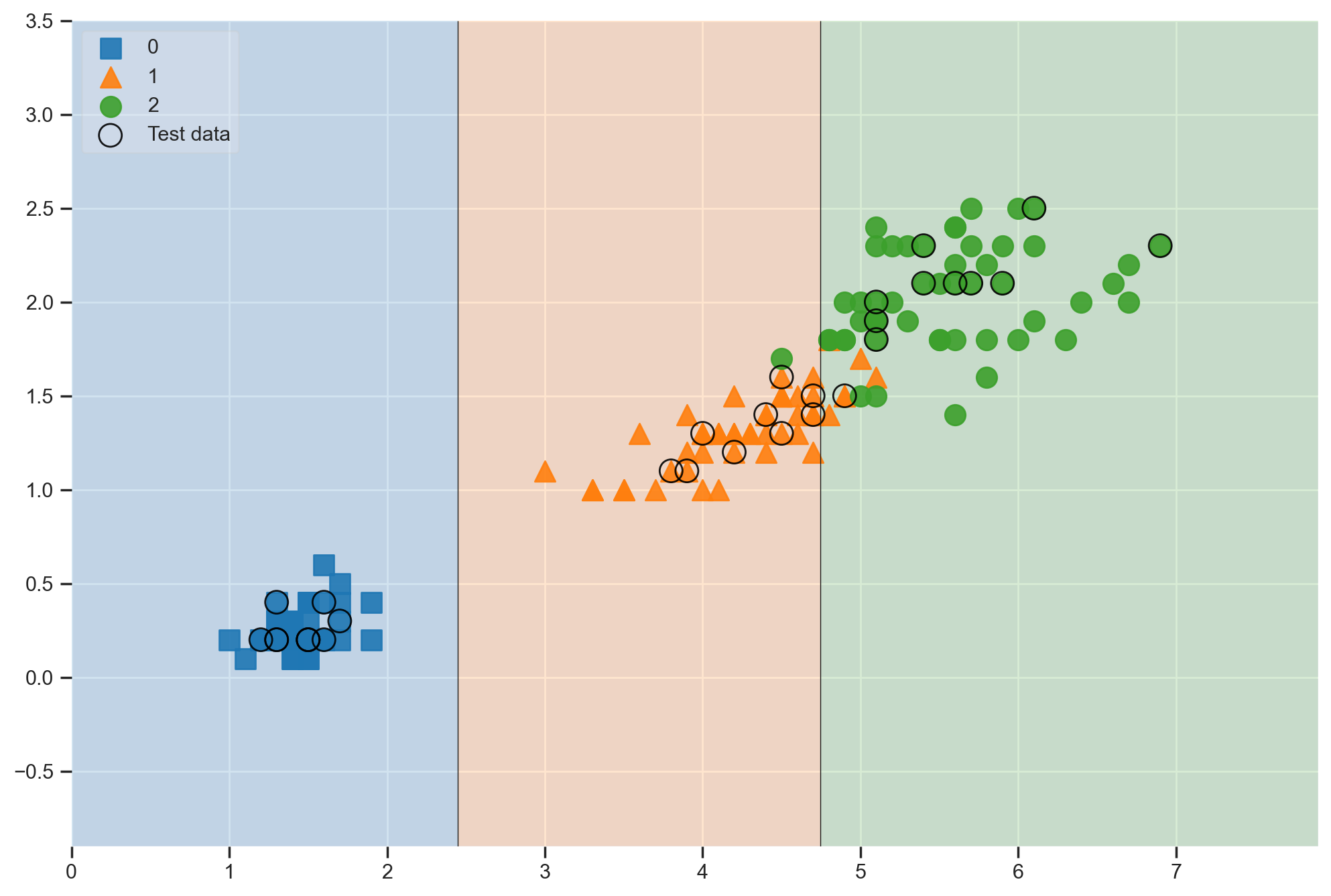
- 모델을 결정하는데 중요 feature를 알고 싶을 때
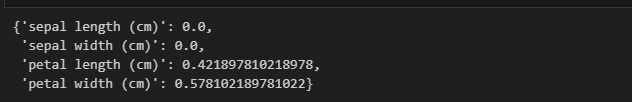
iris_tree.feature_importances_
# 좀 더 보기 편함
iris_clf_model = dict(zip(iris.feature_names, iris_tree.feature_importances_))
iris_clf_model