[2023 MICCAI] Segment Membranes and Nuclei from Histopathological Images via Nuclei Point-Level Supervision
Paper Review
연구그룹: 중국 북서대, 중국 청두 전기공학, 상하이 후단대, 상하이 Second 의대
요약: IHC 염색 이미지에서 세포핵에 대한 point supervision로 세포막 (membranes)를 분할하는 방법을 제안하는 연구로 세포막과 세포핵에 대한 분할 결과를 생성하는 Seg-Net과 분할결과를 의미적 포인트로 변환하는 Tran-Net의 2개의 신경망으로 이루어진 딥러닝 프레임워크를 제안함.
키워드: Membrane segmentation · Point-based supervision · Immunohistochemical image
1. Introduction
IHC 세포막 염색 이미지들의 정확한 계측은 질병 평가에 중요하다. 임상 진단에서 염색된 세포막 영역의 비율을 수동으로 추정하거나 염색된 세포막의 completeness를 평가하는 방식으로 질병의 등급을 매기는데, 이런 방식은 tedious, time-consuming, error-prone.
그러므로, 진단 효율을 개선하고 객관적인 정량적 증거를 제공하는 정확한 자동 IHC 세포막 분석 방법에 대한 urgent need가 있다고 본다.
H&E 염색에서는 세포핵 (cell nuclei)를 탐지하는 많은 딥러닝 연구들이 제안되었음에도 불구하고 IHC 이미지에서 세포막을 분석하는 것엔 별로 관심도가 없었다.
최근 들어 몇몇의 fully supervised IHC membrane segmentation 방법들이 소개되었는데, 이는 딥러닝 기반 membrane segmentation의 우수함을 입증하였다.
그러나! fully supervision 시간 많이 들고 세포막과 세포핵 픽셀 단위로 어노테이션하는 수고 (effort) 가 엄청나게 든다. 반면에 세포핵의 중심만 어노테이션하는 건 훨씬 더 적은 수고를 요구하고 nuclei segmentation을 위한 weakly supervised learning으로 연구되었다. 그럼에도 불구하고, point annotation으로 cell membrane segmentation을 어떻게 supervise할 지는 여전히 연구중 (under investigation) 에 있다.
이 연구는 기존 방법에 비해 annotation cost를 크게 줄일 수 있는 point-based cell membrane segmentation 방법을 제안한다. (Fig. 1.)
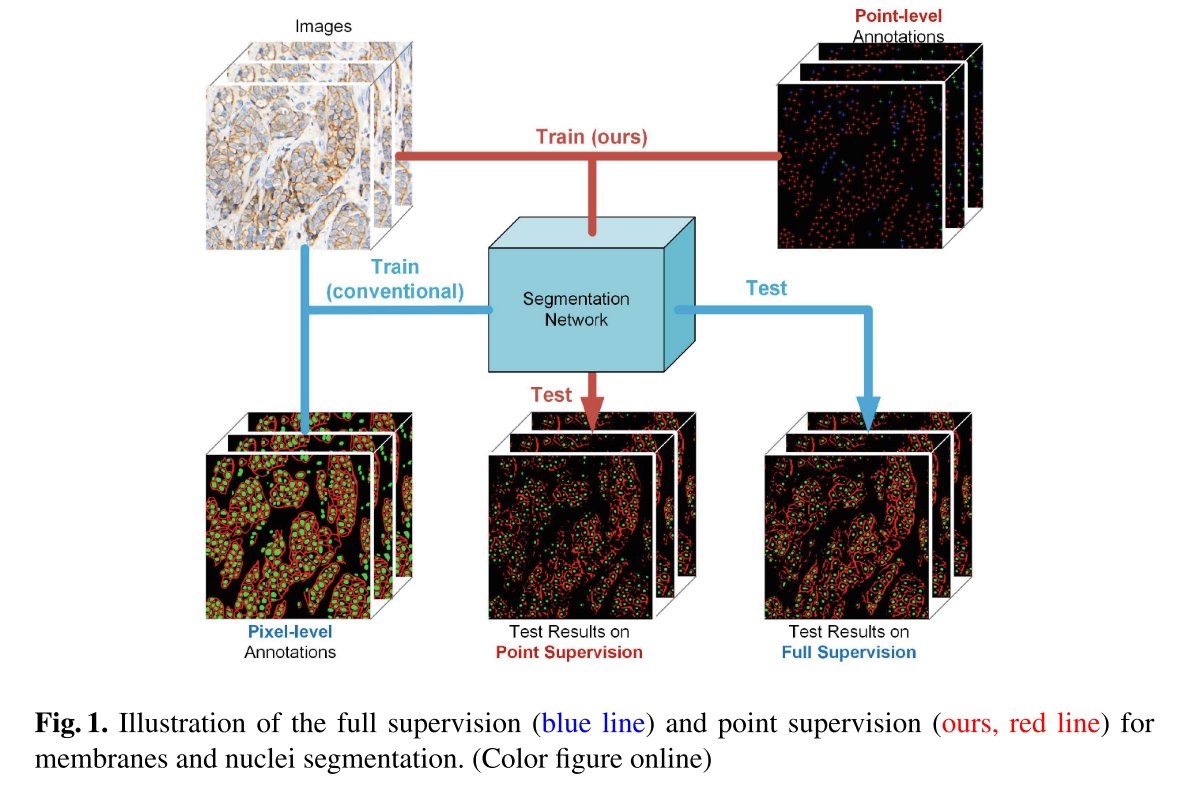
세포막의 발현 상태를 묘사하기 위해 point annotation의 category를 사용한다. (complete-stained, incomplete-staind, and unstained). 이 semantic points의 정확도가 곧바로 segmentation 결과로 이어지기 때문에, segmentation quality는 semantic points와 point anntoations 간의 loss로 묵시적으로 supervised 될 수 있다. (Fig. 2.)
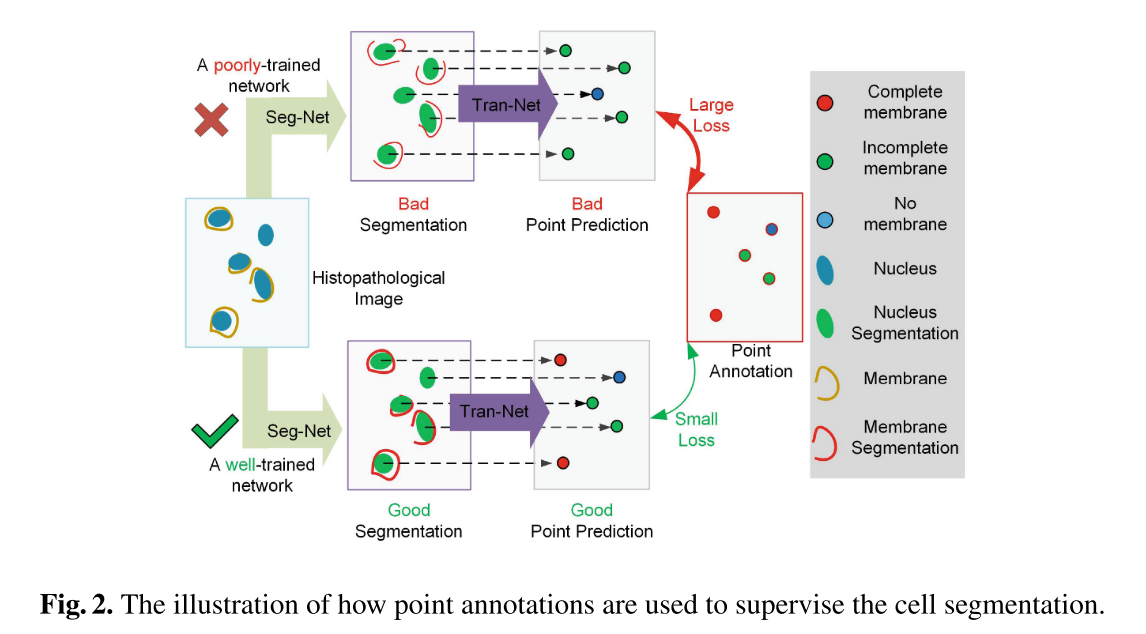
저자들이 아는 한, IHC 이미지로부터 세포막 segmentation을 하기 위해 point-level supervision을 사용하는 첫 연구다. 제안하는 방법은 point annotations으로 두 가지 objects (세포핵/세포막)를 동시에 supervise할 수 있는 것으로도 첫번째다. 광범위한 실험을 통해 fully supervised data로 훈련한 모델과 comparable한 성능을 얻었다.
2. Related Works
H&E 염색 이미지로부터 cell nuclei segmentation를 위한 딥러닝 기반 segmentation 방법들이 다양하게 개발되었다.
다만, IHC 염색에서 세포막 이미지를 분석하는 것에서 pixel-level annotation의 어려움이 있었기 때문에, 기존 연구들은 대부분 watershed, active contour, color deconvolution과 같은 정통 unsupervised algorithm들을 적용했고 성능이 그렇게 좋지 않고 staining intensity와 abnormal statining impurities에 취약하다. 최근, fully supervised 세포막 분할 방법들도 나왔는데, 데이터 어노테이션의 높은 비용은 다양한 세포막 분석 task로의 applicability를 제한한다 (limits their applicability).
조직병리학 이미지에서 세포핵 분할을 위한 annotation cost를 줄이기 위해 weakly supervised segmentation 훈련 방법들이 주목을 받았다. 그 중 첫 번째는 adversarial 기반의 unsupervised cell nuclei segmentation 방법은 H&E와 IHC 같은 시각적으로 복잡한 병리 이미지를 다루기에는 model parameter의 search space가 제약에 어려움이 있었다. 그 중 두 번째는 point annotation으로 weakly supervised 세포핵 분할 방법이다. H&E 염색에서 세포핵의 모양은 대부분 타원이기 때문에 Voronoi diagram이랑 결합된 point annotations가 반복적인 모델 훈련과 정제에서 pseudo-annotations을 생성하는 데 사용되었다. 이런 방식으로 IHC 이미지에서 세포핵을 수행하는 건 되었지만, 보통 세포막을 분할하는 데에는 효과적이진 않았다.
그러므로, 이 논문이 이 분야의 a major challenge를 해결하는 point-supervised cell membrane segmentation 방법을 제안한다. 또한, 처음으로 point supervision으로 세포핵/세포막 둘 다 분할하는 방법의 feasibility를 탐구한다.
3. Method
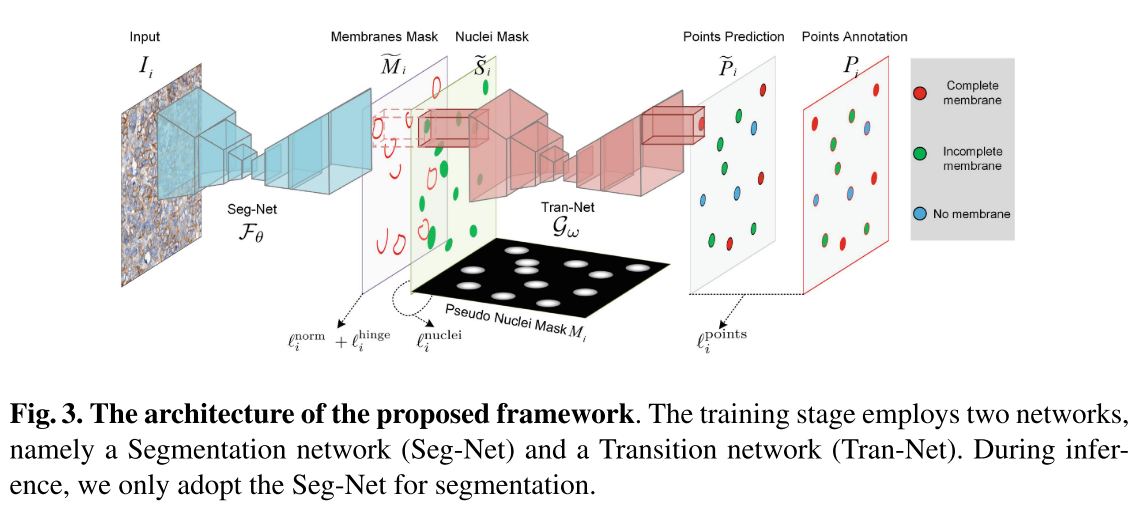
3.1. Formulation of the Point-Level Supervised Segmentation Problem
Seg-Net은 이와 같이 수식화할 수 있음.
여기서 M은 Membrane mask (H x W x 1) 이고, S는 nuclei mask (H x W x 1)
point annotation을 사용해서 세포막과세포핵을 분할하는 Seg-Net을 훈련시키기 위해 입력과 segmentation, point annotation에 대한 관계를 정립할 필요가 있다. (Fig. 3., Eq. (1))
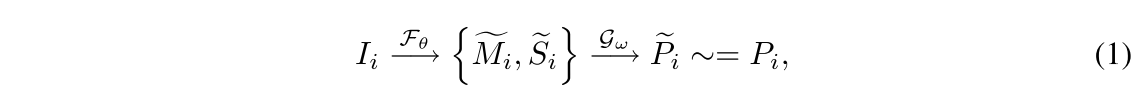
는 세포핵, 세포막 마스크를 semantic points (H x W x (c+1)) 로 변환하는 transition netwrok (Tran-Net)이다.
분할 성능이 Tran-Net에서 semantic point를 예측하는 데에 굉장히 중요하므로 point anntoation을 fitting함으로써 segmentation도 can be supervised되는 것이다.
3.2. Network Architecture
Seg-Net, Tran-Net 모두 original U-Net을 사용했는데, Tran-Net의 경우, 마지막 convolution kernel을 1x1에서 9x9로 교체했다. Tran-Net은 세포막과 관련된 세포들의 중심 포인트들인 semantic points의 category를 예측하는 것이기 때문에 kernel size를 키워 receptive field를 늘려주었다.
Inference 동안에는 Seg-Net만 사용하게 된다.
3.3. Decouple the Membranes and Nuclei Segmentation
저자들의 목표는 세포막이랑 세포핵 둘에 대한 mask 모두를 segment하는 Seg-Net을 사용하는 것이다.
근데, 두 채널이 interdependent하면 세포핵과 세포막이 inseparatively 분할되게 될 수 있게 되는데, 이걸 극복하기 위해서 단일 채널의 supervised mask 를 사용해서 nuclei segmentation을 내뱉도록 강제한다. 이 연구에서 각 point annotation을 20의 radius로 원을 만들어 가공한 를 만들고, 다른 채널은 semantic points를 예측하는 Tran-Net한테 semantic 정보를 제공하기 위해, 세포 염색 상태를 묘사하는 정보를 포함해야한다. 그렇게 해서 membrane segmentation이 decouple하게 된다.
point annotation을 가공한 로 nuclei segmentation을 supervision함으로써 다른 채널은 세포막 염색 상태를 묘사할 수 있는 정보를 담은 세포막 분할을 담당하게 만든다. L1 loss!
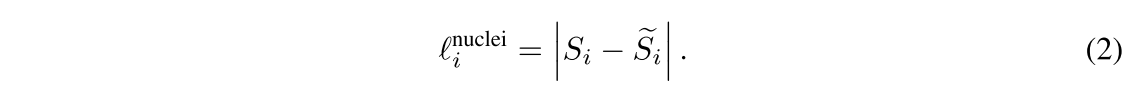
3.4. Constraints for Membrane Segmentation
픽셀 단위의 세포막 annotation이 없기 때문에, 신경망은 세포막에 대해서 원치 않는 과도 분할 (unwanted over-segmentation)을 초래할 수 있다. 과도 분할은 염색 불순물에 대한 분할, 세포핵에 대한 분할 두 가지 형태로 생길 수 있다. 이러한 이슈를 해결하기 위해 normalization loss term을 제안하는데, network가 아주 작은 염색 영역이나 세포핵을 잡지 말고 smoother한 형태로 membrane segmentation하는 걸 학습시키는 걸 독려시키는 목적이다.
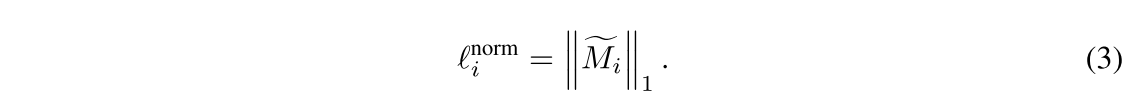
하지만, 이것에만 의존하면 trivial solution을 초래할 수 있는데, 이 손실함수는 예측하는 결과의 평균 값을 최소화하는 것이기 때문에 세포막 분할에 대한 minimal maximum confidence (e.g., 0.03) 를 초래할 수 있다. membrane mask의 L1 norm이 작게 만드는 penalty를 주는 거라 최대 신뢰도가 너무 낮은 값을 갖는 문제를 이야기하는 것 같다.
이러한 이슈를 방지하기 위해, hinge-loss-like loss function을 제안한다.
hinge-loss function의 hyperparameter 는 더 큰 는 더 작은 기댓값에 해당하게 되는 결과의 기댓값을 결정한다. 예로 가 1이나 2면 결과의 기댓값은 각각 1 이나 0.5가 된다. 를 적절한 값으로 결정하므로써 normalization loss term의 negative impact를 방지할 수 있다.
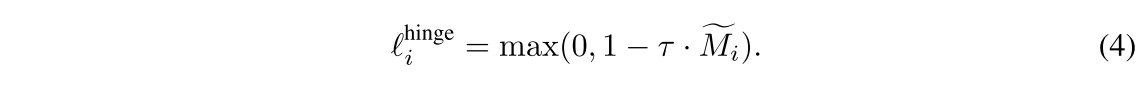
식에서 를 0.5라고 생각하면 membrane mask는 hinge loss를 줄이는 방향으로 2에 가까운 값을 가져가야함.
결과의 기댓값을 조절하는 차원의 scale constraints로 봐도 될 것 같다.
3.5. Point-Level Supervision
Tran-Net으로 세포핵 중심을 찾는 건 전형적인 semantic points detection task이다. 다른 점은 Tran-Net의 input이 Seg-Net의 output라는 점이다. 그럼에도 불구하고 point-level supervision에 대해서 cross entropy를 도입했다.
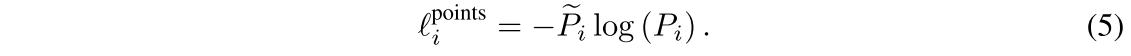
각 point annotation의 공간적 지도 정보 (spatial supervision information)을 강화하기 위해 각 point annotation을 5 픽셀 radius의 Gaussian circle로 확장했다.
3.6. Total Loss
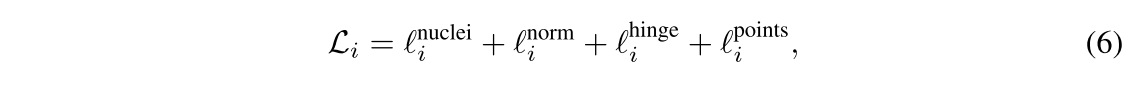
normalization term과 hinge loss term은 적대적으로 hinge loss에서 는 1로 설정하면 둘의 이상적인 값은 0.5에 가깝다. (고로, membrane mask의 기댓값도 0.5에 수렴한다는 뜻?)
4. Experiments
PD-L1, HER2 두 개의 IHC 세포막 염색 데이터셋에서 실험을 수행했음.
HER2 실험은 segmentation 성능을 검증하는데에 쓰이고 반면에 PD-L1 실험은 segmentation results를 임상적으로 관련깊은 indicators (clinically relevant indicators)로 변환하는 것의 효과를 검증하기 위해 사용되었다.
4.1. Dataset
400배율 WSIs에서 1024 x 1024 해상도의 이미지 648 HER2, 1076 PD-L1 이미지를 수집했고 PD-L1은 병리학자가 세포막 염색에 기반해 positive, negative만 분류한 point-level annotation만 있고 HER2 data는 pixel-level과 point-level annotation을 둘 다 가지고 있는데, 병리학자가 세포핵과 세포막을 pixel-level로 윤곽을 그리고 (delineate) 세포막 염색을 기반으로 세포를 분류한 point-level annotation이 있다.
Pixel-level annotation은 testing과 fully supervised 실험에만 사용되었다. 데이터를 1:1로 학습 세트과 평가 세트로 나누고 pixel-level anntoations 없이 훈련시키기 때문에, validation set은 사용하지 않는다.
4.2. Implementation Details and Evaluation Metric
모델 훈련
- 모델 최적화; Adam optimizer with initial learning rate of 5 x 10^-4, momentum 0.9, 50 epoch
- 데이터 증강; 512 x 512로 random crop, random rotation/flip
- 손실함수; hinge loss term에서 는 1로 설정 됨
평가 metric; IoU segmentation metric과 pixel-level F1 score.
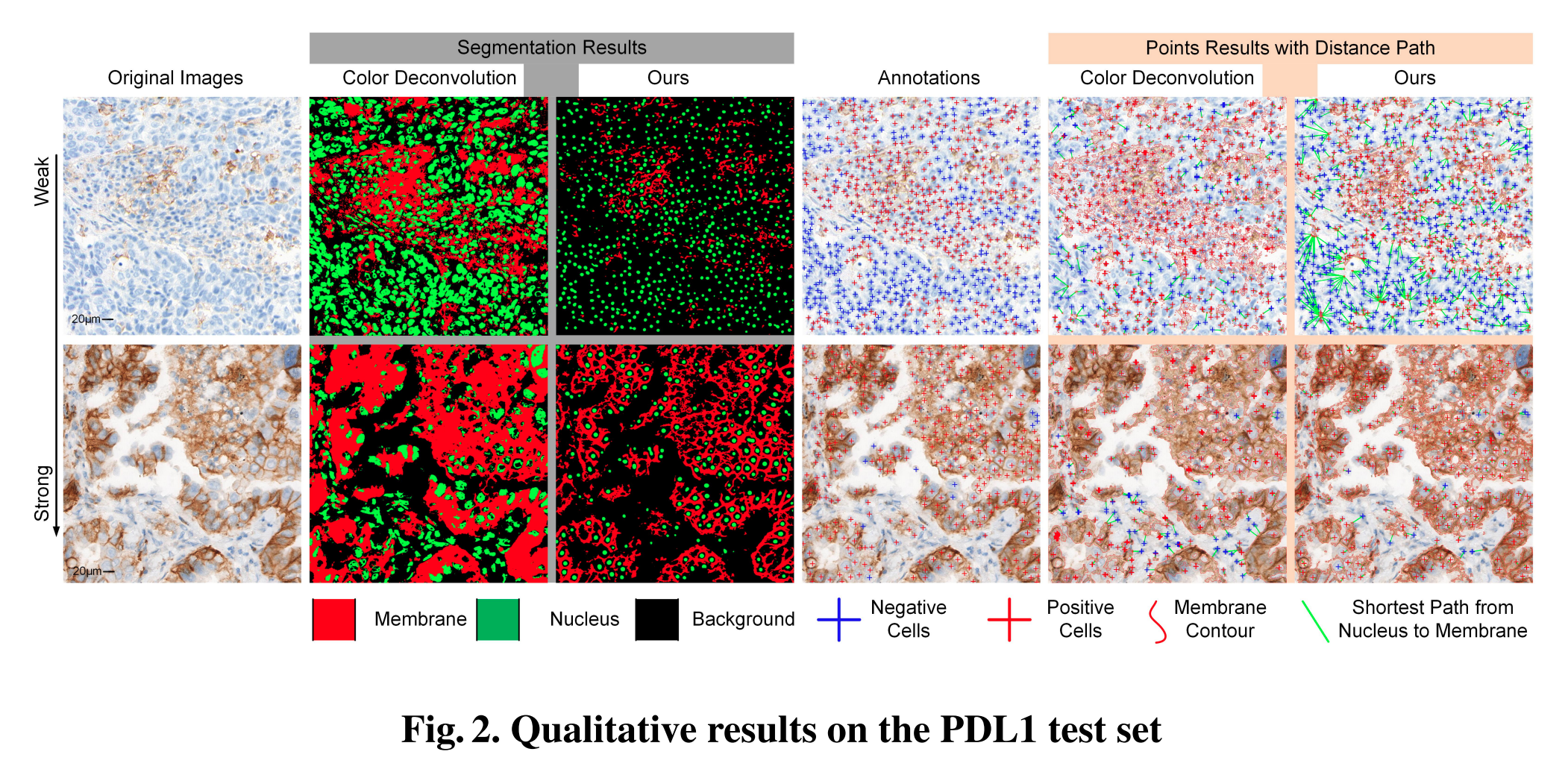
PD-L1 데이터셋은 point-level annotation밖에 없으니 segmentation을 key point predictions으로 변환함으로써 point-level에서 segmentation performance를 평가할 수 있었다.

각 세포의 중심점과 membrane 사이의 최소 거리를 계산하고 20 pixel보다 멀면 negative로 하는 process로 거쳐 key point predictions를 얻은 것으로 보임 (Supplementary Fig. 1.)
Point Result의 그림이 align이 안 되어있음.. 조금 더 확대되어서 아래가 짤린 것으로 보임.
4.3. Result Analysis
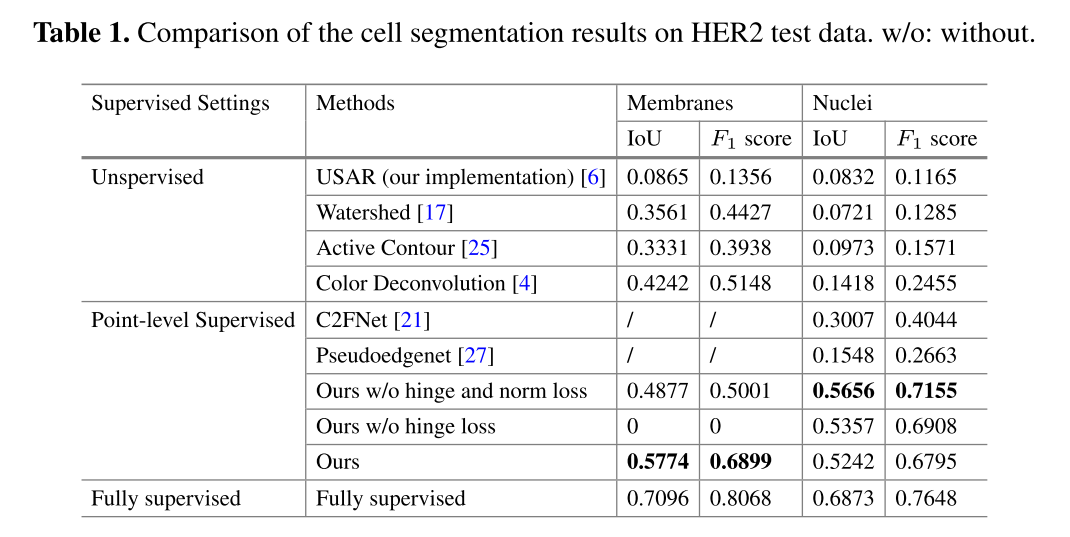
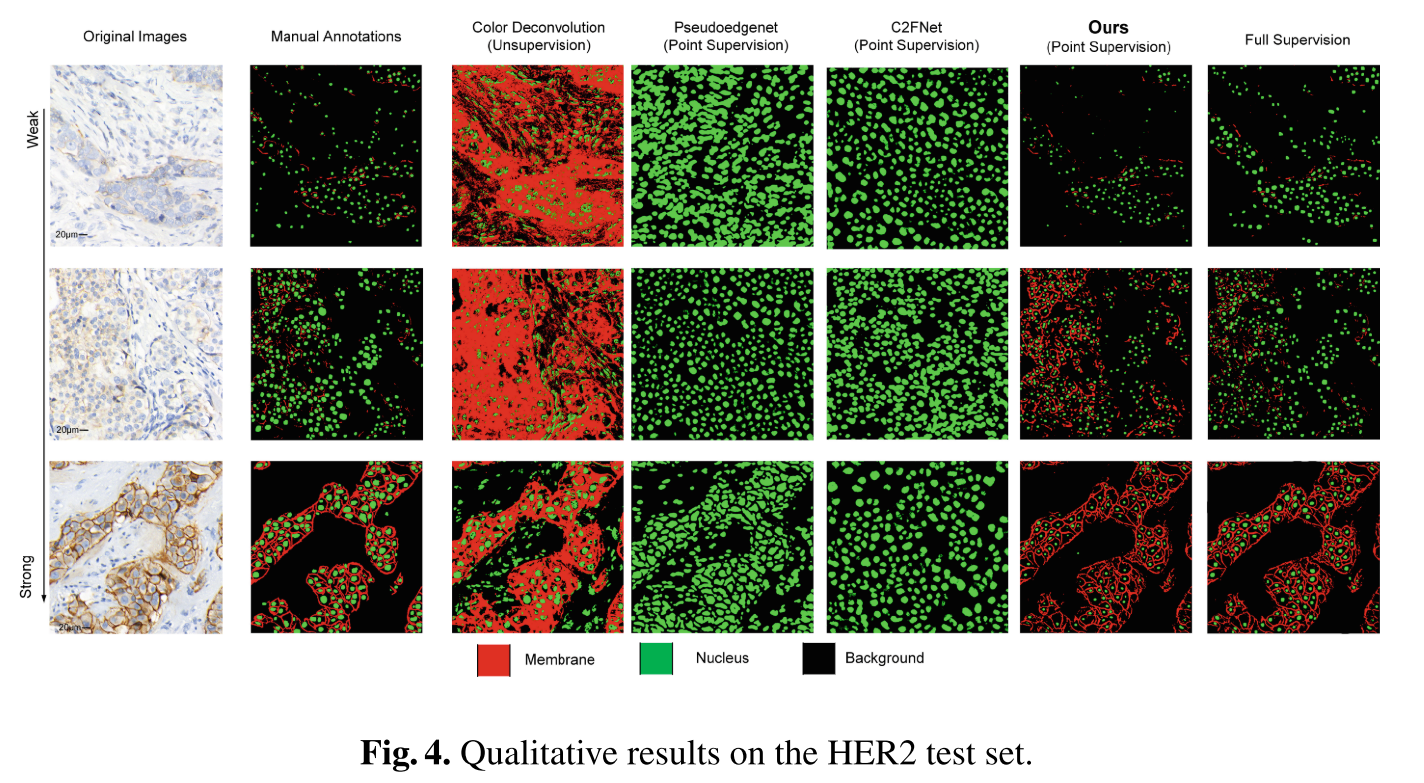
HER2 데이터셋 정량적 평가 결과, IoU score of 0.5774 and an F1 score of 0.6899 for membranes, and an IoU score of 0.5242 and an F1 score of 0.6795 for nuclei의 성능을 달성하며 unsupervised 방법들과 다른 point-level 방법들을 앞섰다.
ablation study를 보니 분할 성능을 개선하는 것에서 hinge loss와 normalization loss가 중요한 역할을 했다. normalization loss랑 hinge loss가 둘 다 없으면 또 어느정도는 되는데, normalization loss만 넣으면 학습이 안 되고 hinge loss로 기댓값을 원하는 값으로 만들어주니 수치 성능이 꽤나 오른 것으로 보임
annotation cost가 적은 기존 방법들을 뛰어넘긴 했지만, Fully supervised에 비해 수치 성능이 낮아보이긴 한다. 샘플의 결과 예시로 보면 그렇게 거슬리는 건지는 모르겠다. medium intensity에서 좀 더 예민하게 잡은 느낌은 있는데, weak, strong에서는 거의 흡사하다.
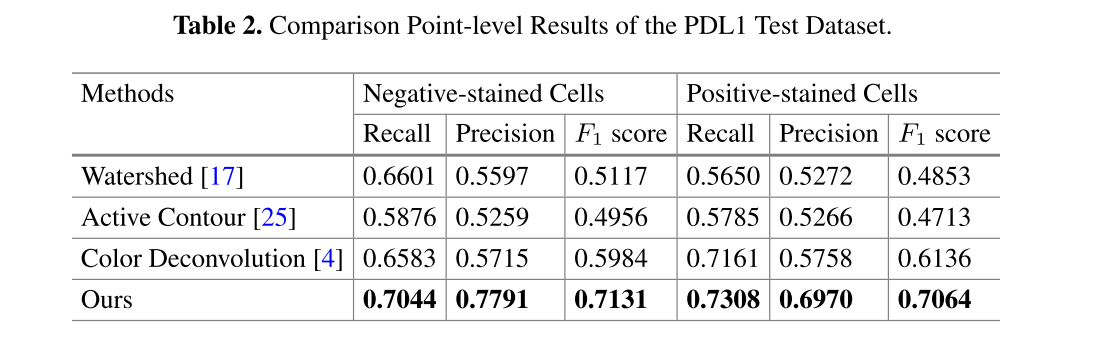
PD-L1 데이터셋 정량적 평가 결과, F1 scores of 0.7131 and 0.7064 for negative and positive-stained cells, respectively
Table 2에서 비교한 방법들에 비해서는 point annotation이 들어갔으니, 당연히 잘해야되는 건 맞다. 문제는 0.7 정도의 평균 F1 score면 병리학자들이 만족하냐이다.
(Supplementary Fig. 2.)
5. Conclusion
이 연구는 point-level supervision만 사용해서 IHC 세포막 염색 이미지에서 세포막과 세포핵을 정확하게 분할하는 a novel method를 선보였다.
fully supervised pixel-level annotation 방법들과 견줄 정도의 성능을 보이면서 다른 pixel-level supervision 방법들과 전통적인 unsupervised 방법들을 앞서는 성능을 보였다.
세포핵 중심으로 인위적인 원을 만들어 supervision을 하다보니 세포핵의 모양/크기를 흉내내는 것은 힘들지만, 세포막의 상태에 대한 semantic points를 예측하는 loss로 이전 입력에 해당하는 세포막 분할을 supervision하는 방법으로 샘플로 보여준 예측 결과 (Fig. 4.) 에도 fully-supervised와도 비슷한 결과를 보이고 있다는 게 굉장히 고무적이다. 세포막은 거의 비슷한 것 같다.. 세포핵의 어떤 status도 point-level category로 annotation할 수 있으면 가능할 지도 모르겠다?
중국 선전에서 열렸던 MICCAI 2019에 견학갔을때, point supervision으로 nuclei segmentation하는 방법론을 카카오인가 네이버가 발표하는 게 인상깊었는데, 그 사이에 기술의 진보가 더 이루어졌음이 느껴진다.
