[2021 Computerized Medical Imaging and Graphics] Deep Multi-Magnification Networks for Multi-Class Breast Cancer Image Segmentation
Paper Review

1. Introduction
- Breast CARINOMA
- 현재의 대부분 deep learning은 single magnification에서의 좁은 영역에서의 individual patch를 보기 때문에 성능이 잘 나오지 못할 수 있다.
- 이 연구에서는 pathologist가 현미경으로 병리 슬라이드를 분석하는 방식과 닮은 Deep Multi-Magnification Networks (DMMNs)을 제안한다
Computational pathology
- Histopathologic feature는 nuclear feature, celluar/stromal architecture나 texture와 같은 pattern 기반 identification을 포함한다
- Computational pathology는 nuclear segmentation에서 크기, 모양, 그들간의 관계 등과 같은 nulcear feature를 추출하기 위해 사용되어옴.
- Adaptive thresholding과 morphological operation을 통해 nuclei density가 높은 영역을 찾아 nuclei segmentation을 하기도 했었음 (Petushi et al., 2006)
- 겹쳐있는 nuclei와 lymphocyte를 segment하기 위해 region, boundary, shape를 기반으로 한 integrated active contour가 제안되기도 했음
- 전립선 조직에서 gland (샘) classification and segmentation이 소개한 연구에선 nuclei, cytoplasm, lumen에서의 structural and contextual features를 artifcat, normal gland, cancer gland를 구분하는 데에 사용하였음
- 이런 nuclei segmentation 기반 접근방식은 nuclei의 모양과 cancer 영역의 structure가 WSI에서 뽑은 tissue sample에 따라 큰 variation을 가지고 있어서 challenging하다.
Single-magnification patch-based DL 연구
- WSI를 Deep Learning (DL) 모델 input으로 사용할 수 없어 보통 일정한 size의 patch를 추출해서 사용한다
- CAMELYON16 challenge에서 우승자는 tissue region을 추출할 때 otsu thresholding을 썼고 tumor/non-tumor를 patch-level로 구분하는 patch-based model을 훈련시킴
- 성능을 향상시키기 위해서 tumor/non-tumor patch 간의 class를 balance있게 했고, rotation, flip, color jittering과 같은 data aumentation을 사용함
- CAMELYON17 challenge 우승자는 patch-overalpping strategy를 개발했는데,
- breast WSIs에서 subtype을 segment하기 위해 하나의 패치가 더 넓은 영역을 포함한 추가적인 더 큰 패치와 함께 processed 됨
- 대안으로, 패치간의 representation을 share하기 위해 patch에서 만들어진 feature들을 결합하는 representation-aggregation CNNs도 개발되었었음
- 하지만, patch-based 접근방식은 realistic하지 않는데 pathologist는 좁은 영역만 보지 않고 zoom level을 자주 바꾸면서 분석하기 때문이다.
DSMN꽈 DMMN을 그림으로 비교한 그림이다. DMMN은 256 x 256에서 20x, 10x, 5x를 볼 때, 더 넓은 영역을 볼 수 있게 함
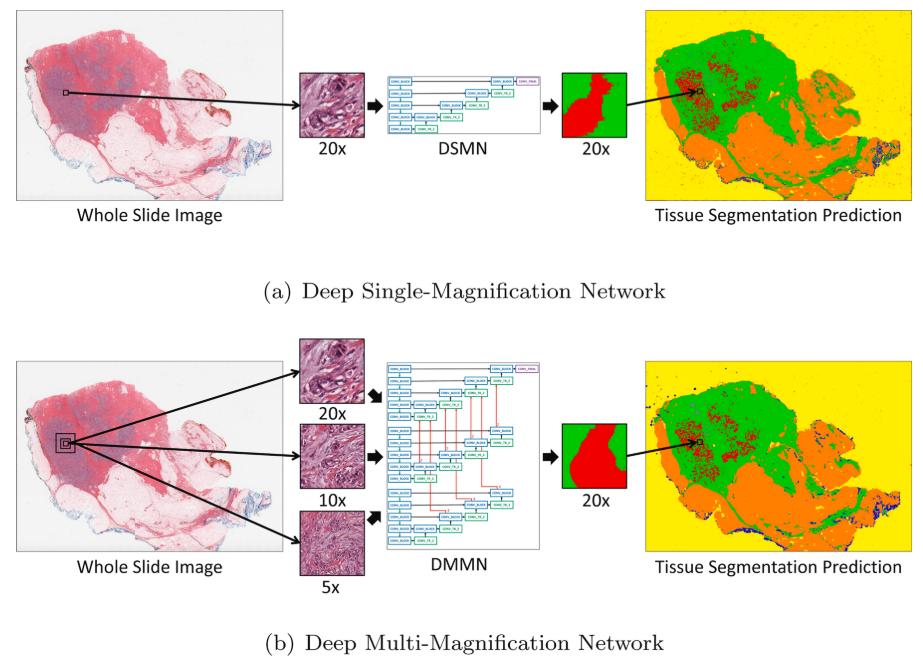
Multiple-Magnification을 사용한 연구들
- four encoders for different mag. and one encoder in tumor region segmentation (CAMELYON dataset)
- 더 최근에는 three expert networks for different mag. + a weighting net to autmatically select weights to emphasize specific mag. based on input pathcs + aggregating net to produce final segmentation prediction
- 하지만, intermediate feature map은 서로 공유되지 않았고 이는 여러개의 배율에서 온 feature map들을 충분히 사용하지 않는다
This study
- 이 연구는 breast cancer를 식별하는 목표와 함께 breast tissue 이미지에서 multiple subtype을 segment하는 것으로 DMMN을 제안하는데,
- DMMN은 multiple encoders, multiple decoders, decoder간의 multiple concatenations로 intermediate layer에서 더 풍부한 feature map을 가진다.
Contribution
- 다양한 magnification에서의 feature map을 결합한 DMMN으로 더 정확한 segmentation
- annotation time을 줄이면서도 높은 성능을 보이는 partial annotation을 제안
2. Method
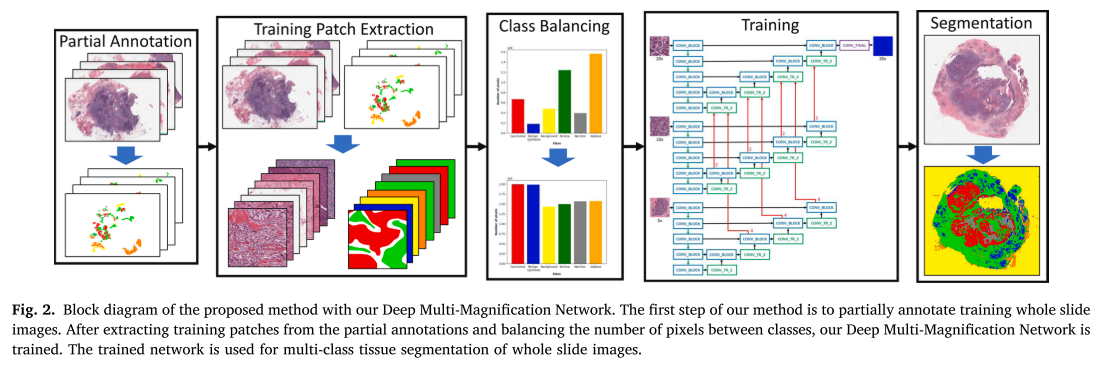
Partial Annotation
- 이 unlabeled region들의 thickness를 최소화하면서 subtype간의 가까운 경계 영역 (close boundary regions)은 annotation하지 않았음 -> 서로 다른 subtype이 가까이 있으면 경계를 annotation하지 않음으로써 하얀 틈을 주는 느낌
- cropping 하지 않고 전체 subtype component들을 annotation했음

Training Patch Extraction
- 256 x 256 in 20x 기준으로 center에서 256 x 256 10x, 5x 추출 three magnification
- 해당 target (output) patch의 1% (pixel)이상 annotation 되어있으면 추출됨
- non-overlap하게 추출
Class Balancing
- biological 구조의 구불구불한 형태때문에 biomedical에서 data aug로 널리 쓰이는 *elastic defromation을 사용하여 class간의 annotation된 pixel의 수를 맞춰주었다.
- grid points는 17 x 17, 로 setting
*elastic deformation 예

Architecture
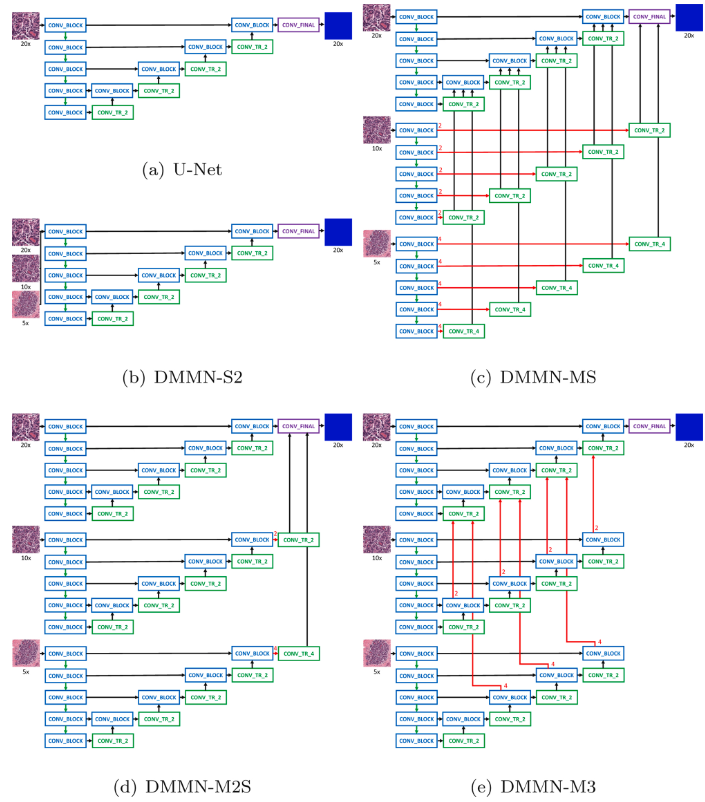
(a)는 U-Net
(b)는 Single-Encoder Single-Decoder (DSMN-S2)인데, multi magnification input을 사용
(c)는 Multi-Encoder Single-Decoder (DMMN-MS), 10x, 5x encoder에서 각 conv_block의 feature map을 center crop해서 single encoder단에서 conv_block 가져와서 concat할 때 같이 concat에서 사용하는 느낌
(d)는 Multi-Encoder Multi-Decoder Single-Concatenation (DMMN-M2S), finaly layer에서의 feature map만 사용
(e)는 이 연구가 제안하는 Multi-Encoder Multi-Decoder Multi-Concatenation (DMMN-M3), 10x, 5x decoder단에서 conv_tr_2와 conv_block이 concat된 것을 다시 center crop해서 20x decoder단의 conv_tr_2단에 concat하는 느낌
CNN training
- weighted cross entropy 사용
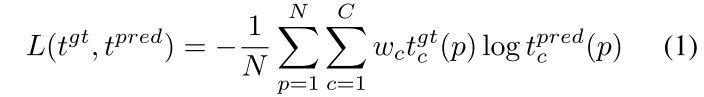
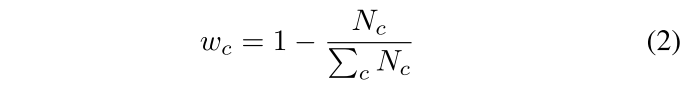
- SGD optimizer, learning rate: , momentum: , weight decay: , epochs
- validation set에서 mIOU가 가장 큰 CNN 모델이 final model로 선택됨
- 훈련 중에 augmentation으로 rotation, vertical and horizontal flip, brightness, contrast, color jittering 사용
Multi-Class Segmentation
- 20x, 10x, 5x magnification에서 256 x 256 pixels 사이즈의 patch 만들기 위해 1024 x 1024를 input patch가 추출됨 (아마도 1024 x 1024 5x 에서 center crop하는 식 10x, 20x를 추출하고 5x와 10x를 256 x 256으로 downsample한 것 같음)
- 왼쪽 가장자리를 start point로 잡고 256 pixel stride (step)로 input patch가 추출되었음
- Zero padding이 WSI 가장자리의 patch를 뽑는데 사용되었음 (1024-256 = 768 정도 padding을 양쪽에 줘야 가장자리에서 256 x 256 in 20x을 center로 잡고 뽑을 수 있을 것 같음)
- background 영역을 제거해 segmentation process를 빨리 하기 위해 Otsu thresholding이 사용됨
3. Experiment Results
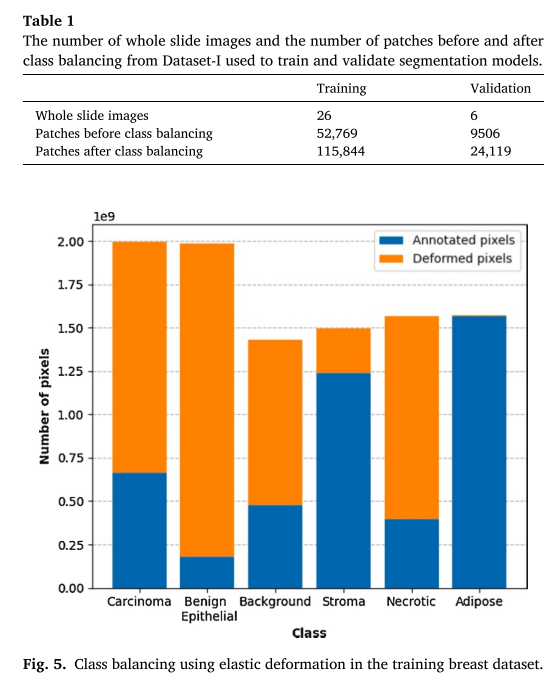
데이터셋, 모델훈련
- Dataset-I (모델 훈련): large invasive ductal carcinoma (IDC) 영역을 포함한 Triple-Negative Breast Cancer (TNBC) dataset
- Dataset-II (추가 검증): IDC와 ductal carcinoma in situ (DCIS)를 포함한 lumpectomy와 breast margin dataset
- 둘 다 H&E stained WSI
- TNBC - 0.4979 mpp in 20x at Aperio XT scanner, breast margin dataset - 0.5021 mpp in 20x at Aperio AT2 scanner
- TNBC dataset split (Total: 38 -> TR: 26, VA: 6, TS: 6)
- 6 classes (TNBC): carcinoma, benign epithelial, background, stroma, necrotic, and adipose
- rates of elastic deformation: carcinoma (2), benign epithelial (10), background (1), stroma (0), necrotic (3), and adipose (0)
- Two Deep Single Magnification Networks (DSMNs) : SegNet과 U-Net
- Four Deep Multi-Magnification Networks (DMMNs) : DMMN-S2 (Single-Encoder Single-Decoder), DMMN-MS (Multi-Encoder Single-Decoder), DMMN-M2S (Multi-Encoder Mult-Decoder Single Concat.), DMMN-M3 Proposed (Multi-Enc. Multi-Dec. Multi-Concat.)
예측, 평가
- adipose (지방)이 pixel intensity가 약해 otsu thresholding에서 제외돼서 TNBC dataset에서 otsu thresholding을 사용 안 했고 하나의 WSI에 대해 segmentation하는 데에 15일 걸림
- 55 testing images from Dataset-I and 35 testing images from Dataset-II
- IOU, Recall, Precision을 사용
Quantitative Result
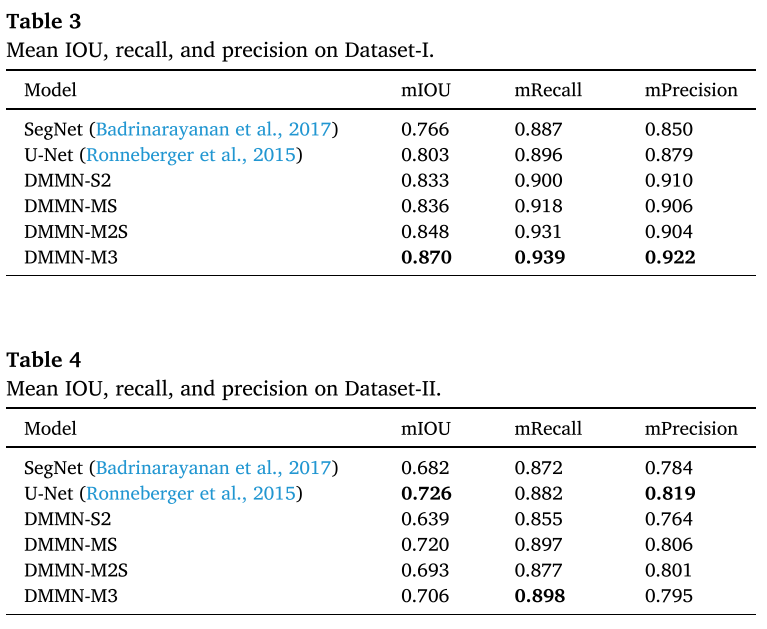
- TNBC dataset (Dataset) 에서는 제안하는 M3 (Multi-Enc. Multi-Dec. Multi-Concat.)이 outperform하였다. 특히 morphological하게 비슷한 carcinoma와 benign epithelial이 challenging한데 그걸 잘 구분했다.
- breast margin dataset에서 carcinoma를 철저하게 annotation해고 그게 전체 pixel에서 0.188% 정도였는데, DSMN-S2, DMMN-M2S는 low precisin을 보였는데, non-cancer 영역을 cancer로 false positive하는 게 많았다.
- DMMN-MS는 이 데이터셋에서 가장 좋은 성능을 보였는데, TNBC에서는 그렇지 않았음
- proposed architecture, DMMN-M3가 두 데이터셋 모두에서 good carcinoma segmentation performance를 보였고 unseen dataset에서 성공적으로 generalize하고 있다는 걸 보인다..? (rich하게 feature map을 사용해서 TNBC에 더 fit한 게 아닌가)
Examples of Prediction Result
- 그림은 Z 방향으로 input -> label (partial annotation) -> DSMN (Deep Single Magnification Network = U-Net -> DMMN-MS (Multi-Encoder Single-Decoder) -> DMMN-M2S (Multi-Encoder Mult-Decoder Single Concat.) -> DMMN-M3 Proposed (Multi-Enc. Multi-Dec. Multi-Concat.)
- carcinoma in red, benign epithelial in blue, background in yellow, stroma in green, necrotic in gray, adipose in orange
- Dataset-I (TNBC dataset) 에서의 결과: Figure 6, 7, 8, 9
- Dataset-II (lumpectomy and breast margin dataset) 에서의 결과: Figure 10, 11, 14
- Ground Truth에서 white background는 unlabel을 뜻함
- Dataset-II는 necrotic, adipose, background가 제외되었음. 1) large necrotic region이 없고 2) Otsu thresholding으로 adipose와 background는 없어졌음
Figure 6 - Dataset-I
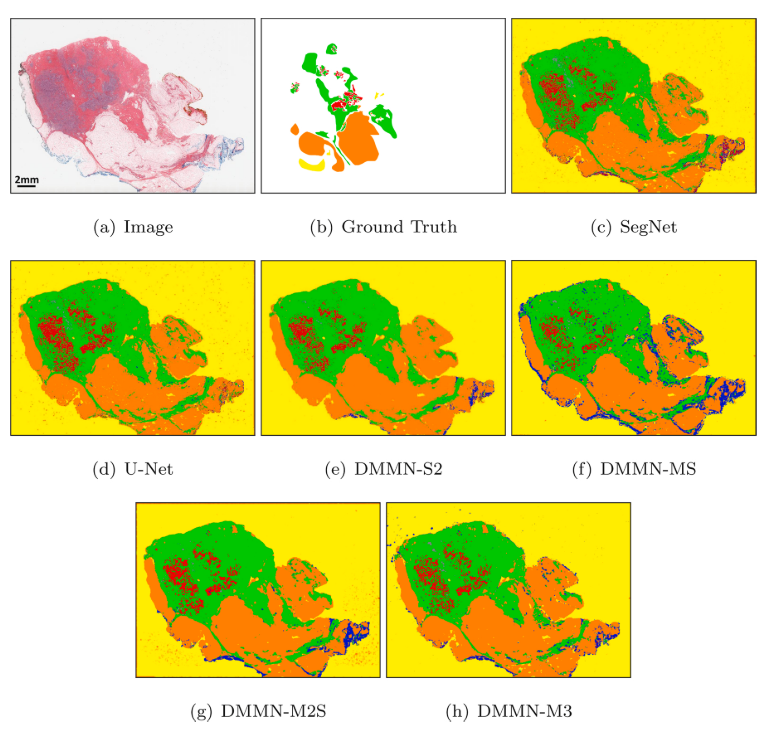
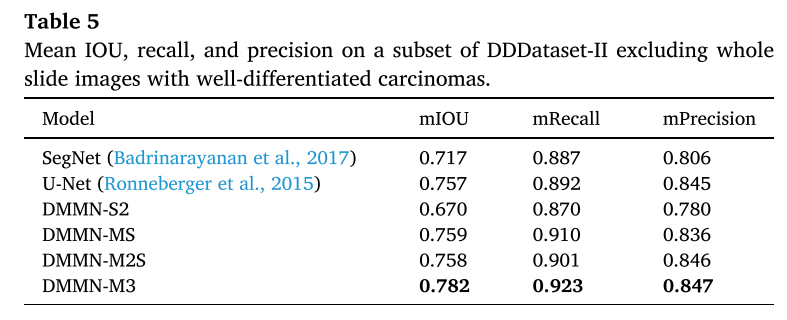
Figure 7 - Figure 6 WSI에서 invasive ductal carcinoma를 포함한 patch에 대한 모델별 segmentation 결과

Figure 8 - Dataset-I
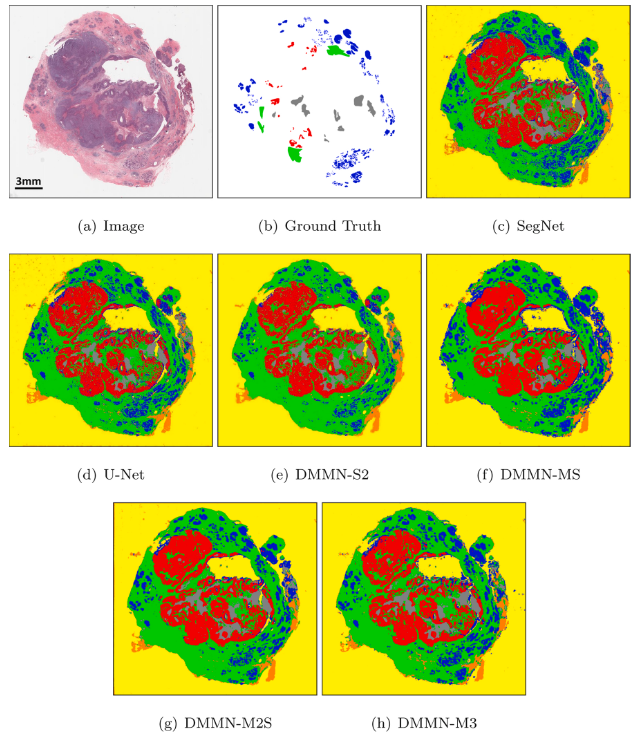
Figure 9 - Figure 8 WSI에서 benign epithelial 포함 patch에 대한 모델별 segmentation 결과
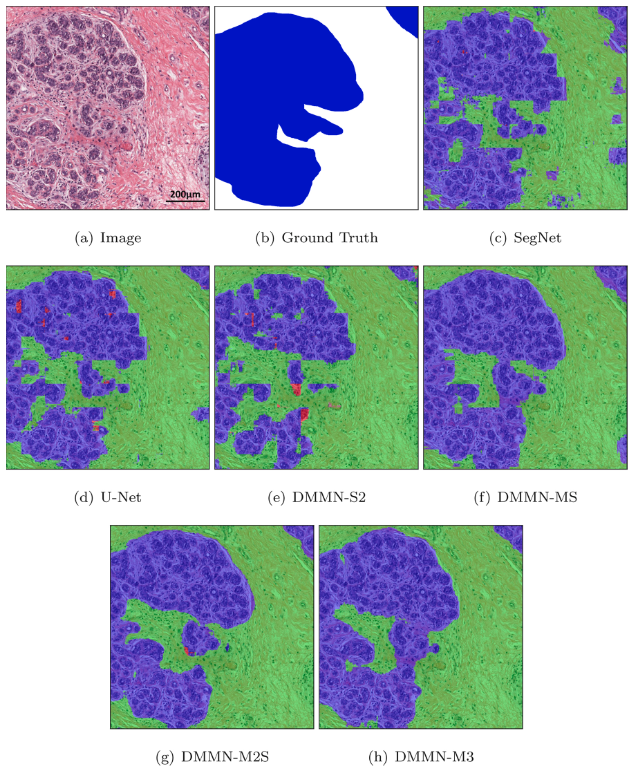
Figure 10 - Dataset-II

Figure 11 - Figure 10 WSI에서 ductal carcinoma in situ 포함 patch에 대한 모델별 segmentation 결과
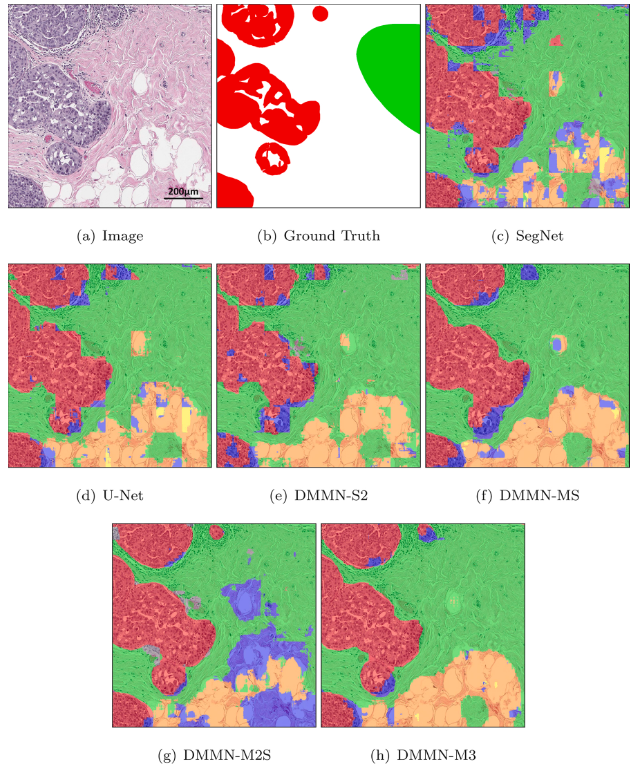
Figure 14 - Well-differentiated carcinoma에 대해 benign epithelial (blue) 로 잘못 예측하는 DMMN-M3
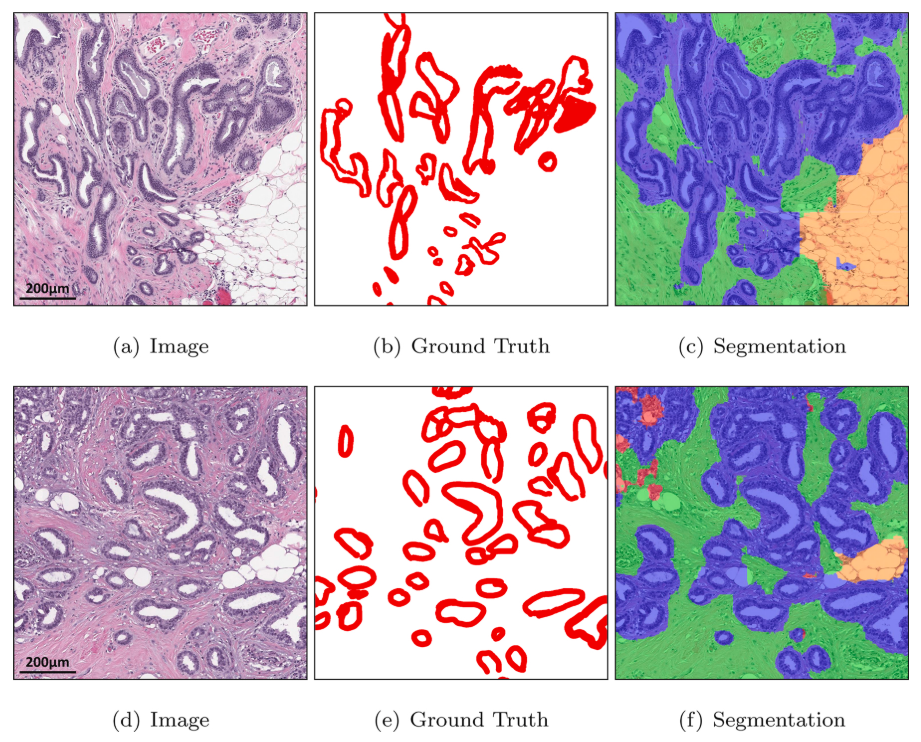
- WSI에서 봤을때, unlabel된 white region을 background (yellow) 아니면 adipose (orange)로 보는 느낌이 강함
- DSMNs는 narrow field-of-view 때문에 subtype 간의 blocky boundary를 형성하는 것을 볼 수 있음 (low mIOU, low mRecall, low mPrecision in Dataset-I)
- DMMNs가 smoother boundaries를 가진다. (Figure 7, 9)
- Dataset-II에서는 DMMN-M2S이 큰 영역에서 benign epithelial로 잘못 segmentation된 것을 보였는데, DMMN-M3는 smoother and clearer boundaries를 형성한 것을 알 수 있었다.
- DMMN-M3가 많은 well-differentiated carcinoma pixel에 대해 benign epithelial로 잘못 segment하면서 성능이 낮아졌는데, well-differentiated carcinoma는 Dataset-I에 없어서이기 때문이다. (unseen data에 대해 취약할 수 있다?)
- 전체적으로 DMMN-M2S, DMMN-M3가 잘 예측했다고 하는데, 특정 sample들을 pick한 느낌이 강하다
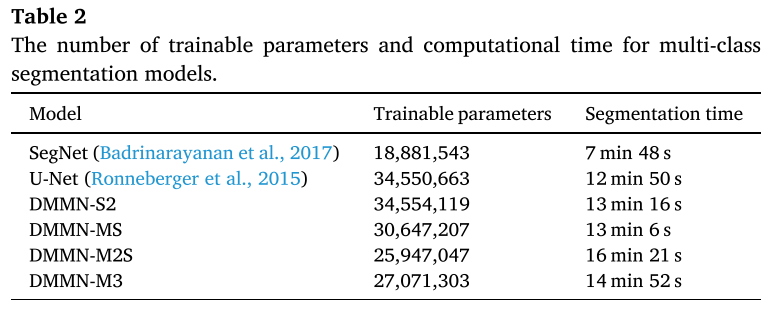
# of Trainable Parameter and Segmentation Time
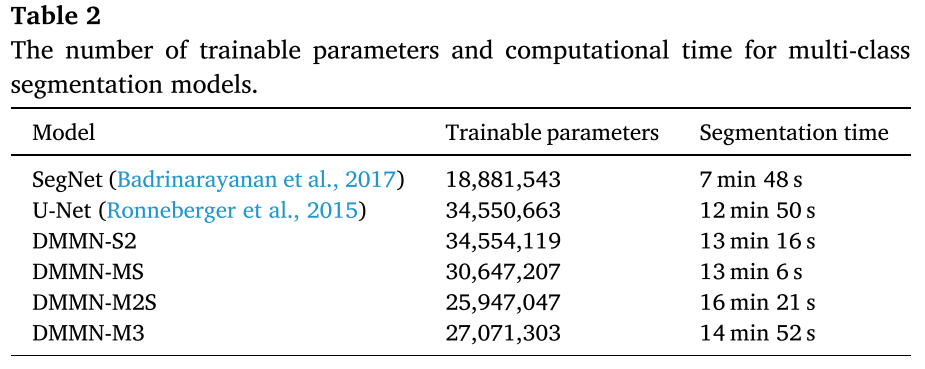
- 31,500 patch가 나오는 53,711 x 38,380 pixels의 WSI를 single NVIDIA GeForce GTX TITAN X GPU에서 사용해서 걸린 시간
- U-Net이랑 DMMN-S2가 parameter 수가 가장 많고, M2S, M3는 parameter수가 좀 적은 대신 segmentation 하는 데에 시간이 좀 오래 걸린다.
4. Conclusions
- Deep Multi-Magnification Network는 Deep Single-Magnification Network보다 성능이 좋음
- 특히나 intermediate layer에서의 feature map을 rich하게 쓴 제안하는 알고리즘 DMMN-M3 (multi-encoder multi-decoder multi-concatenation)
- partial annotation으로 annotator에게 시간과 노력을 줄이면서도 모델은 spatial characteristics과 class간의 spatial 관계를 배웠다..?
- 학습데이터에 없는 유형인 well-differentiated carcinoma에 대해 성공적으로 segment하지 못했고, background noise에 민감해서 다른 스캐너에서 digitize된 WSI라면 잠재적으로 mis-segmentation을 초래할 수 있다.
5. Comments
Performance
- single magnification이 narrow view를 보면서 그걸로는 부족하다고 생각했었는데, multi-magnification을 input으로 사용하고 feature map을 추출해 aggregation하는 게 사용하고 있는 데이터셋 상에서 더 좋은 성능을 낸다는 건 분명하다는 걸 입증해줬음
- single-mangification만 사용한 모델과 input단에서 multi-magnifiaction을 단순히 concat한 blocky한 boundary를 형성하고, multi-magnification로 intermediate layer에서 rich feature map을 사용한 모델은 smoother boundary를 형성해서 더 좋은 성능
Computational Cost
- U-Net이 3500만 훈련파라미터, DMMN-M3가 2700만 훈련파라미터로 rich한 feature map을 사용하는 데에도 훈련파라미터수는 더 적다?
- multi-encoder (MS) + multi-decoder (~ M2S) + multi-concatenation (~ M3) 순으로 heavy해지는 거 아닌가..?
- inference할 때는 DMMN-M2S (16분 21초), DMMN-M3 (14분 52초), U-Net (12분 50초)
- batch size는 각각 어떻게 넣은 거지? single GPU로 20 epoch만 돌렸다는 데 그래도 되나
Robustness
- U-Net이 Dataset-II에서 좋은 성능을 냈는데, 오히려 unseen type에 대해 robust하다고 해석할 수 있다.
- Multi-magnification으로 중간 중간 featuremap을 끌어다가 결합하는 식으로 다양한 magnification에서의 rich한 feature map을 배운다는 게 그 데이터셋을 더 잘 학습하면서도 fit될 수도 있다고 해석할 수 있다.
- Multi-magnification을 input에서 concat에서 쓴 DMMN-S2보면 Dataset-II에서 성능이 크게 떨어진다. SegNet보다도 낮은 수준
Partial Annotation?
- partial annotation이 효과가 있다는 걸 보여주려면 entire? annotation이랑 비교를 했었어야 하지 않을까 이건 잘 모르겠다
