[MIA 2020] Triple U-Net: Hematoxylin-aware Nuclei Segmentation with Progressive Dense Feature Aggregation
Paper Review
1. Introduction
Digital Pathology 이미지는 diagnosis, prognosis, metastasis 등과 임상 예측에 널리 쓰이고 있다.
특히, 이런 임상 application에서 Nuclei segmentation은 WSI에서 nuclei pattern들의 morphological signature를 분석함으로써 vital한 역할을 한다.
segmented cell을 통해 tumor cell, epithelial cell, immune cell들의 분포를 측정하며 malignant tissue를 시각적으로/정량적으로 평가할 수 있게 한다.
하지만 nuclei segmentation은 여전히 이런 면에서 어렵다.
1. non-uniform manual operation
ex) manual한 H&E 염색과 다른 digital scanner로 인해 color inconsistency와 unclear nucleus boundaries가 발생함
2. malignant cell의 growth rate (성장률?)이 높을수록, normal tissue보다 neoplastic tissue에서 malignant cell의 밀도가 엄청 높아지게 되고, tumor cell과 normal cell 사이의 squeezing
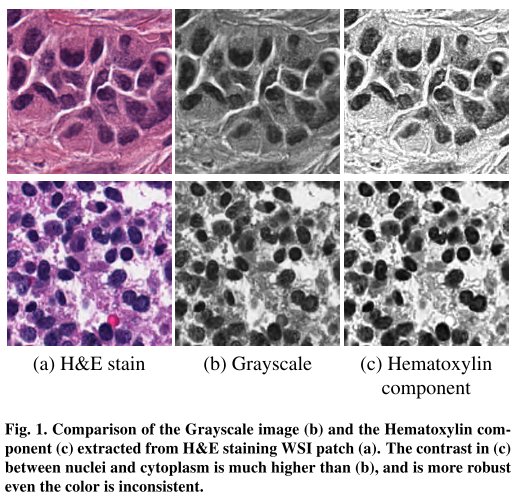
Beer-Lambert's Law에 따라 Ruifork가 제안한 color decomposition 방법으로 Hematoxlylin을 추출하면 좀 더 분명한 nucleus view를 얻을 수 있음.
그리고 nuclei와 cytoplasm, stroma 사이의 contrast가 gray-scale보다 hematoxylin component를 뽑았을때 훨씬 크게 되면서 오히려 color inconsistency에 robust하다
저자가 말하는 main contribution
- color normalization이 필요없는 Hematoxylin-aware network with Triple U-Net architecture for nulcei segmentation
- Hematoxylin 성분이 더 정확한 nucleus boundaries를 가지는 segmentation 결과를 예측하기 위해 좀 더 정확한 contour awareness를 제공한다
- 다른 도메인의 feature들을 progressively aggregate하는 PDFA 모듈 제안함
2. Related Works
2.1. CNN-based Natural Image Segmentation
Long et al., 2015 (FCN): only convolutional layers, input of arbitrary size images
Zhao et al., 2017 (PSPNet): global context by pyramid pooling module
Chen at al., 2017, 2018 (DeepLab series): atrous(dilated) convolution, CRF...
He at al., 2017 (Mask-RCNN): extending the region proposal based object detection task to instance seg
Liu et al., 2018 (PANet): adaptive feature pooling method to propagate the features from the encoding phase to the proposal subnetworks
Yang et al., 2019 (Deeperlab): a single-shot, bottom-up image parser to process the semantic and instance segmentation tasks jointly
2.2. CNN-based Medical Image Segmentation
CT, MRI, ultrasound, whole slide image
WSI -> cell-level, tissue-level
Ronneberger et al., 2015 (U-Net)
Zhou et al., 2018: replaced the simple skip connections with dense skip connections
Li et al., 2018: hybrid densely connected U-net for CT volume segmentation
Man et al., 2019: RL with geometry aware U-net to activate the searching strategy for CT
2.3. Nuclei Segmentation
Naylor et al, 2018: formulated the segmentation problem as a regression task of distance maps
Graham, Rajpoot, 2018 (Sams-net) : stain-aware loss tailor-made for the stain variations in H&E images
Graham et al, 2019 (HoVer-net) : nuclei segmentation과 classification을 동시에 하는 architecture
Liu et al, 2019 : panoptic model incorporating instance segmentation with semantic segmentation
Zhou et al. 2019 (CIA-Net): contour supervision -> more precise nucleus bondaries
3. Method
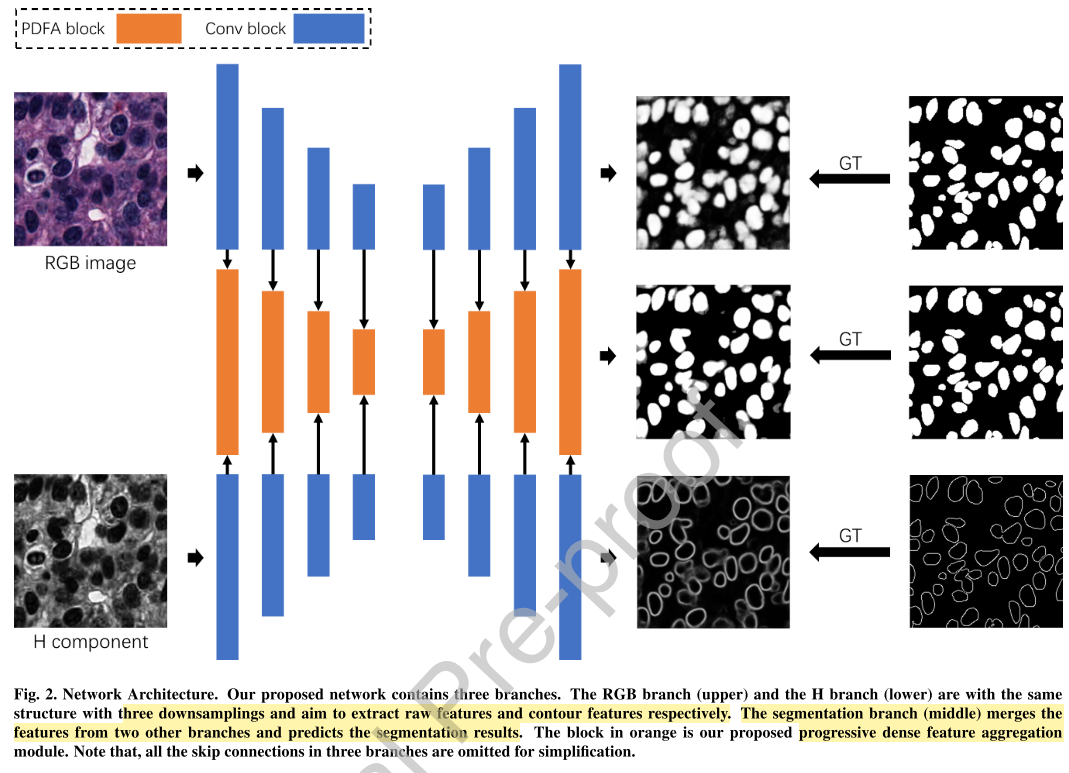
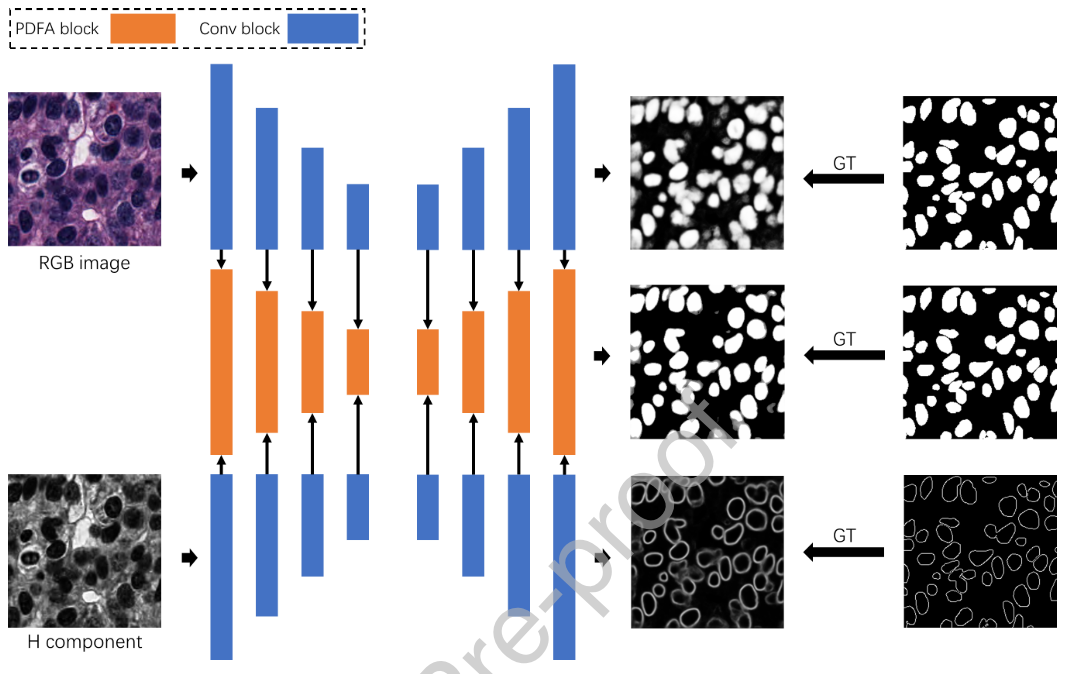
RGB branch
- segmentation task의 raw feature를 추출
H branch
- nucleus edge detection task의 Hematoxylin-aware feature 추출
- Hematoxylin component는 color inconsistency에 strong robust해서 color normalization 사용 안 함
Segmentation branch
- 위 두 개의 feature를 fuse해서 최종 segmentation 결과를 예측
3.1. Hematoxylin Component Extraction
각 RGB 챼널의 gray level이 light transmission rate T와 linear하다고 가정한다
(는 transmitted light, 는 incident light)

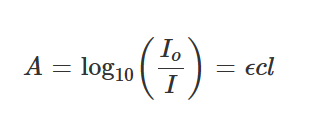
= Molar extinction coefficient
= Path length
= Concentration of sample
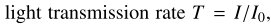
각 특정 stain은 각 RGB 채널에 빛의 absorption factor c로 나타낼 수 있고, Beer-Lambert's Law를 사용해 stain들과 그의 absorption 사이의 관계를 모델링할 수 있음
각 RGB채널의 에너지 I는 다음과 같이 적을 수 있음
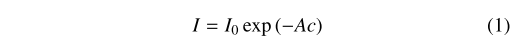
비선형성 각 채널의 상대적인 세기의 비선형성 때문에 이미지의 세기 값인 I는 stain color decomposition에 바로 사용될 수 없음. 그래서 absorbing stain의 concentration과 linear한 specimen optical density 를 계산한다.
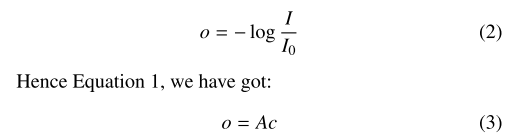
hematoxylin 염색은 RGB 각 채널에서 [0.18, 0.20, 0.08]의 value가 나온다. 이를 통해 RGB를 각 특정 stain의 color space로 mapping할 수 있다.
이 연구에서는 Ruifrok et al. (2001)에서 제안한 color convolution method로 Hematoxylin component르 추출했다.
3.2. Progressive Dense Feature Aggregation Module
RGB branch와 Hematoxylin branch의 feature를 결합해서 segmentation branch를 형성하는데 densly connection을 가지는 softer feature fusion strategy인 Progressive Dense Feature Aggregation (PDFA)를 제안한다.
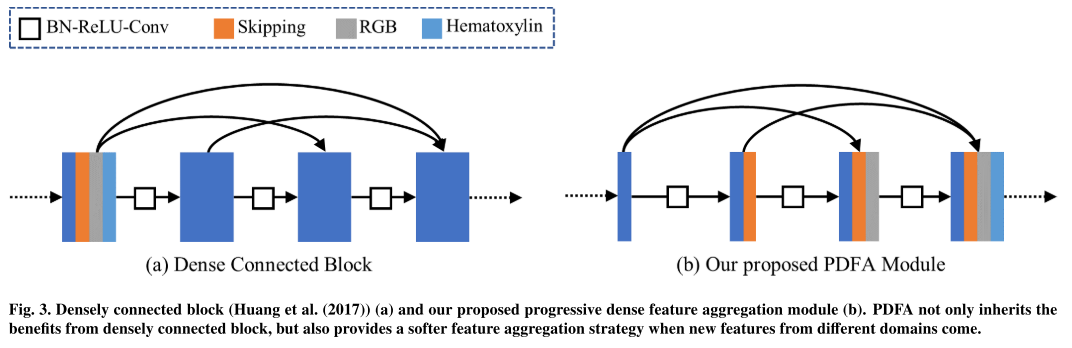
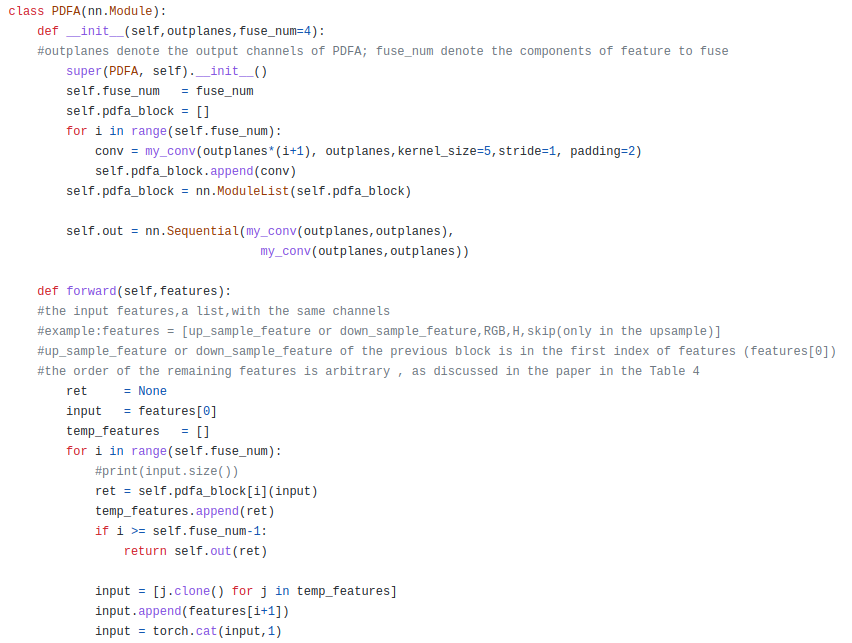

conv(feature_1)
conv([conv(feature_1), feature _2])
conv([conv([conv(feature_1), feature _2]), feature_3])
conv = BN-ReLU-Conv
feature_1 = 그 전 block에서 나온 downsample or upsample feature
feature_2 = hematoxylin feature
feature_3 = RGB feature

conv(feature_1)
conv([conv(feature_1), feature _2])
conv([conv([conv(feature_1), feature _2]), feature_3])
conv([conv([conv([conv(feature_1), feature _2]), feature_3]), feature 4])
conv = BN-ReLU-Conv
feature_1 = 그 전 block에서 나온 upsample feature
feature_2 = sementation branch encoder에서 온 feature
feature_3 = hematoxylin feature
feature_4 = RGB feature
skipping은 segmentation encoding branch에서 나온 feature map들을 뜻함
코드는 hematoxylin 먼저 들어감
3.3. Loss function

3.3.1. RGB branch
Binary Cross Entropy Loss

3.3.2. H branch
Soft Dice Loss (좀 더 nuclear contour 정보를 학습하는 데에 집중시키기 위해)
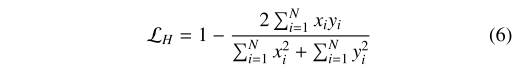
3.3.3. Segmentation branch
CIA-Net (2019)에서 제안한 Smooth Truncated Loss랑 Soft Dice Loss랑 Soft Dice Loss
pixel-level nuclei segmentation을 위한 manual annotation에서 mislabelled nuclei와 inaccurate boundaries는 불가피한데, Smmoth Truncated Loss가 outlier로부터 inference를 suppress할 수 있다고 함
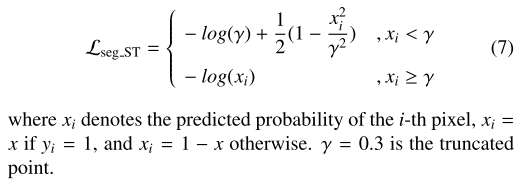
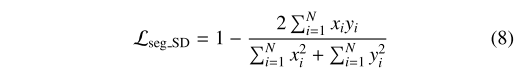
3.4. Implementation and Training Details

4. Experiments
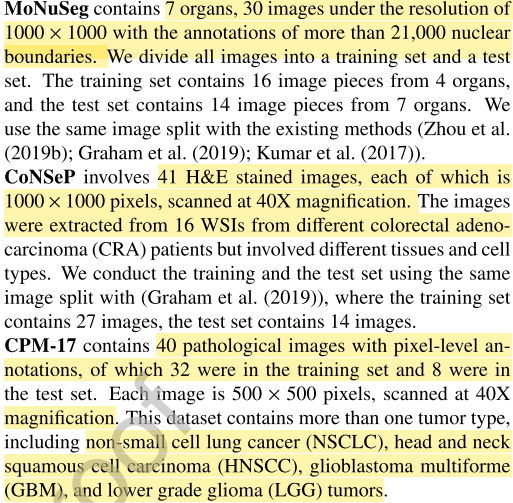
당시 SOTA인 HoVer-Net과 CIA-Net을 제치는 결과, color normalization 없이
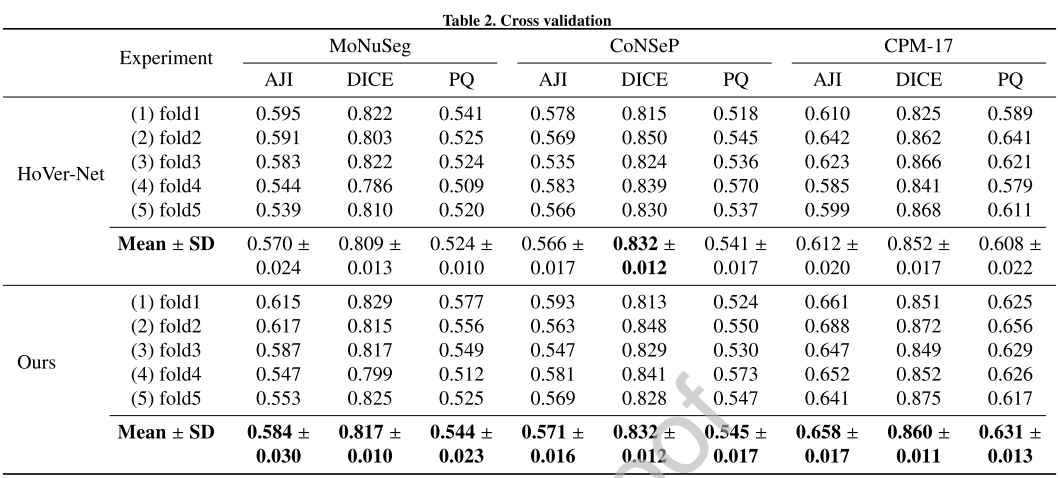
그 중 HoVer-Net과 5-fold CV로 비교해도 전체적으로 성능이 앞서더라
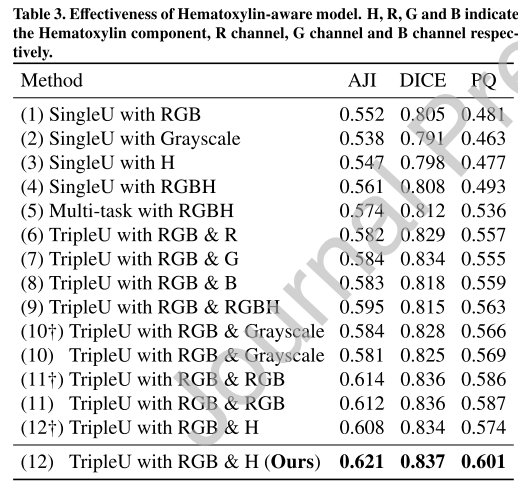
RGBH가 가장 많은 정보를 담고 있으니 Single U에서 잘 되고
RGBH로 contour detection이랑 nuclei segmentation multi-task로 시키면 더 성능이 개선되고
거기다가 feature aggregation을 통해 segmentation branch를 더 만드는 Triple UNet frame이 성능을 더 개선함
십자가 표시는 color normaliztion을 해준 것인데, Triple UNet에서 hematoxylin input에 대해서 해주면 오히려 성능이 많이 떨어진다..?
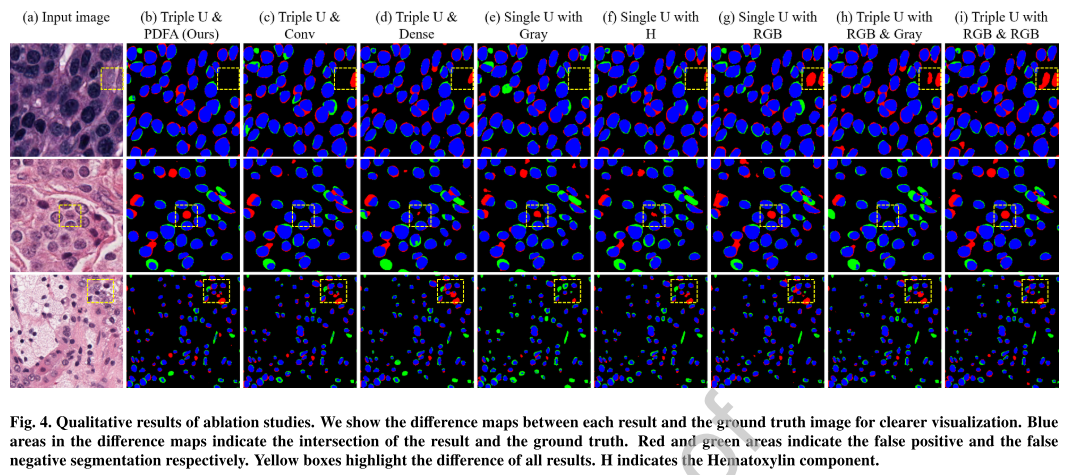
ST Loss랑 Soft Dice Loss 같이 쓰면 성능 오르더라
HoVer-Net에서는 ST Loss를 쓰면 오히려 성능이 떨어지는데, multi-task network (np, hover, nc) 로 하나의 encoder에서 뻗어나가는데, 하나의 specific task에 크게 constraint을 주면 network 훈련의 불균형을 초래했을 것을 추정함.
각각 다른 branch로 나온 feature map을 결합하여 만든 segmentation branch에서는 그런 문제가 발생하지 않는다.
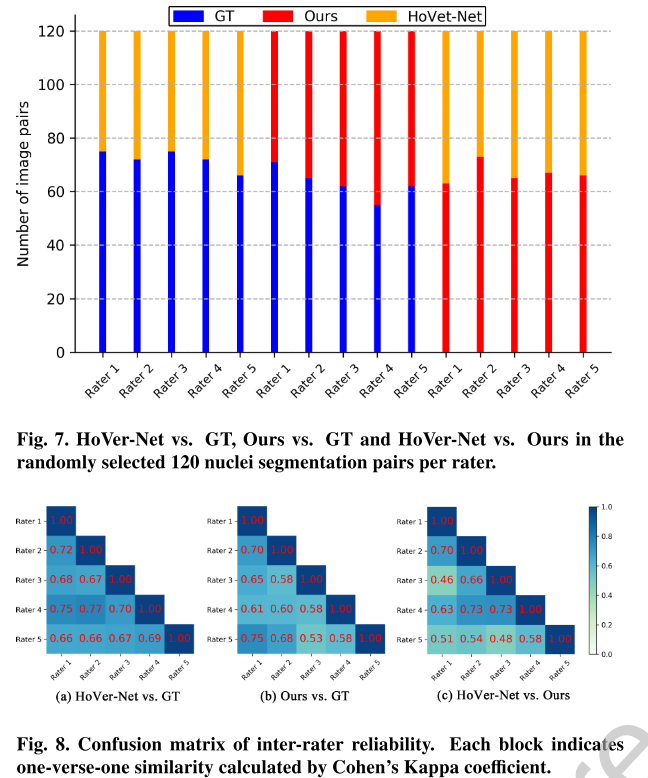
120장 random하게 뽑아서 5명의 rater를 대상으로 GT vs Ours, GT vs HoVer-Net, Ours vs Hover-Net을 비교했을 때 전반적으로 Ours가 HoVerNet 결과보다 우세했다.
음 근데 Kappa coefficient로 inter-rater reliability를 봤을때 뭐가 더 낫다라고 하는 것의 상관성이 HoVer-Net vs GT가 더 높아보이는데..?
5. Conclusion
contour (nuclear edge)를 detection하는 Hematoxylin branch를 따로 구성해 이를 cell segmentation하는 RGB branch와 Progressively 결합하는 Triple UNet은 SOTA를 달성했다. Hematoxylin component는 color inconsistency에 robust하여 제안하는 알고리즘에선 더 이상 color normalization이 필요없다.
Hematoxylin component가 color inconsistency에 robust하다는 것..
각 task별로 network를 온전히 두면서 각각에서 나오는 feature map을 aggregation하는 것이 효과적일 거라는 intuition
DAB 덩어리를 정확히 예측하는 network를 따로 두고 이런 식으로 DAB-aware triple UNet을 해도 괜찮지 않을까?
