[2022 npj Digital Medicine] Unleashing the potential of digital pathology data by training computer-aided diagnosis models without human annotations
Paper Review
목록 보기
10/25

1. Introduction
Annotation Cost
- 딥러닝 알고리즘 성능 개선을 위해 필요한 annotation 양을 medical specialist가 해내지 못한다 (lack of availability)
Using Weak Label, MIL
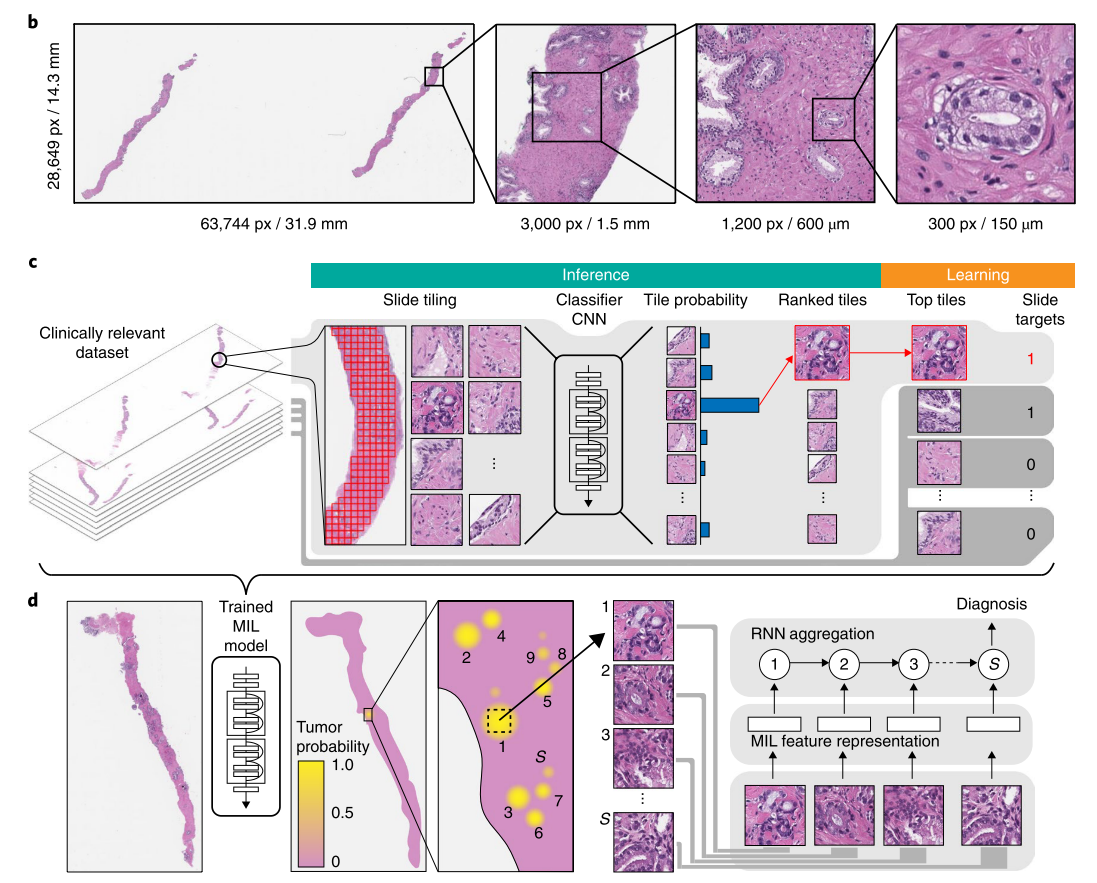
- 2019년 Nature Medicine에 게재된 Campanella et al.의 연구는 weak label을 사용하는 Multiple Instance Learning (MIL)을 적용해서 암 분류 AUC = 0.986로 좋은 성능을 달성했는데,
- weak label에 pathologist에 의해 만들어진 것이며 binary class (cancer vs non-cancer)라는 것의 한계를 지적하고 있음
Contribution
- Diagnostic Report에서 Weak Label을 추출해 이를 CNN 기반 Multiple Instance Learning 방식으로 훈련하는 알고리즘을 제안
- Human Annotation 없이 Diagnostic Report와 Pathology Image 정보로 했을 때와 Pathologit의 Annotation로 했을 때와 성능에서 통계적으로 유의미한 차이가 없음을 보여줌
Overview of Proposed CAD Algorithm
2. Results
Data and Generating Weak Labels using SKET


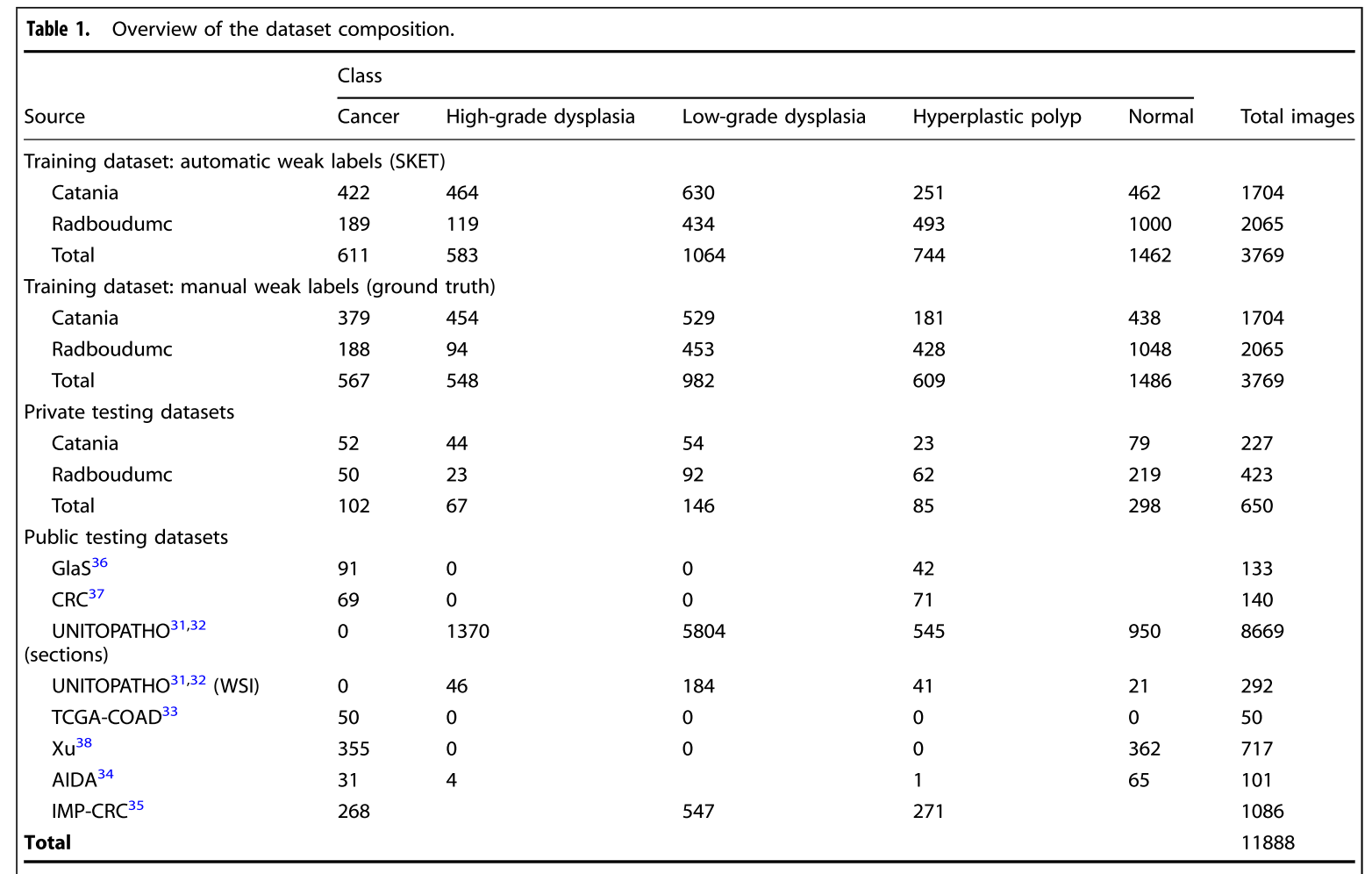
- 초당 3.19 report에 대한 weak label 생성
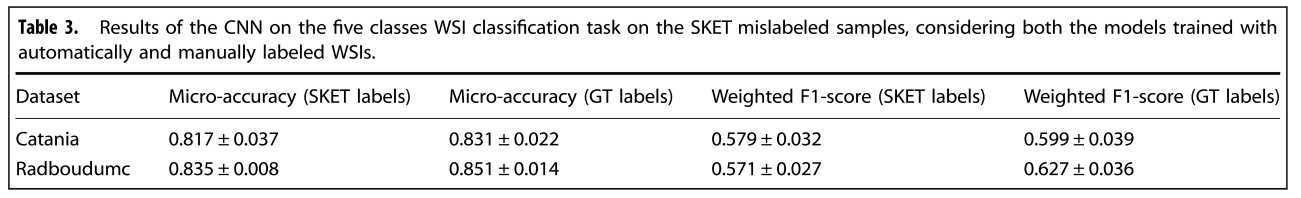
WSI-level performance
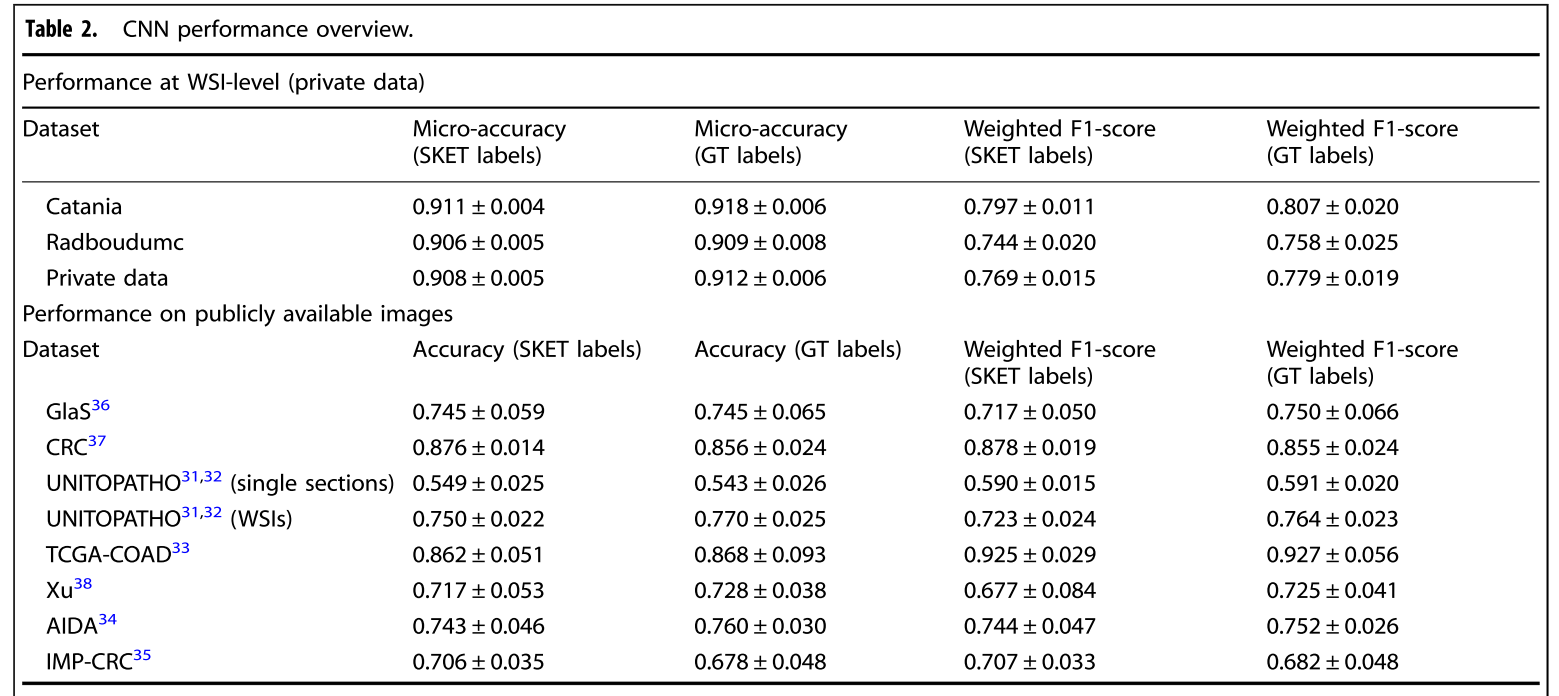
Per-class (Patch/WSI) performance
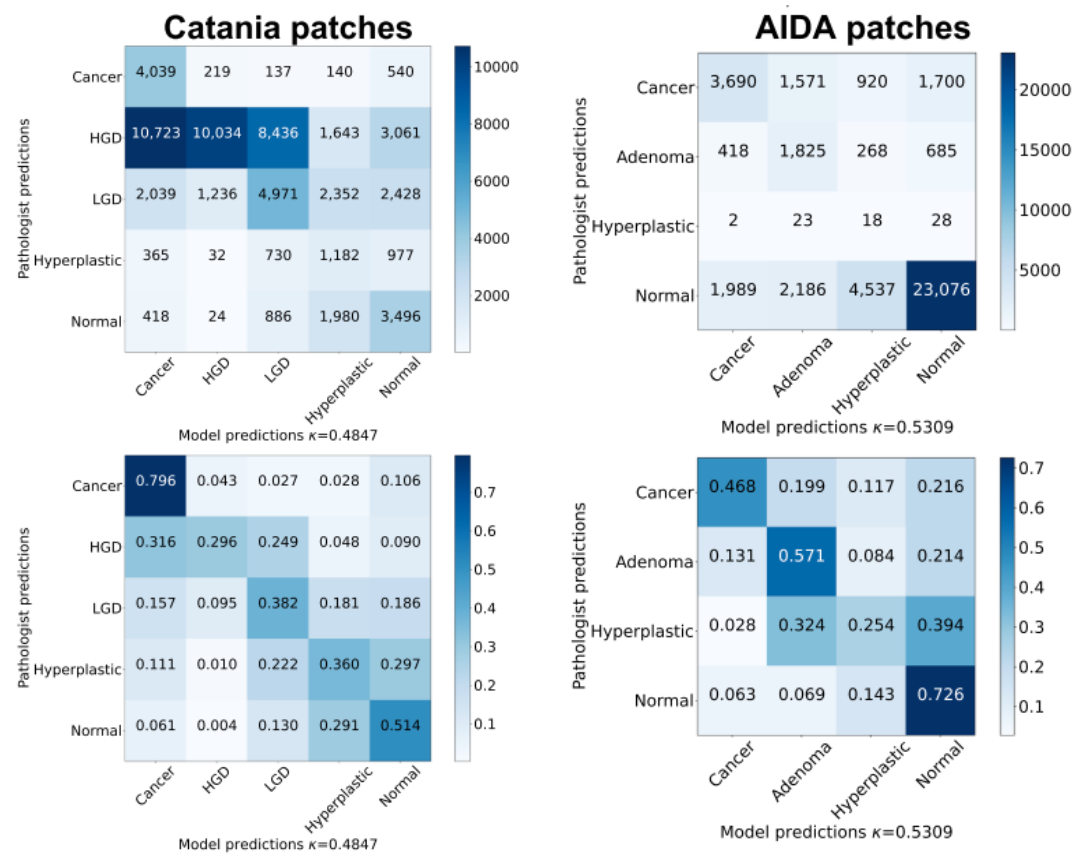
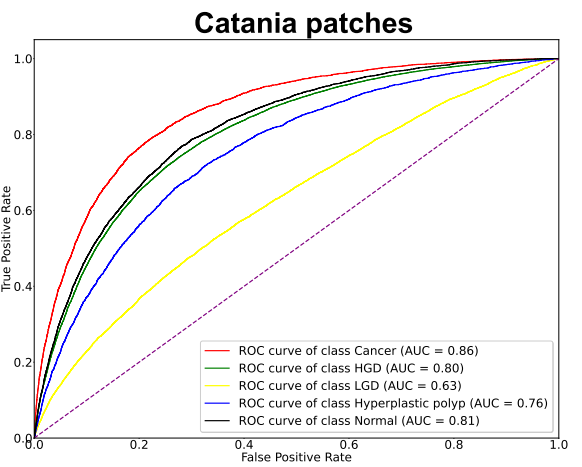
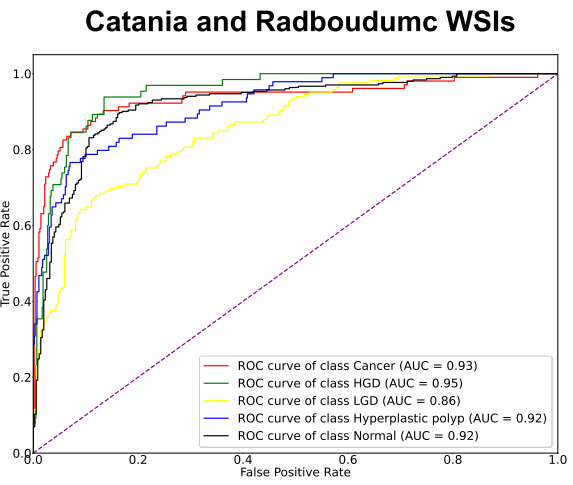
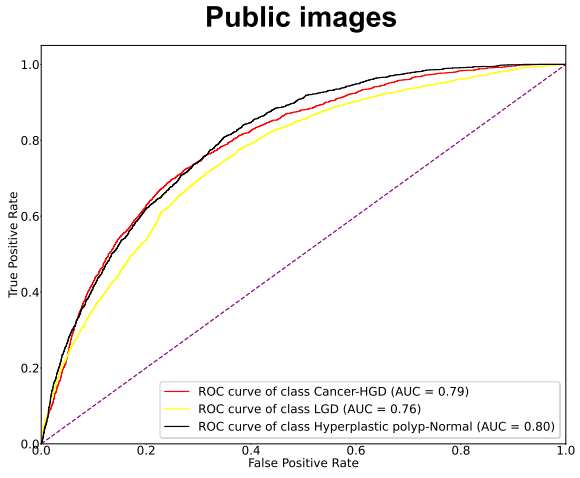
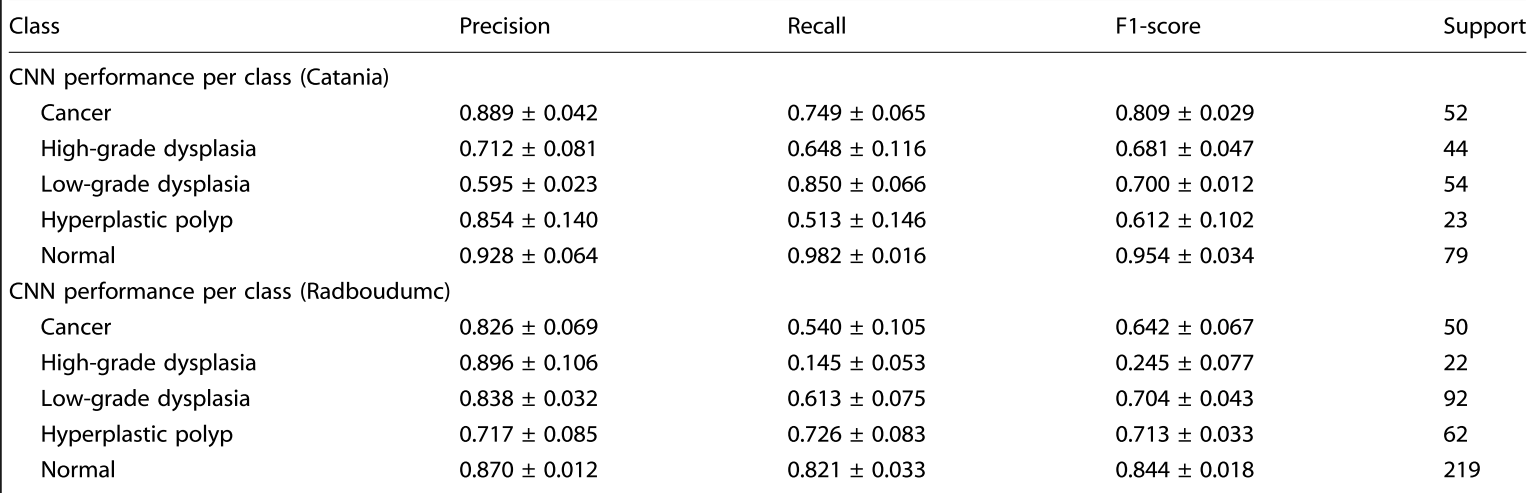
- 성능이 낮게 나온 class에 대해서는 훈련에 들어간 case수가 적거나 유독 WSI에서 작은 영역에 나타나거나 다른 class와 morphology가 비슷해 다른 class로 오인했을 가능성, report에서조차 애매모호한 진단명으로 pathologist도 구분하기 어려운 class라 그랬을 것이라고 추정함
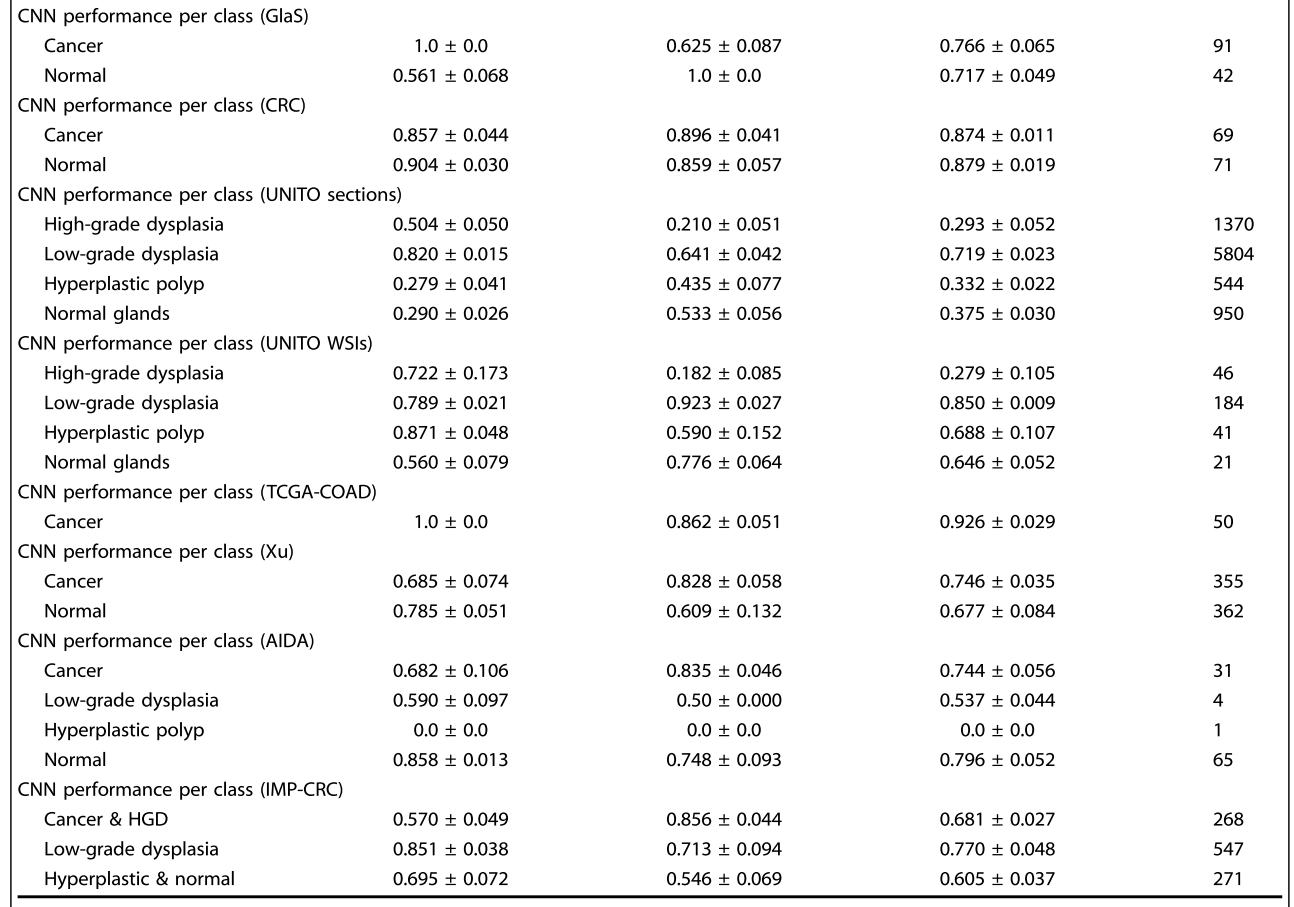

Feature Visualization
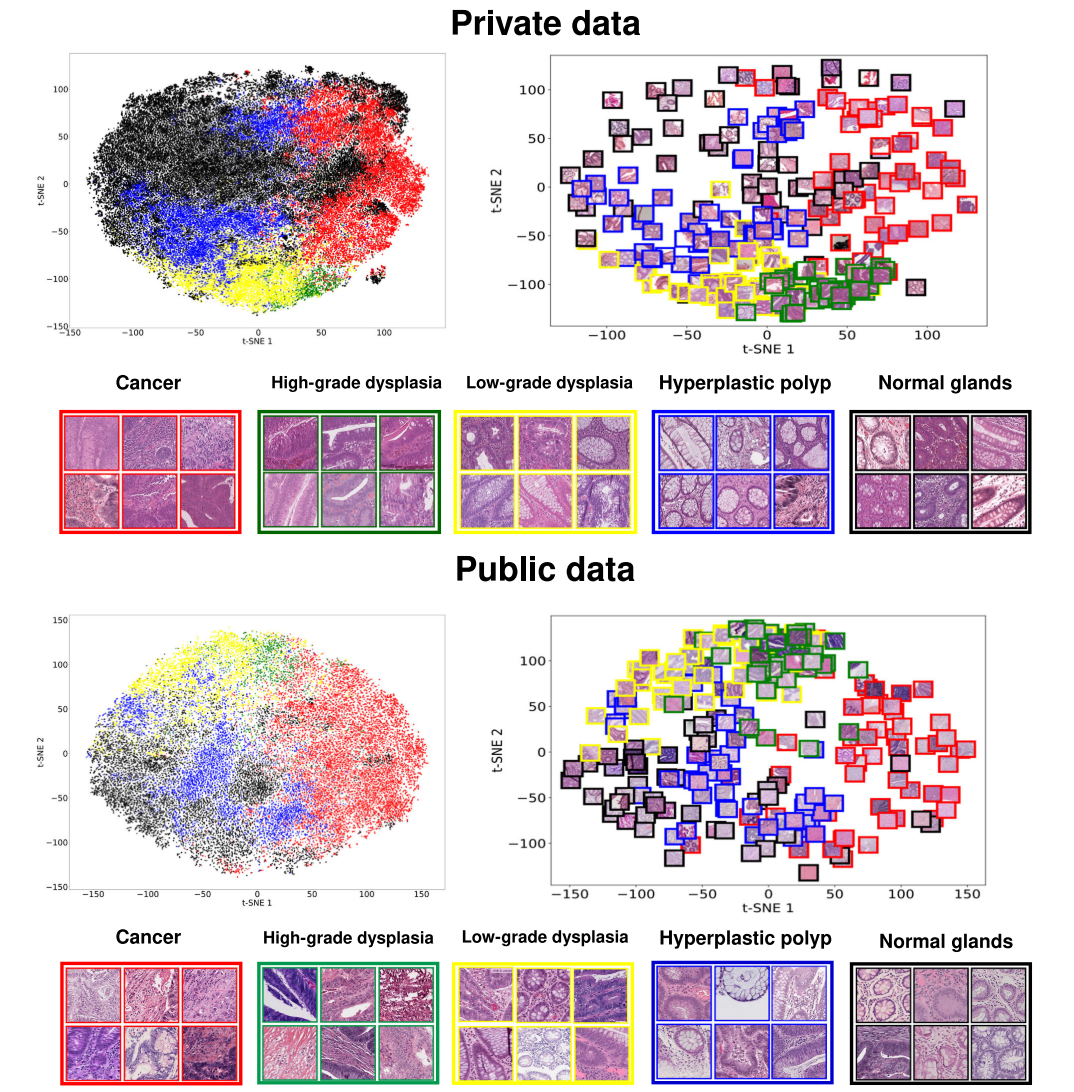
Heatmaps from Attention Network
3. Methods
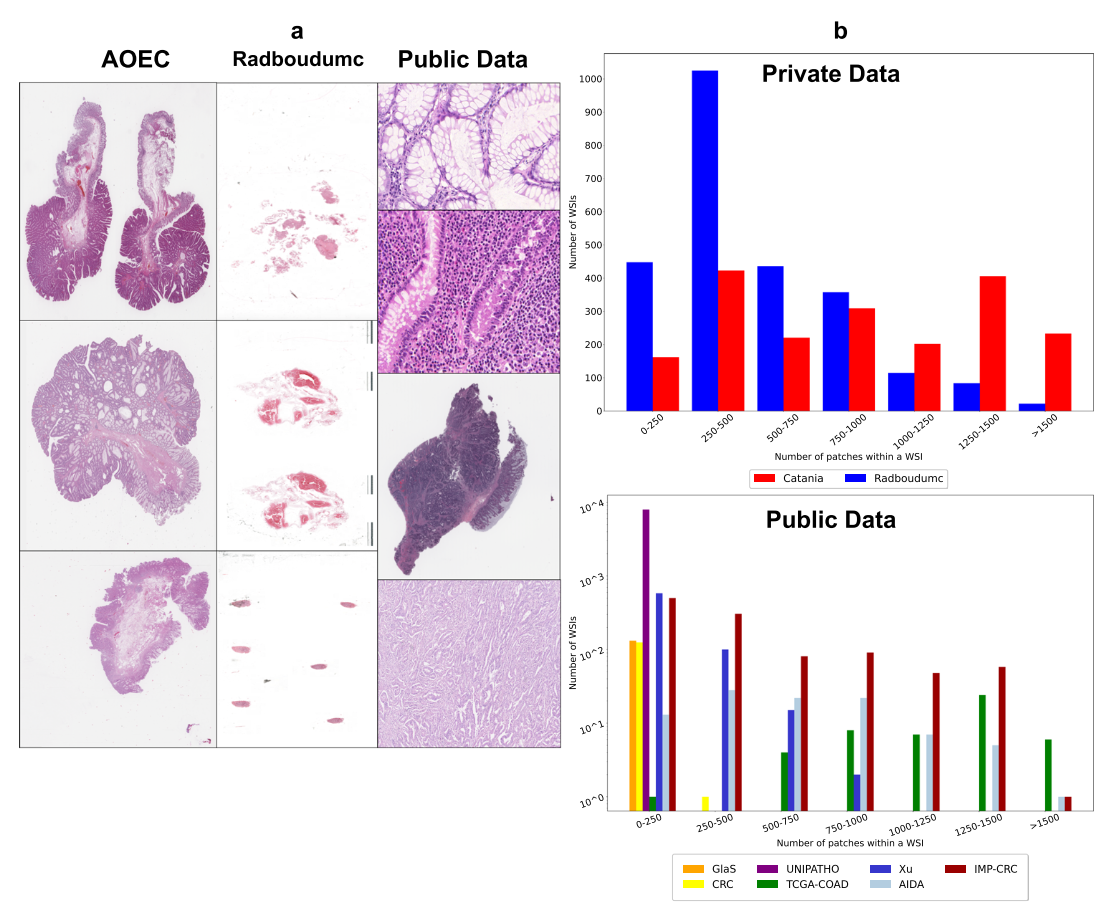
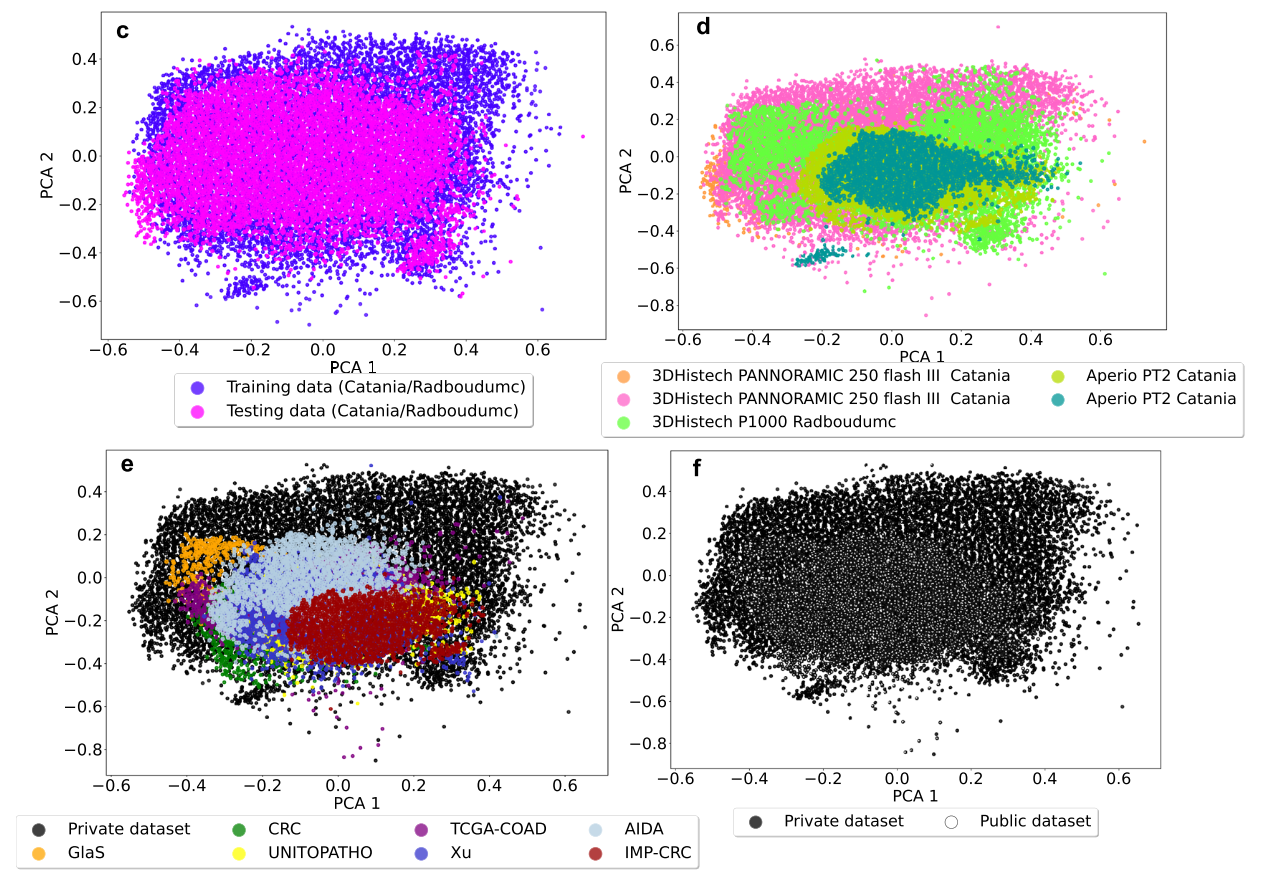
Datasets
-
Data from clinical pathology workflows
-
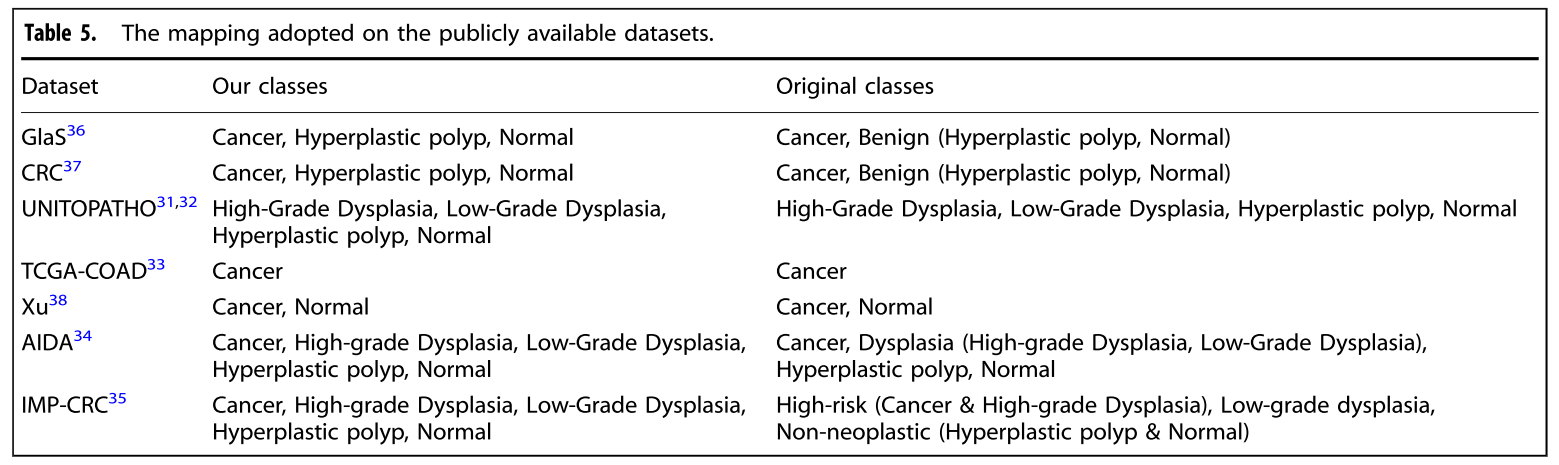
Data from publicly availble datasets

Heterogenity in Datasets
-
Patholgy reports heterogeneity
- Language Catania (Italian), Radboudumc (Dutch)
- Fields: Catania (전체 WSI), Redboudumc (WSI 내의 일부분)
- Input 방법: Catania (의사가 직접 typing), Redboudumc ('speech to text' tool 사용)
-
Images heterogeneity
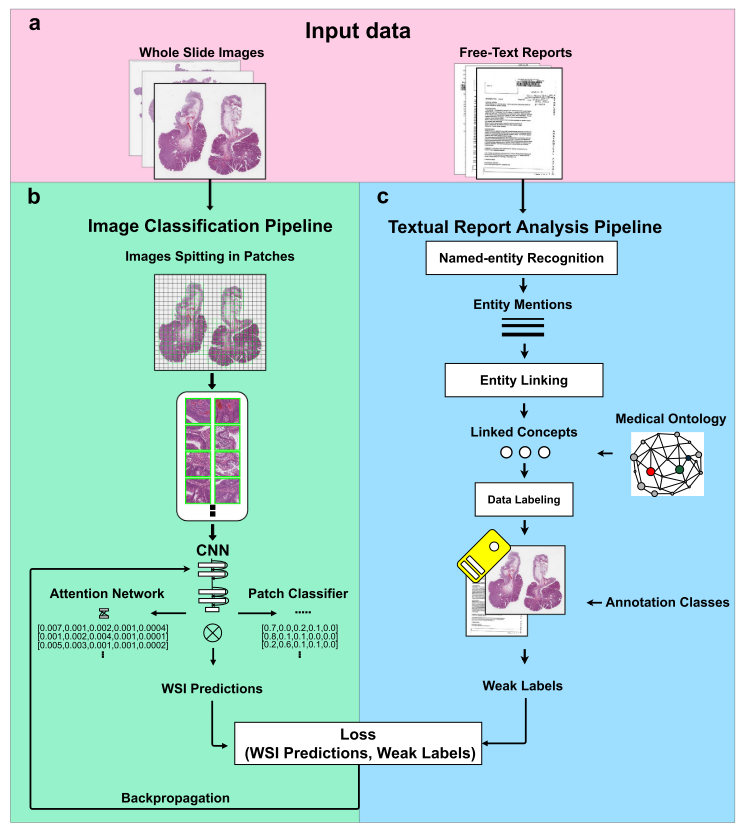
Textual Report Analysis Pipeline
- Diagnostic Reports -> Weak Labels
- Semantic Knowledge Extractor Tool (SKET)
- Named-entity Recognition
- Entity Mentions
- Linked Concepts
- Data Labeling
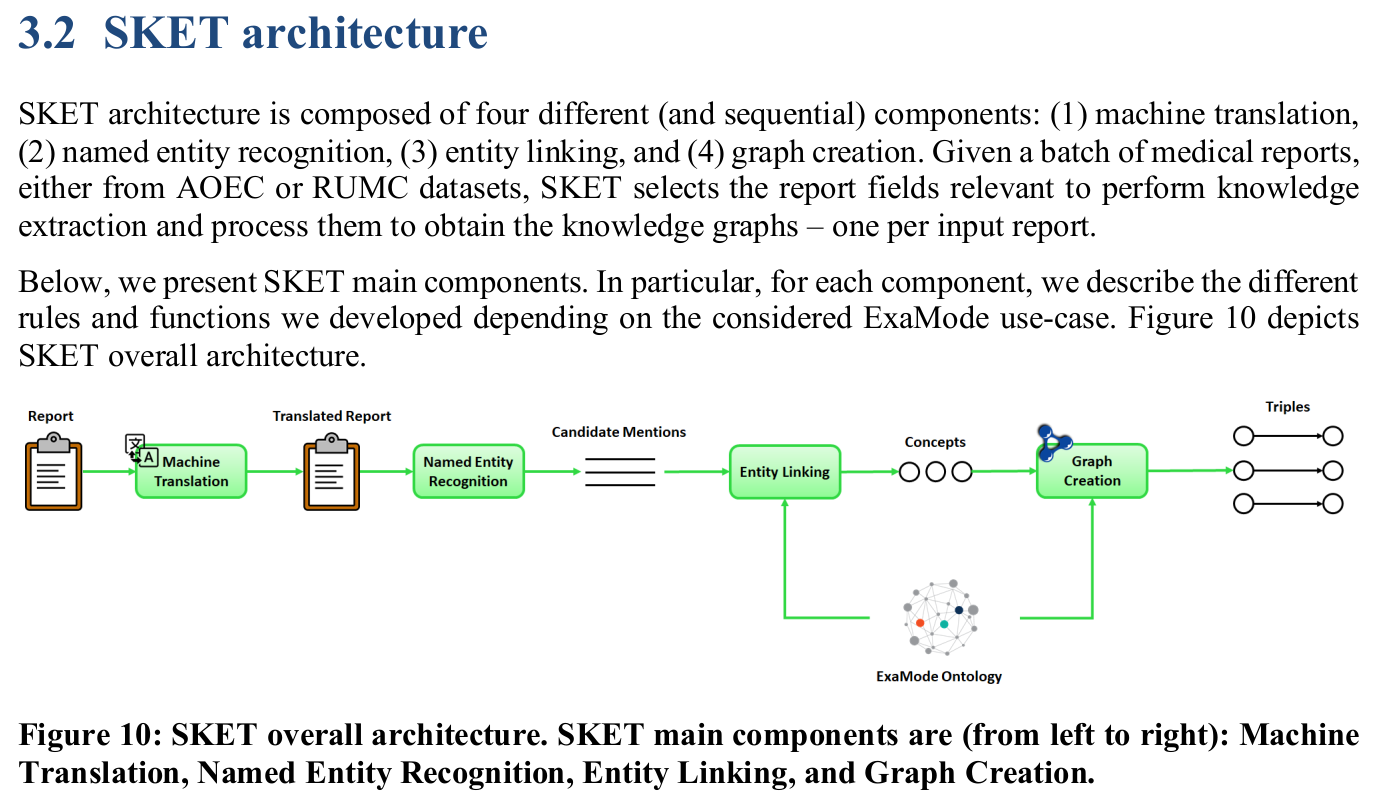

Image Classification Pipeline
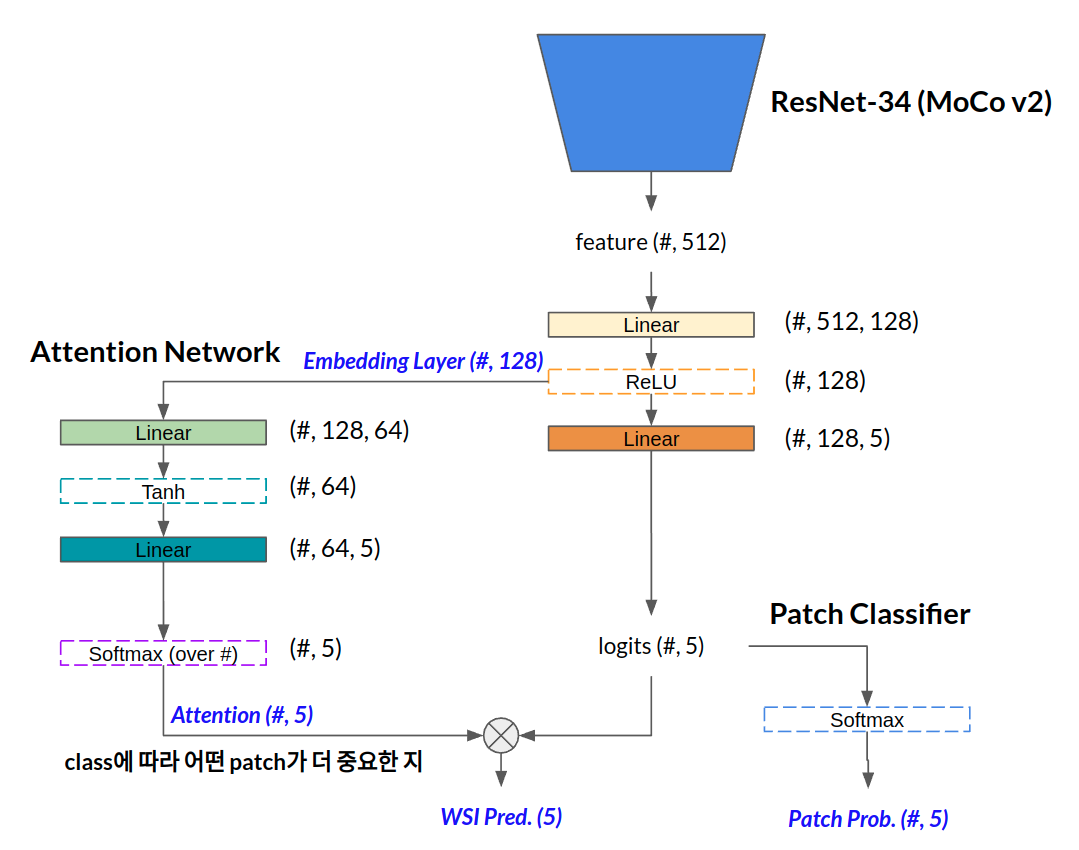
- WSIs and Weak Labels (WSI-level categorization for 5 classes)
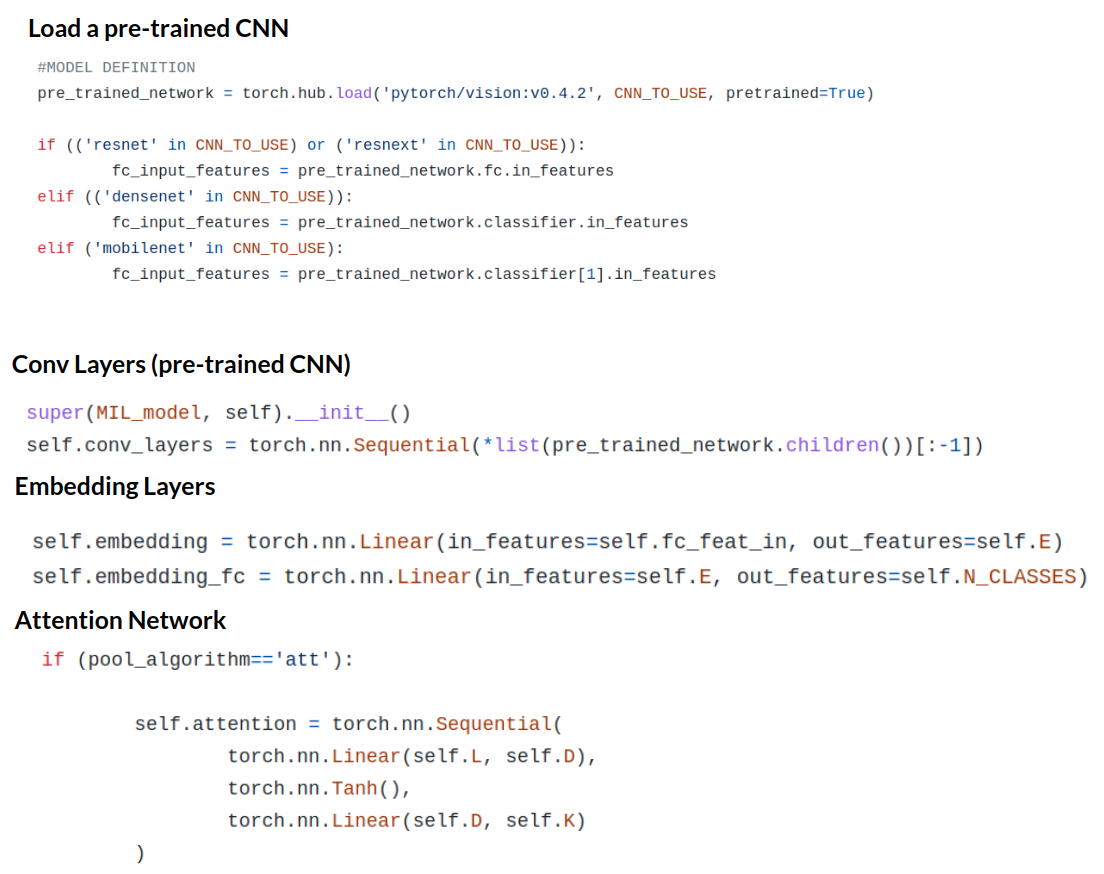
- CNN, Multiple Instance Learning (MIL)
- ResNet34 pre-trained on ImageNet
- patch size: 224 x 224
- feature vectors with 512 elements
- embedding layer, 512 -> 128
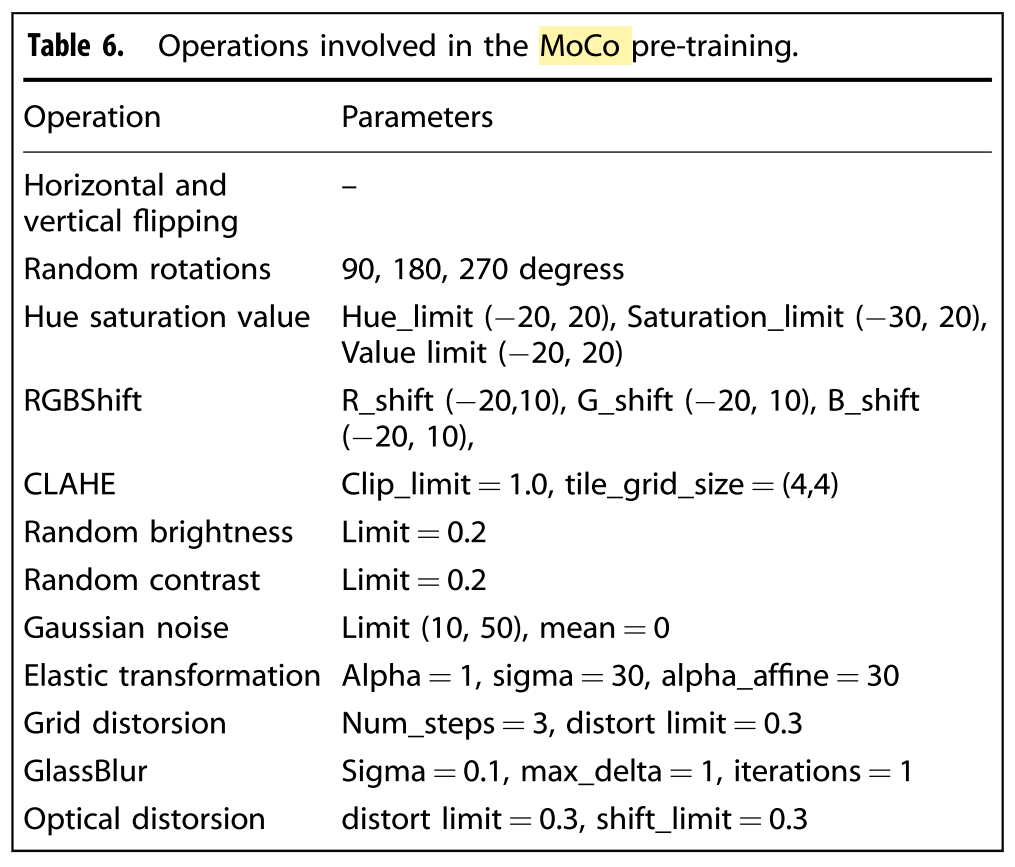
- MoCo v2
4. Conclusion & Discussion
No Human Annotations for WSI classification via NLP & CV
- Digital Pathology에 Annotation cost를 줄이기 위해 MIL를 적용하는 연구가 늘어나고 있는데, 거기엔 여전히 pathologist가 WSI-level categorization해줘야하는 Annotation cost가 남아있었다.
- 이 연구에서는 Dignostic Report를 활용해 그 annotation 마저도 DL algorithm으로 대신하여 MIL을 적용하는 알고리즘을 제안했고 이는 ground truth로 훈련한 모델과 통계적으로 유의미한 차이가 없는 성능을 보였다.
- Report Analysis, Pathology Image Analysis 각 분야에 딥러닝 연구가 따로 진행될텐데, 이를 결합해 Human Annotation 없이 Histopathology Image를 분류할 수 있는 Pipeline을 제안한 것은 굉장히 brilliant하고 동시에 개선할 수 있는 다양한 부분들을 이야기하고 있기 때문에, Diagnostic Report와 Pathology Image를 다 수집하는 쪽에서는 이 연구를 토대로 알고리즘을 발전시킬 수 있을 것으로 보인다.
시각적 정보만 담긴 Histopathology Image와 다른 modal의 정보를 latent vector상에서 결합해 성능을 높이는 차원으로 고도화하는 생각은 해본 적이 있는데, 이 연구는 Report Analysis, Pathology Image Analysis에 쓰이는 딥러닝 알고리즘에 대한 폭넓은 이해를 바탕으로 "Annotation 없이 해버리자"라는 생각을 실험적으로 입증한 느낌이다.
우리는 NLP 안 한다는 생각이 박혀있었는데, 전통하진 못하더라도 여러 분야를 폭넓게 이해하고 있으면 실현가능한 연구의 영역이 넓어진다는 게 느껴진다..
→ 이미 진단보고서를 활용하기 위해 NLP 분석 툴을 보조적으로 사용하지만, pathologist의 도움을 받는 게 기회비용 측면에서 더 낫다고 한다..
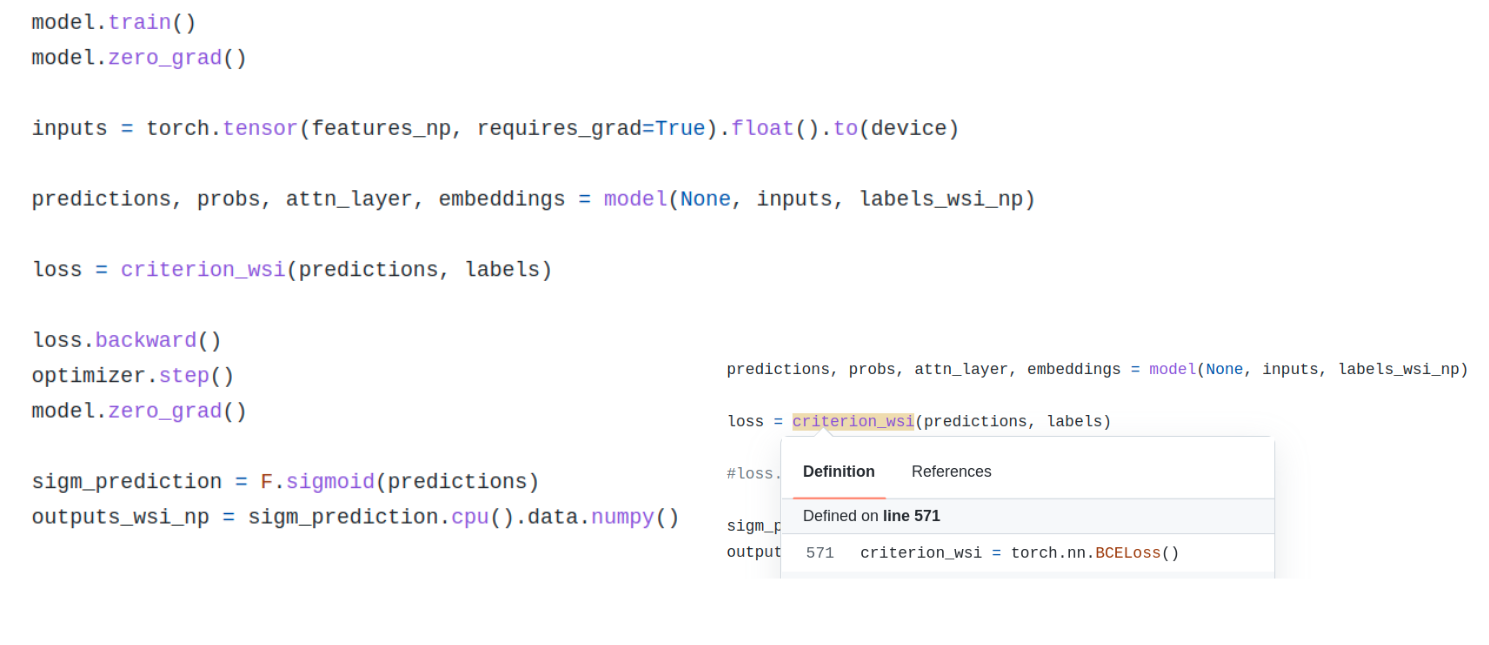
5. MIL detail