https://dacon.io/competitions/official/236382/leaderboard
오랜만에 dacon 대회에 참여하게 되었다!
h&e 사진을 input 으로 받아 3467개의 유전자의 발현정보를 예측하는 대회였는데, metric 때문에 쉽지 않은 대회였다..순위권 안에는 들지 못했지만 기록할 겸 올린다!!

유전자 리스트를 모르는데 성능을 어떻게 높이나요...ㅠㅠ
아무튼 주어진 정보 내에서 최대한 성능을 높이려고 해보았다.
우선 평가 산식을 보면 알 수 있듯이 상관계수가 매우 중요하기 때문에 loss를 mseloss 이외에 상관계수를 반영할 수 있는 loss 를 사용하고자 했다.
-
-pearsoncorrloss
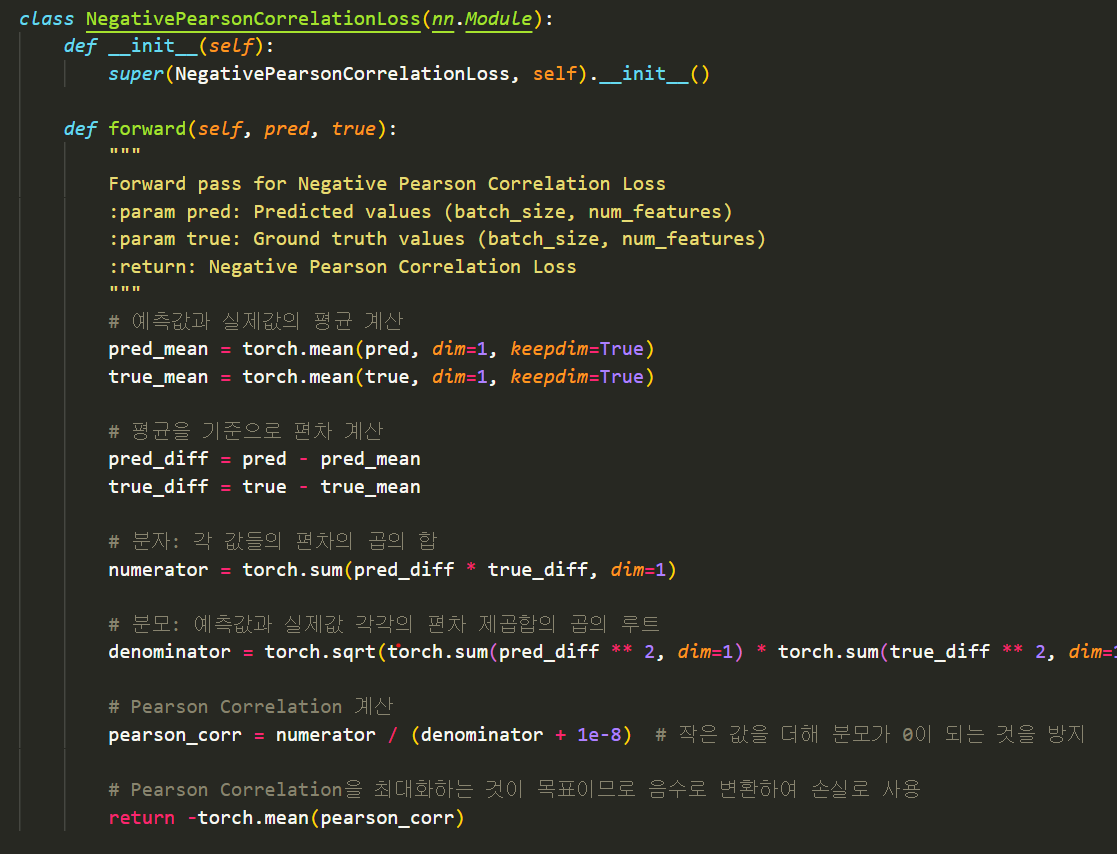
사실 이 코드는 팀원이 짠거긴한데 나는 1-pearsoncorr 가 맞지않나...라고 생각했다...-1~1 범위니까 1에서 빼줘야 loss 범위가 0~2 가 되는거 아닌가?
-
mse+lambda(1-pearson corr)
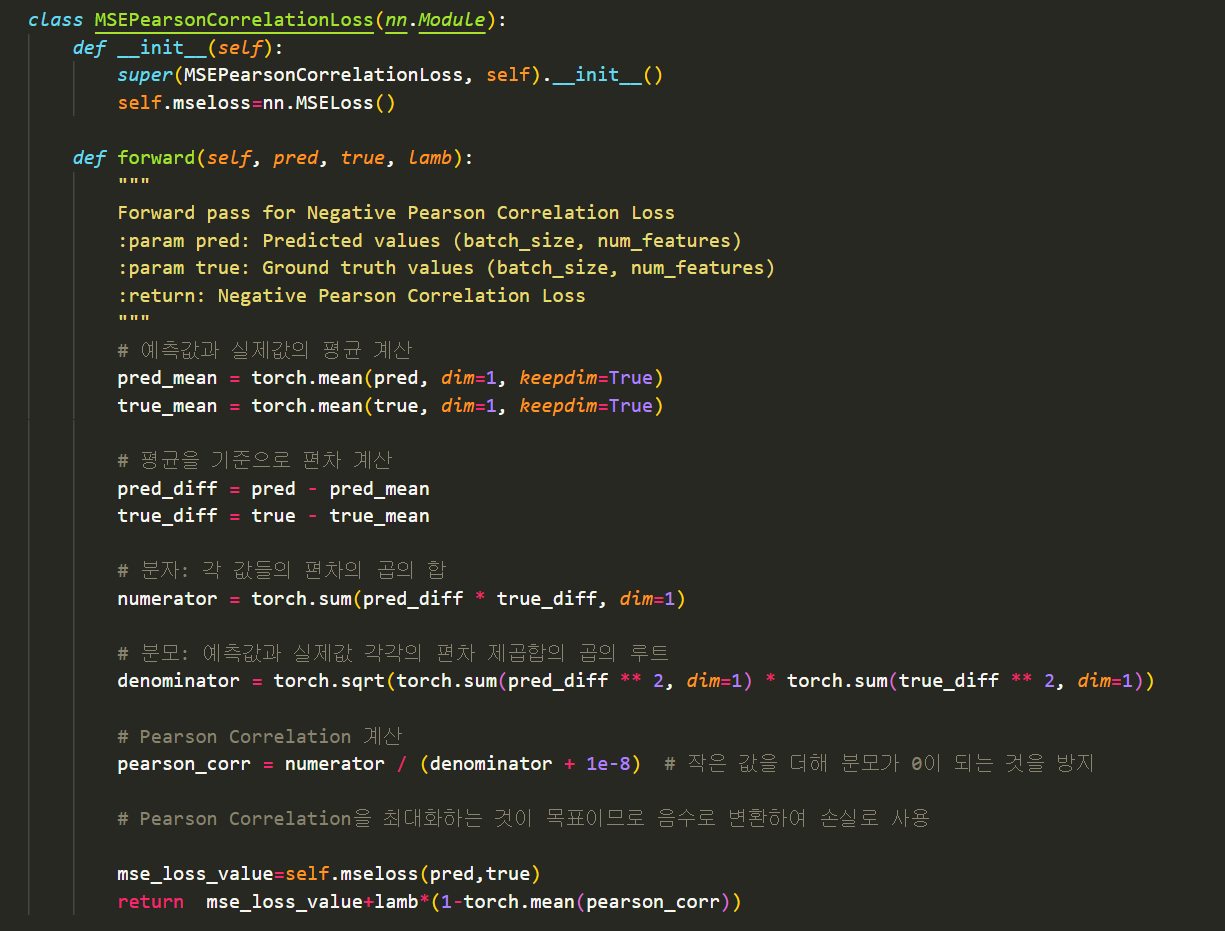
-
1-pearsoncorr loss

등 여러가지 loss 를 시도해봤다.
모델도 resnet50 이 baseline 이였는데,
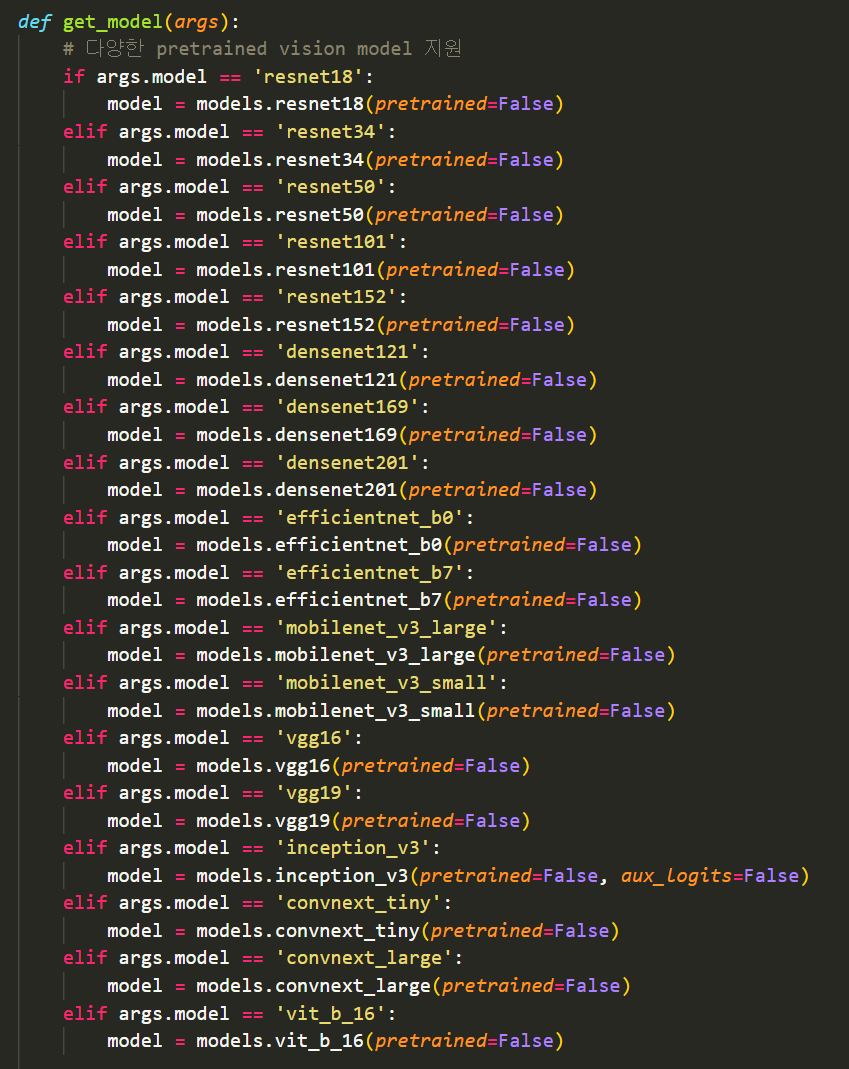
이렇게 모델을 바꿔가며 train 해보기도 했다. resnet 152가 가장 성능이 좋았다.
처음엔 pretrain 된 weight 을 불러와서 freeze 하고 학습시켰다가 이후에는 freeze 를 전혀 하지 않고 학습시켰는데, 이게 제일 성능이 좋았다.
추가적으로 output 인 gene 의 개수가 너무 많기 때문에 마지막 출력 레이어이전에 fc layer 나 mlp layer 를 추가해보기도 했는데, 성능에는 영향이 없었던 것으로 기억한다.
결과적으로는 높은 등수는 아니였지만, 나름 재미가 있었던 것 같다. 특히 이전과 다르게 팀원중에 서버가 있는 친구가 있어서 이것저것 실험해보기 훨씬 좋았다!!(예전에 colab 으로 했을때는 코드 한번 돌릴때가 스트레스였다😡) 중간고사랑 기간이 겹쳐버려서 아쉬움이 많이 남는다. 다음에 좋은 대회가 있으면 또 나가는걸로~!
