01. What is survival data?
- 생존데이터는 사건이 발생할 때까지의 시간 구간을 측정한 데이터이다.
- ex) Time to surgery to death, Time from start of treatment to progression, Time from response to recurrence
- 생존분석은 의학 통계 뿐만 아니라 다양한 영역에서 사용되고 있다. 아래와 같은 이름으로도 불리운다.
- Reliability analysis, Duration analysis, Event history analysis, Time-to-event analysis
02. What is censoring?
- censoring(중도절단)은 다양한 이유로 발생한다. 중도 탈락, 연구 참여 철회, 연구 기간동안 이벤트가 발생하지 않는 경우 등.
- censoring은 생존 분석에서 가장 중요한 개념이다. 크게 두 가지로 나눠서 생각할 수 있다.
- right censoring : 관찰 기간 보다 event 발생 시각이 느린 경우. 주로 censoring은 right censoring을 의미한다. right censoring은 연구 종료, 타겟 이벤트가 아닌 다른 이유로 사망, 중도 탈락 등으로 발생한다.
- left censoring : 관찰 기간보다 event 발생 시각이 빠른 경우.
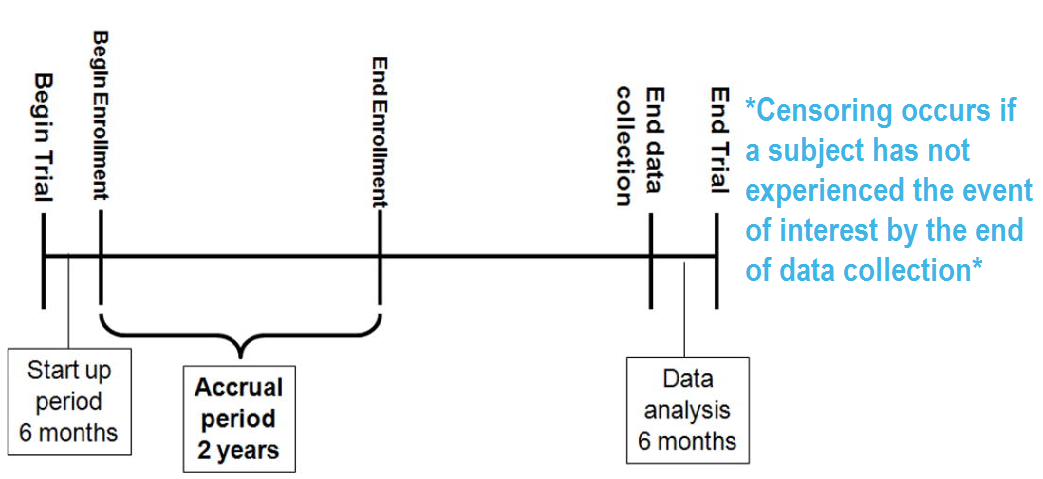
RICH JT, NEELY JG, PANIELLO RC, VOELKER CCJ, NUSSENBAUM B, WANG EW. A PRACTICAL GUIDE TO UNDERSTANDING KAPLAN-MEIER CURVES. Otolaryngology head and neck surgery: official journal of American Academy of Otolaryngology Head and Neck Surgery. 2010;143(3):331-336. doi:10.1016/j.otohns.2010.05.007.
- censoring 데이터 또한 분석에 중요한 재료로 쓰인다. 사용 가능한 특정 시점()을 파악한 후 분석에 활용하는 것이 생존분석이 중요한 이유다.
- 특정 시점()를 파악하기 위해 각 그룹 별 시간 분포도를 확인할 필요 있다.
03. Components of survival data

-
: 사건 발생 시간
-
: censoring (중도절단) 발생 시간
-
Ovserved time = min(, ), 관측시간
-
Event indicator (중도절단여부, censoring indicator)
-> 1 :
-> 0 : -
즉, 데이터는 형태를 띄게 된다. 여기에서 는 = 이면 censoring time, = 이면 survival time을 의미한다.
04. Survival probablily
- : 환자의 생존시간
- : 특정 시간
- = ( ) : 특정 시점t에서 살아 있을 확률.
- = = ( ) : 특정시점 t까지 event가 발생했을 확률.(누적분포함수)
- f() = F() : 특정 시점 에서 event가 발생할 확률. 의 에 대한 미분이다.
- 생존함수는 아래와 같은 성질을 갖고 있다.
- ,
- , 단조감소
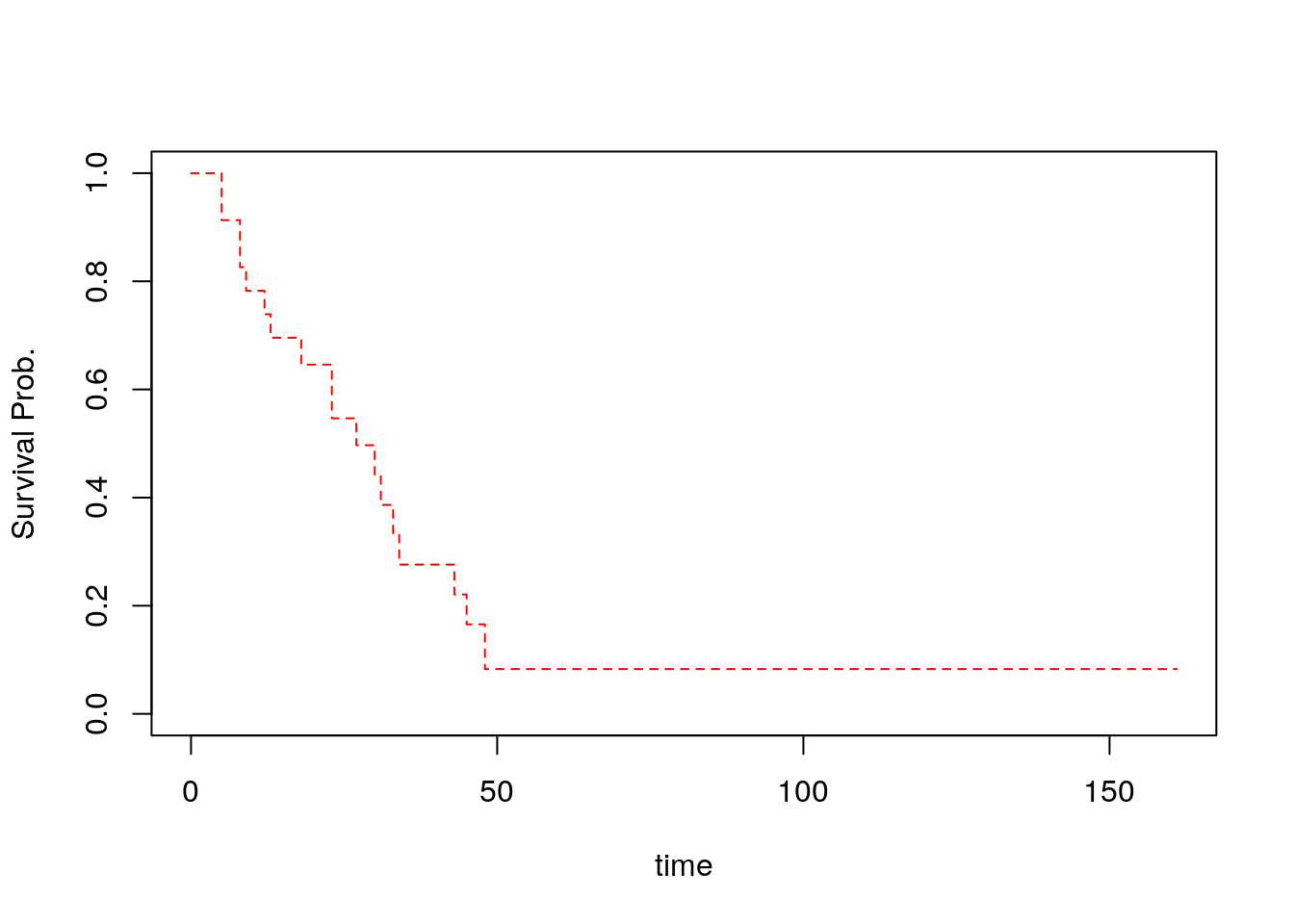
- censoring 데이터로 인해 마지막 시점에서 0이 아닐 수도 있다.
- 위 그림에 따르면, 연구 종료까지 환자 중 10%는 event가 발생하지 않았다.
05. Kaplan-Meier method(누적 한계 추정법)
- censoring이 있는 데이터에서 생존 함수를 추정하는 대표적인 비모수적인 방법.
- 비모수 : 데이터가 정규분포가 아니며 데이터의 표본 수가 적거나 부족하고 데이터가 서로 독립적인 경우.
- 전체 관측시간 데이터를 순서대로 정렬한 후 각 사건의 발생시점의 생존률을 계산하는 방식. 즉,censoring이 있을 때, 그 사람이 t시점까지 살았다는 것을 활용하여 각 시점에서 survival rate을 구하여 계속 곱하면서 생존 함수를 추정한다.

06. The Cox regression model(Cox의 비례위험 회귀모형_준모수적 방법)
-
시간과 사건(Event) 사이의 예측 회귀 모형을 만드는 통계법으로, KM method는 타겟하는 특성 외의 다른 요인들을 통제할 수 없는 것에 반해, Cox 비례위험모형은 다양한 관측치들에 대해 사건 발생에 미치는 영향을 분석하는 다변량 분석법임.
-
Cox 모형의 결과는 HR(Hazard Ratio, 위험비율)로 해석함. 비례 가정이 만족되지 않을 경우 time dependent covariate approach.
-
위험 함수 (Hazard Function) : t 시점까지는 생존했다고 가정하고 바로 직후 사망할 확률.
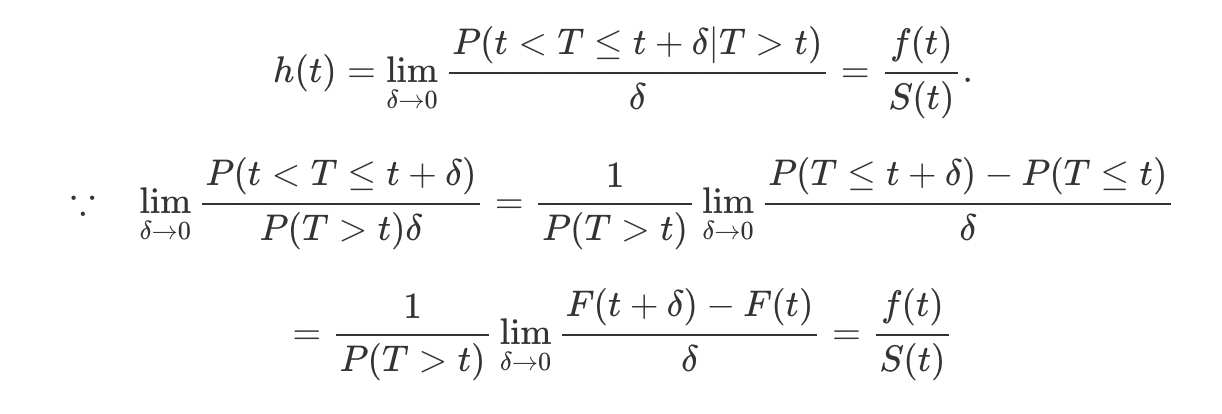
-
생존시간 에 영향을 주는 변수(공변량)들 , , , .... , 가 존재할 때, 위험함수는 다음과 같다.
-
= + ... +
-
= +
-
= baseline hazard, 즉, 모든 들이 0일 때의 위험 함수
-
Hazard Ratio (위험비율) : 특정 시점(t)에서 두 그룹간의 위험 비율을 의미.
-
HR 1 : 사망 위험 증가
-
HR 1 : 사망 위험 감소
-
ex)
-
남자 : t = +
-
여자 : t = +
-
두 로그위험함수식의 차를 구하면 회귀계수()과 일치함.
-
Hazard ratio : = e
07. R 실습
# library
library(survival)
library(survminer)
library(lubridate)
library(tidyverse)# data
date_ex <-
tibble(
sx_date = c("2007-06-22", "2004-02-13", "2010-10-27"),
last_fup_date = c("2017-04-15", "2018-07-04", "2016-10-31")
)
date_ex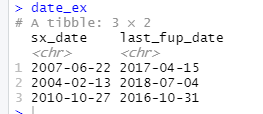
# Formatting dates
# base R
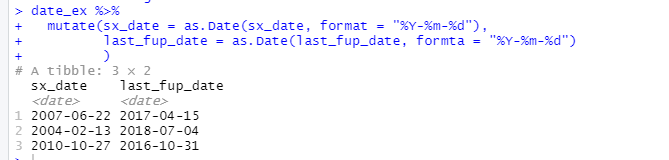
date_ex %>%
mutate(
sx_date = as.Date(sx_date, format = "%Y-%m-%d"),
last_fup_date = as.Date(last_fup_date, format = "%Y-%m-%d")
)
# Lubridate package
date_ex %>%
mutate(
sx_date = ymd(sx_date),
last_fup_date = ymd(last_fup_date)
)- 데이터 형태가 정형화 되어 있으면 lubridate, 데이터 형태가 비정형 상태면 base R 사용!
# Calculating survival times
# base R
date_ex %>%
mutate(
os_yrs =
as.numeric(
difftime(last_fup_date,
sx_date,
units = "days")) / 365.25
)
# lubridate package
date_ex %>%
mutate(
os_yrs =
as.duration(sx_date %--% last_fup_date) / dyears(1)
)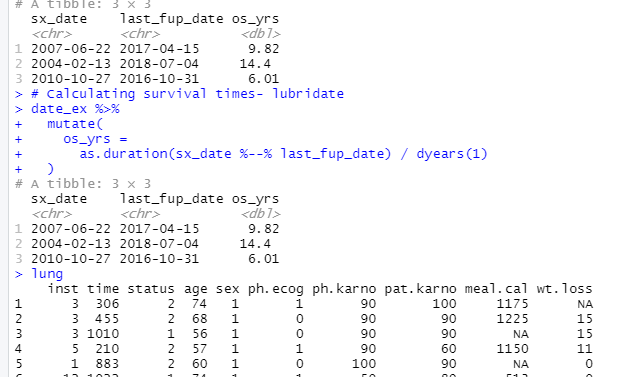
# Creating survival objects
Surv(lung$time, lung$status)[1:10]# Estimating survival curves with the Kaplan-Meier method
f1 <- survfit(Surv(time, status) ~ 1, data = lung)
names(f1)
# Kaplan-Meier plot
# base R
plot(survfit(Surv(time, status) ~ 1, data = lung),
xlab = "Days",
ylab = "Overall survival probability")
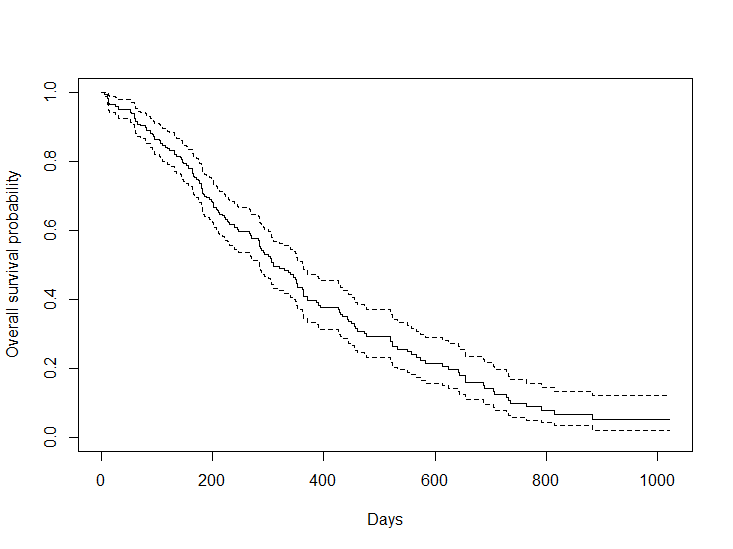
# ggsurvplot
ggsurvplot(
fit = survfit(Surv(time, status) ~ 1, data = lung),
xlab = "Days",
ylab = "Overall survival probability")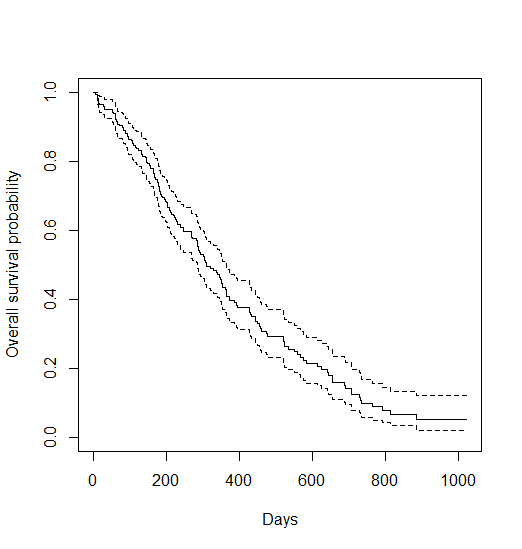
# Estimating x-year survival
summary(survfit(Surv(time, status) ~ 1, data = lung), times = 365.25)# Estimating median survival time
survfit(Surv(time, status) ~ 1, data = lung)# Median survival is often estimated incorrectly
lung %>%
filter(status == 2) %>%
summarize(median_surv = median(time))# Comparing survival times between groups
survdiff(Surv(time, status) ~ sex, data = lung)
# Extracting information from a survdiff object
sd <- survdiff(Surv(time, status) ~ sex, data = lung)
1 - pchisq(sd$chisq, length(sd$n) - 1)+
ezfun::sdp(sd)# The Cox regression model
coxph(Surv(time, status) ~ sex, data = lung)# Formatting Cox regression results
broom::tidy(
coxph(Surv(time, status) ~ sex, data = lung),
exp = TRUE
) %>%
kable()
coxph(Surv(time, status) ~ sex, data = lung) %>%
gtsummary::tbl_regression(exp = TRUE) 