1. 딥러닝과 데이터
데이터의 양도 중요하지만 '필요한' 데이터도 중요하다.
데이터가 머신 러닝과 딥러닝에 효율적으로 사용되게끔 가공되었는지도 중요
데이터 분석에 가장 많이 사용하는 파이썬 라이브러리
판다스 , 맥플롯립
2. 피마 인디언 데이터 분석하기
피마 인디언, 인디언 부족이 비만이 없다가 문물을 받아들이면서 엄청난 비율의 당뇨, 비만으로 고통받음. <-- 유전학적인 이유
딥 러닝을 구동시키기 위해서는 속성과 클래스를 구분해야한다.
모델의 정확도를 향상시키기 위해 데이터를 추가하거나 재가공해야할 가능성
3. 판다스를 활용한 데이터 조사
데이터를 다룰 때는 데이터를 다루기 위해 만들어진 라이브러리를 사용하는것이 좋다.
지금까지는 넘파이 라이브러리를 사용했는데, 넘파이의 기능을 포함하면서도 다양한 포맷의 데이터를 다루게 해 주는 판다스 라이브러리를 사용해서 데이터를 조사해보자.
판다스와 시본
코랩은 기본적으로 제공하지만 주피터를 사용한다면 다음 명령으로 라이브러리를 설치해야함.
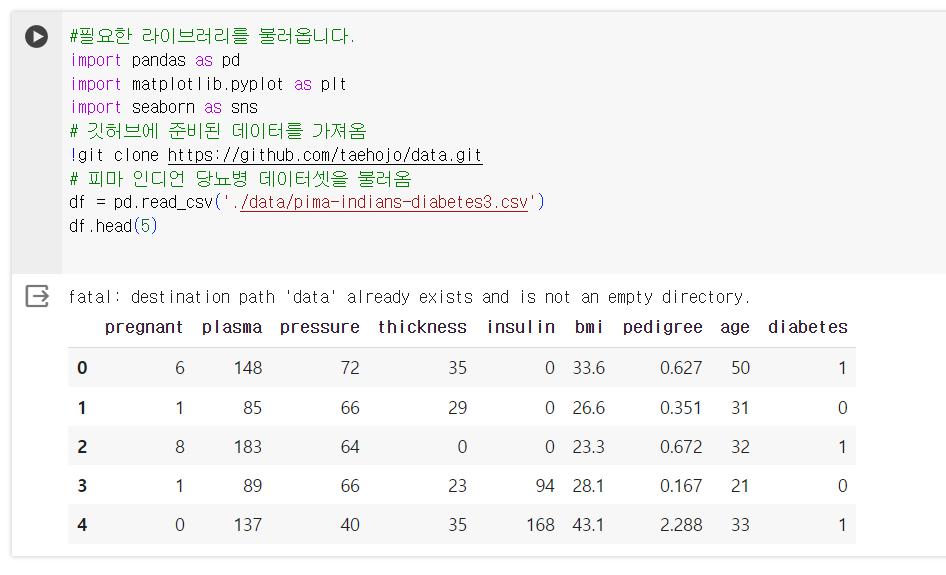
판다스 라이브러리의 read_csv()함수로 csv 파일을 불러와 df라는 이름의 데이터 프레임으로 저장.
csv : comma separated values의 약어
쉼표(,)로 구분된 데이터들의 모음이라는 뜻
csv파일에는 데이터를 설명하는 한 줄이 파일 맨 처음에 나옴.
이를 헤더(header)라고 함
head()함수를 이용해서 df.head(5) 데이터의 첫 5줄을 불러옴
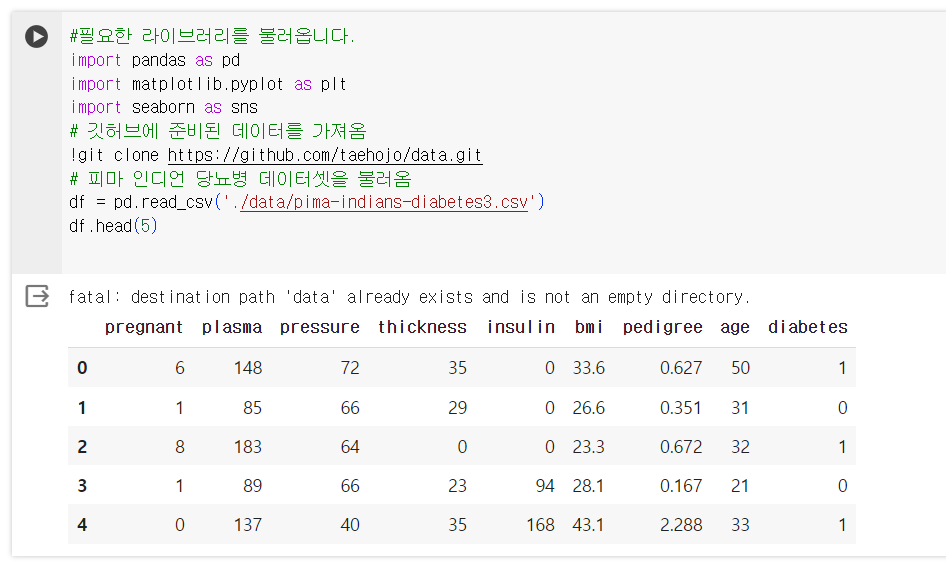
불러온 데이터 프레임의 특정 칼럼을 불러오기 위해서는 df["칼럼명"]
value_counts()함수를 이용하여 각 컬럼의 값이 몇 개씩 있는지 알려줌
df["diabetes"].value_counts()
정보별 특징을 더 알고싶은 경우 describe()함수를 이용
샘플 수, 평균, 표준편차, 최솟값, 백분위 25%, 50%, 75% 그리고 최댓값
df.describe()

a97e-4005-9807-a47d9c83f6b9/image.png)
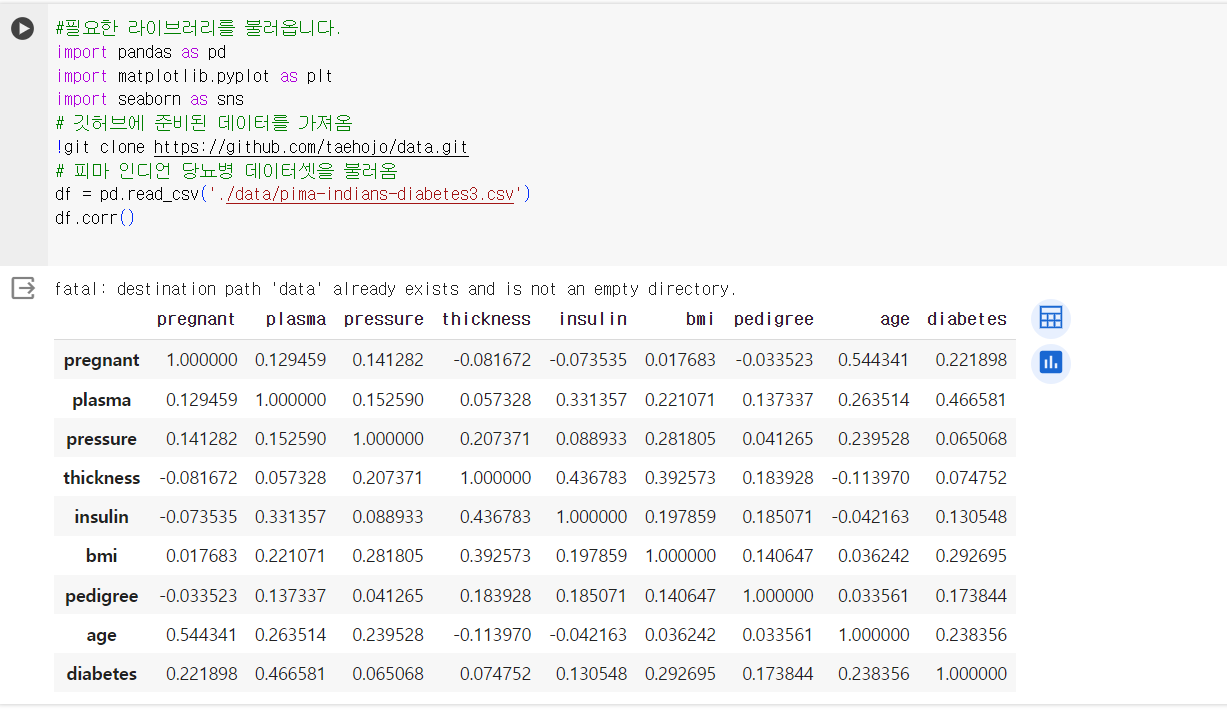
각 항목이 어느 정도의 상관관계를 가지고 있는지 알고 싶으면
df.corr()
맥플롯립은 파이썬에서 그래프를 그릴 때 가장 많이 사용되는 라이브러리
시본라이브러리는 이를 기반으로 더욱 정교하게 그래프를 그리게 해줌
colormap = plt.cm.gist_heat #그래프의 색상 구성을 정함
plt.figure(figsize=(12,12)) #그래프의 크기를 정함
heatmap()함수: 시본 라이브러리 중 항목 간 상관관계를 나타냄
두 항목씩 짝을 지은 후 각각 어떤 패턴으로 변화하는지 관찰하는 함수
두 항목이 전혀 다른 패턴이면 0 비슷하면 1
colormap = plt.cm.gist_heat # 그래프의 색상 구성을 정함.
plt.figure(figsize=(12,12)) # 그래프의 크기를 정함.
sns.heatmap(df.corr(), linewidths=0.1, vmax=0.5, cmap=colormap,
linecolor='white', annot=True)
plt.show()

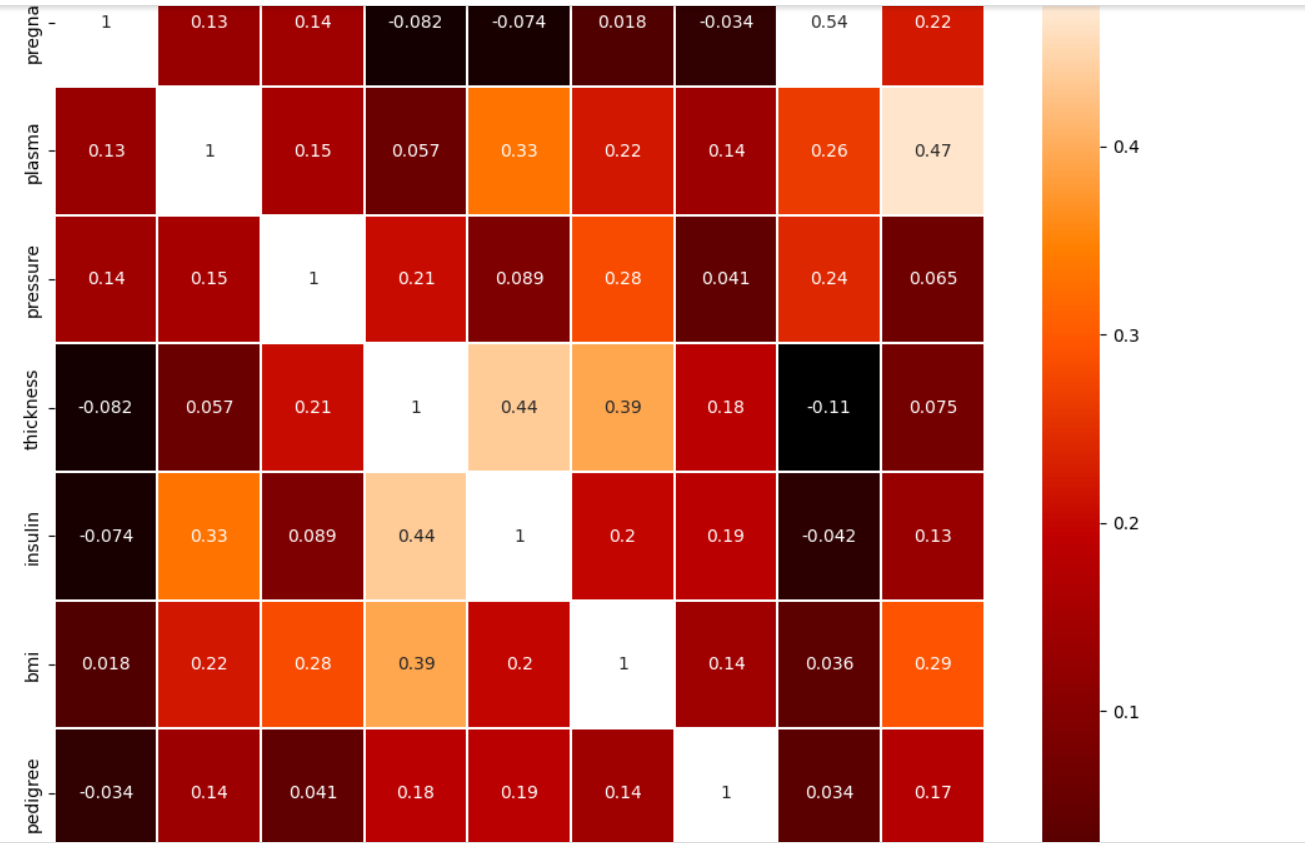
vmax는 색상의 밝기를 조절하는 인자라고 함. cmap은 미리 정해진 맥플롯립 색상의 설정 값을 불러온다.
상관도가 숫자로 표시되어 있고 숫자가 높을수록 밝은 색을 띄는 중
4. 중요한 데이터 추출하기
plasma항목과 BMI항목은 데이터에서 diabetes와 높은 상관관계를 가짐.
두 항목을 중요한 데이터라고 생각하고 다음을 보자.
히스토그램을 그려주는 맥플롯립 라이브러리의 hist() 함수
plt.hist(x=[df.plasma[df.diabetes==0], df.plasma[df.diabetes==1]], bins=30, histtype='barstacked', label=['normal','diabetes'])
plt.legend()
위와 같은 코드는 가져오게 될 칼럼을 hist()함수 안에 x축으로 지정
여기서는 df 안의 plasma 칼럼 중 diabetes 값이 0인 것과 1인 것으로 구분
bins는 x축을 몇개의 막대로 쪼개어 보여 줄 것인지 정하는 변수
barstacked 옵션은 여러 데이터가 쌓여있는 형태의 막대바를 생성하는 옵션
불러온 데이터를 normal과 diabetes로 정함. 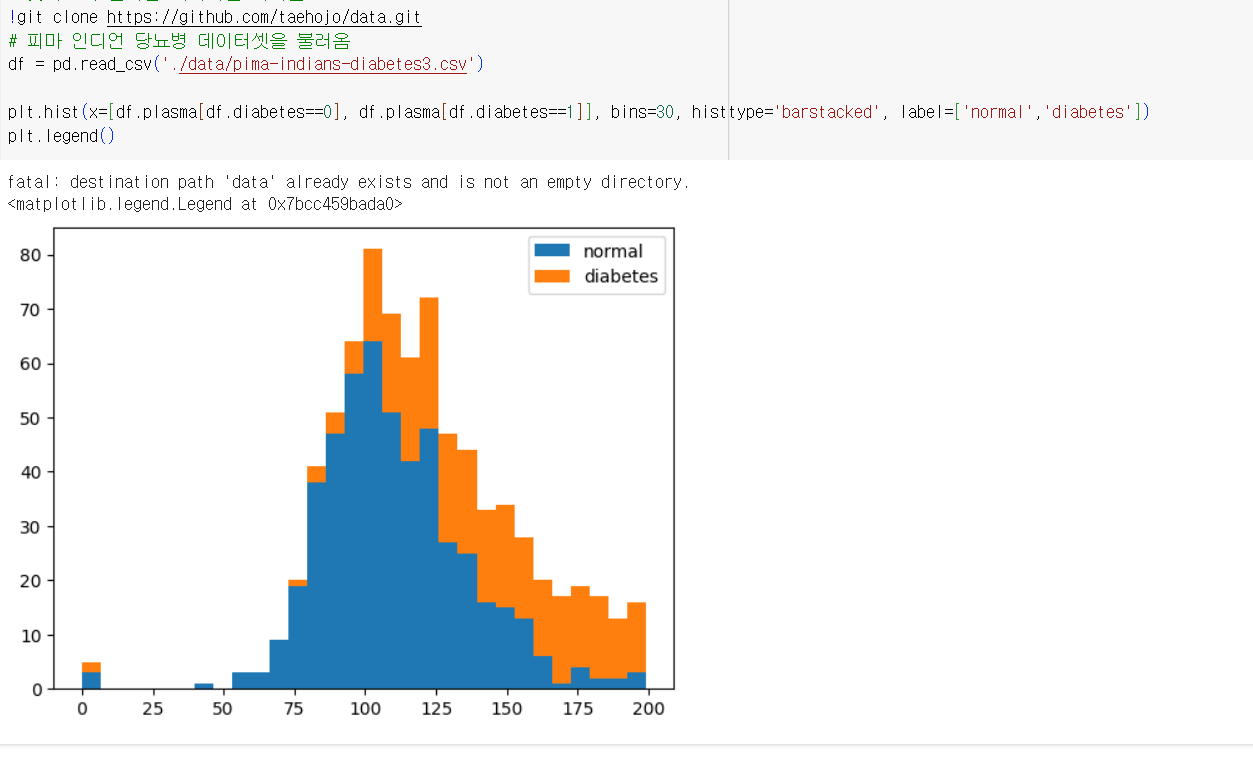
plasma 수치가 높아질수록 당뇨인 경우가 많음을 알 수 있습니다.
BMI를 기준으로 각각 정상과 당뇨가 어느 정도 비율로 분포하는지 보자
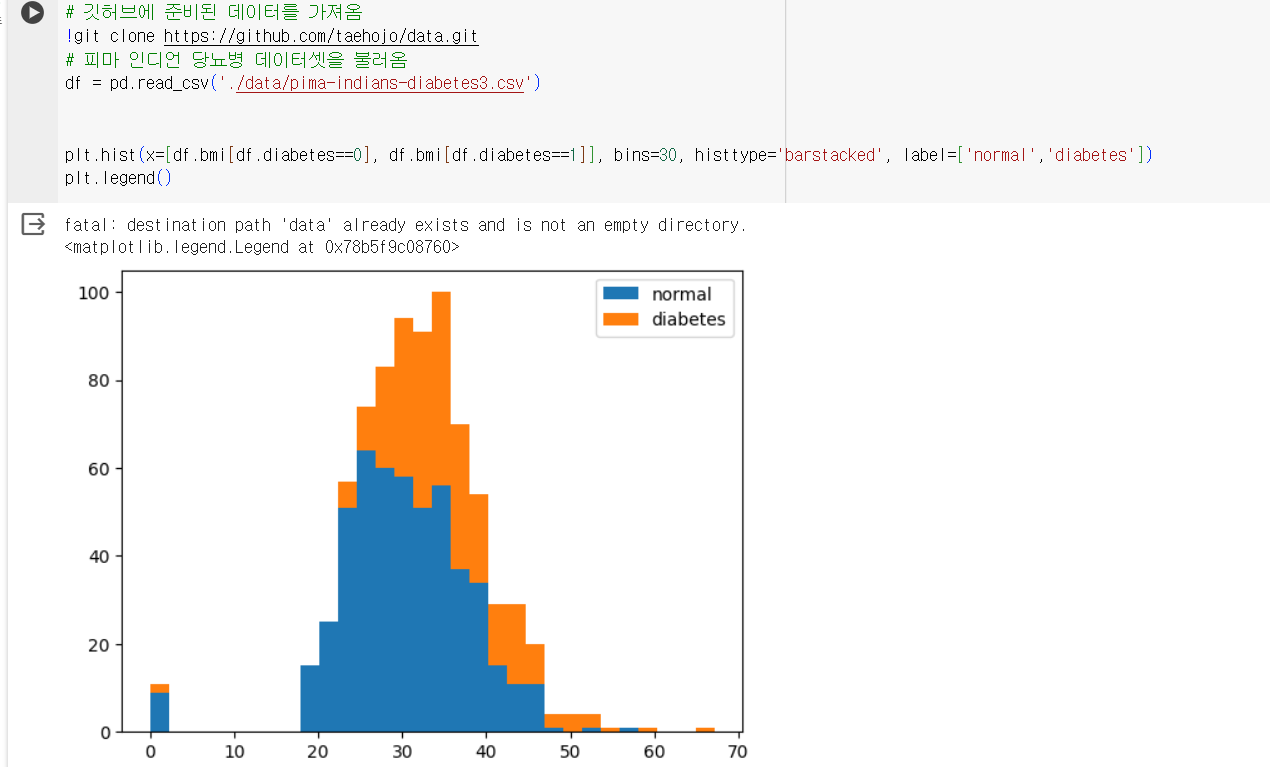
5. 피마 인디언의 당뇨병 예측
텐서플로의 케라스 + 판다스 라이브러리
from tensorflow.keras.models import Sequential
from tensorflow.keras.layers import Dense
#pandas 라이브러리를 불러옵니다.
import pandas as pd
#깃허브에 준비된 데이터를 가져옵니다.
!git clone https://github.com/taehojo/data.git
#피마 인디언 당뇨병 데이터셋을 불러옵니다.
df = pd.read_csv('./data/pima-indians-diabetes3.csv')
X = df.iloc[:,0:8] # 세부 정보를 X로 지정합니다.
y = df.iloc[:,8] # 당뇨병 여부를 y로 지정합니다.
#모델을 설정합니다.
model = Sequential()
model.add(Dense(12, input_dim=8, activation='relu', name='Dense_1'))
model.add(Dense(8, activation='relu', name='Dense_2'))
model.add(Dense(1, activation='sigmoid', name='Dense_3'))
model.summary()
#여기서는 은닉층이 하나 더 추가 됨.
#그리고 층과 층의 연결을 한 눈에 보게해주는 model.summary
#모델을 컴파일합니다.
model.compile(loss='binary_crossentropy', optimizer='adam', metrics=['accuracy'])
#모델을 실행합니다.
history = model.fit(X, y, epochs=100, batch_size=5)
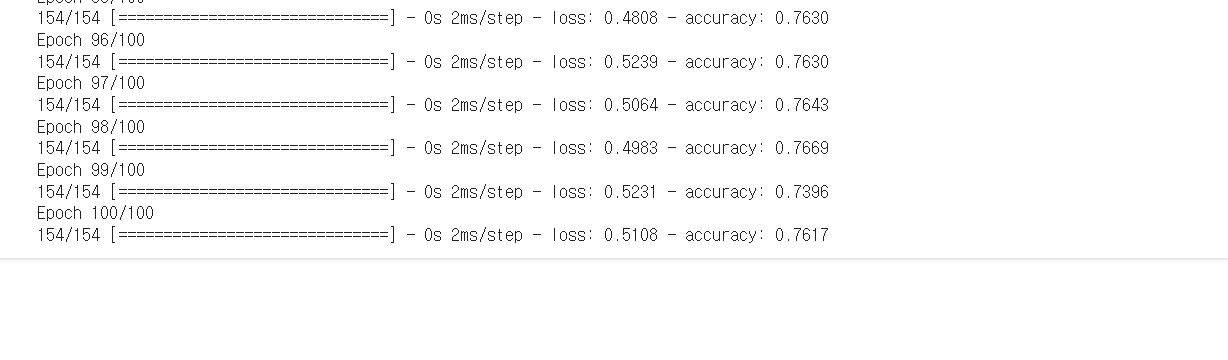
epoch 100에서 0.7617 loss0.5705

- layer 부분은 층의 이름과 유형을 나타냄.
- Output부분은 각 층에 몇 개의 출력이 발생하는지 나타냄
- param 부분은 파라미터 수, 즉 총 가중치와 바이어스의 합
- 부분은 전체 파라미터 합산
