DTI 관련 논문에서 Datasets 부분을 보면
"The benchmark datasets used in this study are the Metz, KIBA, and Davis datasets."
이 항상 등장하는 것을 볼 수 있다. 이게 과연 무엇일까?
Kinase Inhibitor Datasets
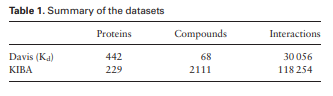
1. Davis
- Dataset : 68개 kinase inhibitors(compound) --- 379개 kinases (Target Protein) [update 0.3.2]
- Task type : Regression
- 결합친화도 점수 : KD / Affinity value : 5.0 ~ 10.8
pip install PyTDC
from tdc.multi_pred import DTI
data = DTI(name = 'DAVIS')
data.convert_to_log(form = 'binding')
split = data.get_split()
split['train']
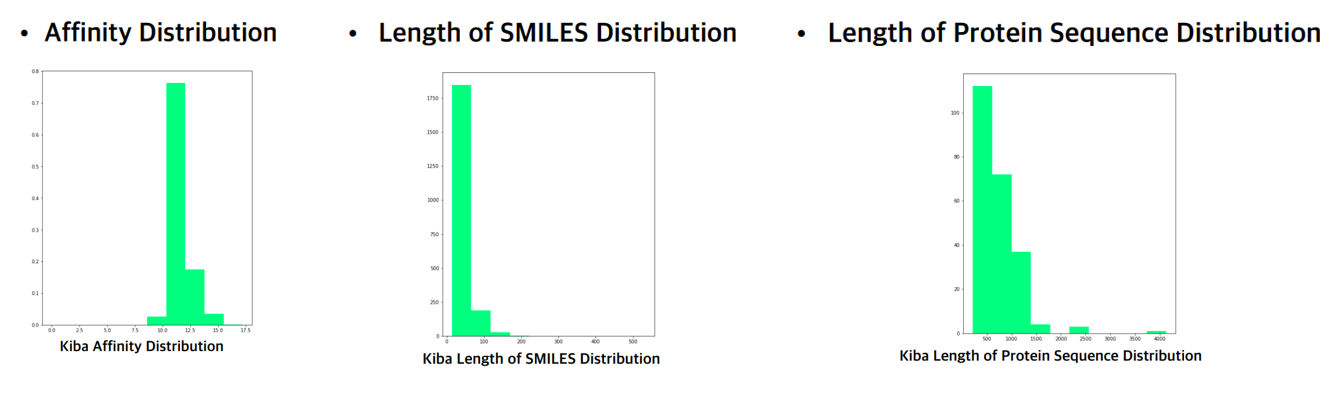
2. KIBA
- Dataset : 2,068개 kinase inhibitors(compound) --- 229개 kinases (Target Protein) [update 0.3.2]
- Task type : Regression
- 결합친화도 점수 : KIBA / Affinity value : 0.0 ~ 17.2
pip install PyTDC
from tdc.multi_pred import DTI
data = DTI(name = 'KIBA')
split = data.get_split()
split['train']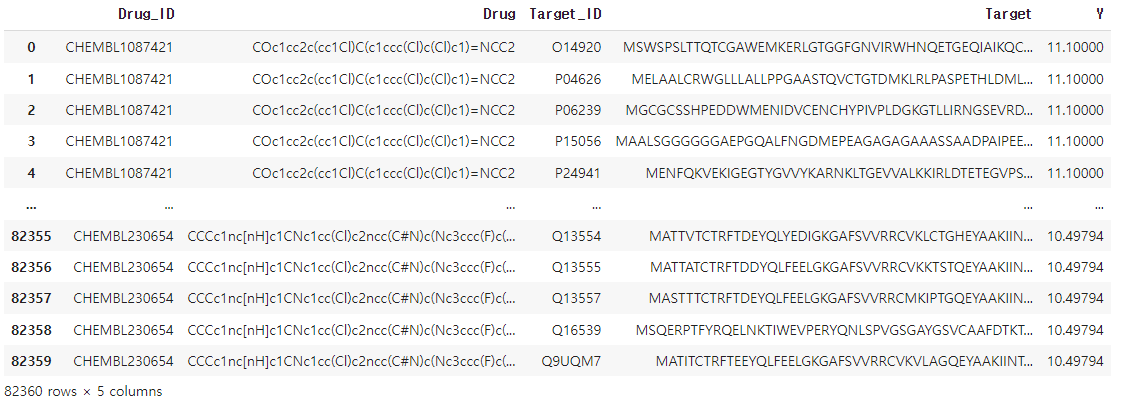
※ Davis & KIBA
같은 Kinase inhibitor dataset 인데 혹시 중복되는 value 가 있지 않을까??
간단한 코드로 중복되는 Drug 와 Target을 찾아보았다.
각각의 Dataset의 train / valid / test 를 하나로 합친 list를 만들고
중복값을 제거해주는 set로 바꾼 뒤 교집합 결과를 확인해보았다.
코드 : https://github.com/bioai96/DTI/blob/main/Davis_%26_KIBA.ipynb
Result
Drug
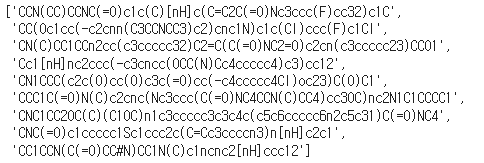
 len(common_Drug) : 9
len(common_Drug) : 9
Target

len(common_Target) : 179
3. Metz

Table S1 Supplementary Excel file containing pKI [-log10(KI)] values for 3858 compounds against 172 protein kinases.
Others
4. BindingDB

Containing ∼20 000 experimentally determined binding affinities of protein–ligand complexes
110 Target Protein <-> ~11,000 Small Molecule Ligand
Data extracted from scientific literature, data collection in Protein Data Bank
5. DUD-E
Database of Useful Decoys
- 22,886 active compounds and their affinities against 102 targets, an average of 224 ligands per target
- 50 decoys for each active having similar physico-chemical properties but dissimilar 2-D topology.

Decoy?
결합을 할 것이라 예측했지만 실제로 결합하지 않는 즉, False Positive 를 말함
이러한 Decoy는 "Docking Algorithms"을 improve하는데 유용함
-> 이것을 Database 화 한 것이 DUD-E