- 정방행렬 => 고유분해를 통해 고윳값, 고유벡터를 찾음
- 정방행렬이 아닌 행렬 => 특이분해를 통해 특잇값, 특이벡터를 찾음
1. 특잇값과 특이벡터
특잇값 분해(Singular value decomposition)
= 특이분해(Singular decomposition)
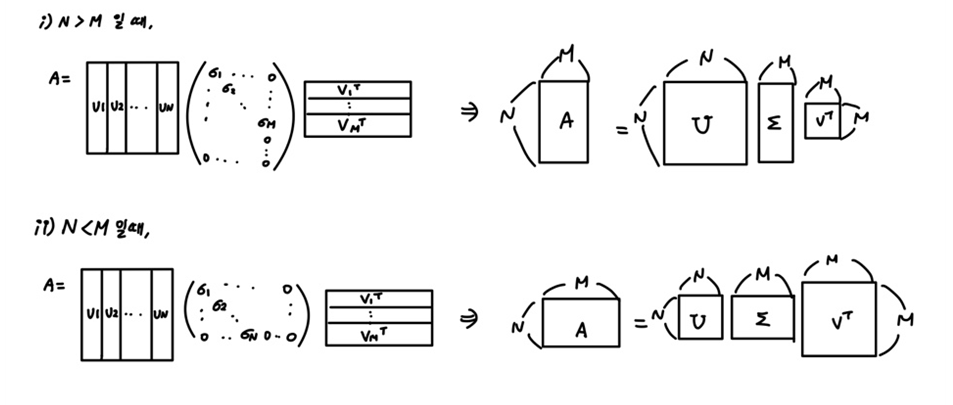
: NxM 크기의 행렬A를 다음과 같이 3개의 행렬 곱으로 나타낸 것
- 특이분해는 모든 행렬에 대해 가능하다

2. 특잇값 분해 행렬의 크기
특잇값 개수는 행렬의 열과 행 중 작은 값과 같다.
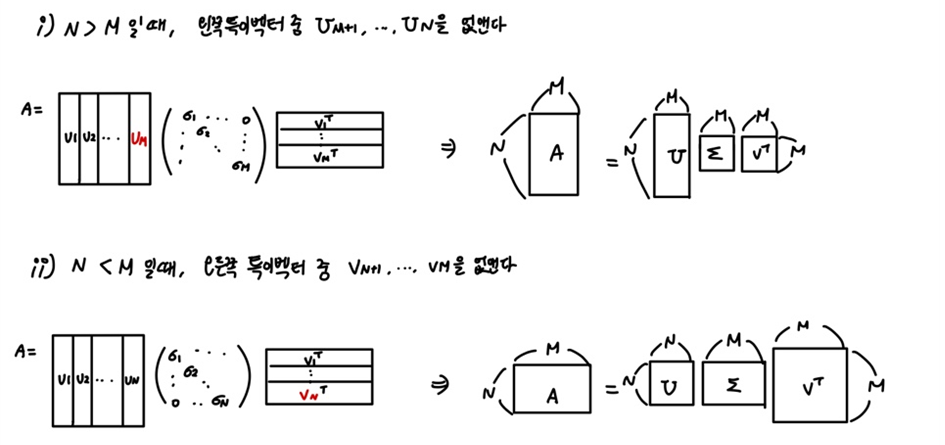
3. 특잇값 분해의 축소형
특잇값 대각행렬에서 0인 부분은 사실상 의미가 없기 때문에, 대각행렬의 0 원소 부분과 이에 대응하는 왼쪽(또는 오른쪽) 특이벡터들을 없애고 축소된 형태로 해도 마찬가지로 원래 행렬이 나온다.
+) 특잇값 분해, 축소형을 python으로 구현
import numpy as np
from numpy.linalg import svd
#특잇값 분해는 svd함수 사용
A = np.array([[3, -1], [1, 3], [1, 1]])
U, S, VT = svd(A)
#U출력
U
#<output>
# array([[-4.08248290e-01, 8.94427191e-01, -1.82574186e-01],
[-8.16496581e-01, -4.47213595e-01, -3.65148372e-01],
[-4.08248290e-01, -2.06937879e-16, 9.12870929e-01]])#S출력
S
#<output>
# array([3.46410162, 3.16227766])#S의 특잇값을 대각성분으로 하는 대각행렬 생성
# 단, 행렬 A가 2X3이었다면 np.diag(np.append(S,0))[:2,:]
np.diag(S, 1)[:, 1:]
#<output>
# array([[3.46410162, 0. ],
# [0. , 3.16227766],
# [0. , 0. ]])#VT출력
VT
#<output>
# array([[-0.70710678, -0.70710678],
# [ 0.70710678, -0.70710678]])# 아래의 결과가 행렬 A와 같음
U @ np.diag(S, 1)[:, 1:] @ VT
# <output>
# array([[ 3., -1.],
# [ 1., 3.],
# [ 1., 1.]])축소형의 경우 인수 full_matrices=False로 지정
U2, S2, VT2 = svd(A, full_matrices=False)
U2 @ np.diag(S2) @ VT2
#<output>
#array([[ 3., -1.],
# [ 1., 3.],
# [ 1., 1.]])4. 특잇값과 특이벡터의 관계
i 번째 특잇값 σi와 특이벡터 ui, vi는
Avi=σiui(i=1,…,min(M,N))
(증명)

5. 특이분해와 고유분해의 관계
- 행렬 A의 특잇값제곱 = 분산행렬 A^TA 의 고윳값
- 행렬 A의 오른쪽 특이벡터 = 분산행렬 A^TA 의 고유벡터
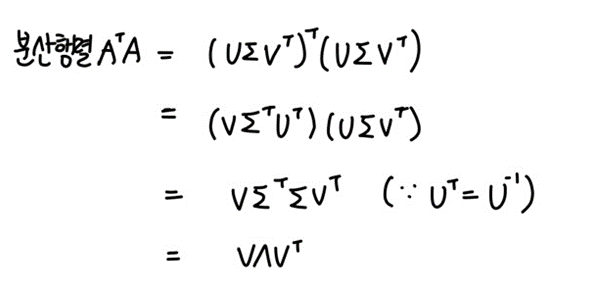
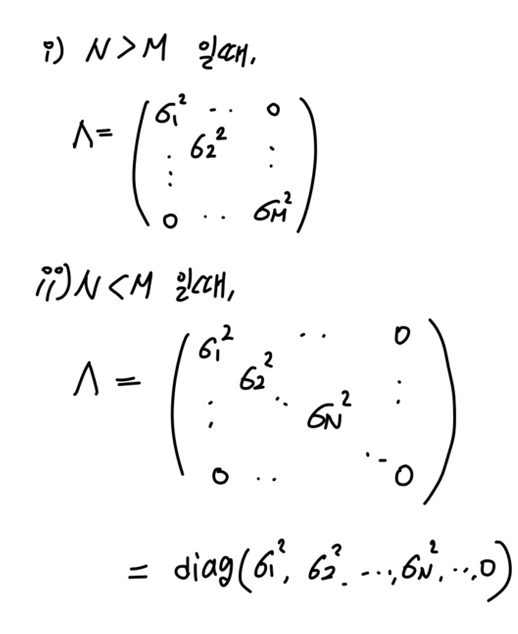
위의 링크를 참고하여 학습했습니다:)