Passage Retrieval and Similarity Search
MIPS (Maximum Inner Product Search)
-
주어진 질문 벡터 q에 대해 Passage 벡터 v들 중 가장 질문과 관련 있는 벡터를 찾아야 함
- 여기서 관련성은 내적(inner product)값이 가장 큰 것
-
-
Challenges
-
실제로 검색해야 할 데이터는 훨씬 방대함
-
더 이상 모든 문서 임베딩을 일일이 보면서 검색할 수 없음
-
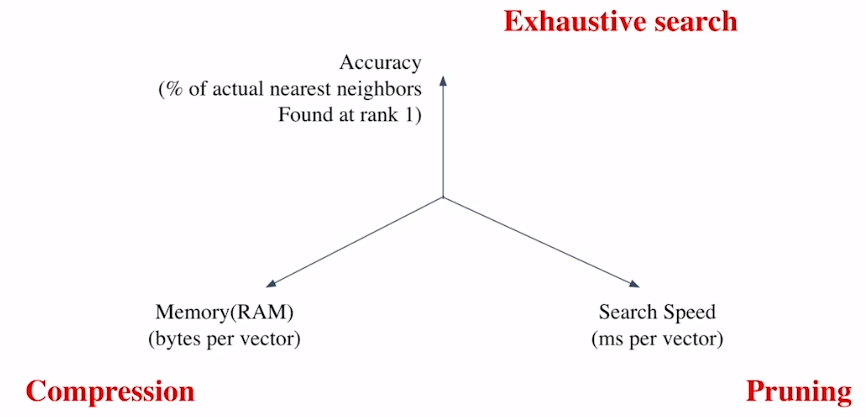
Trade-offs of Similarity Search
-
Search Speed
- 가지고 있는 벡터가 클 수록 더 오래 걸림
-
Memory Usage
-
RAM에 모두 올려둘 수 있으면 빠르지만, 많은 리소스가 필요
-
디스크에서 계속 불러온다면 속도가 느림
-
-
Accuracy
- 속도를 증가시키려면 정확도를 희생해야 하는 경우가 많음
-
속도(search time)와 재현율(Recall)의 관계
-
더 정확한 검색을 하려면 더 오랜 시간이 소모 됨
-
corpus 크기가 커질 수록,
-
탐색 공간이 커지고 검색이 어려움
-
저장해 둘 memory space가 많이 요구
-
Sparse Embedding의 경우 이러한 문제가 훨씬 심함
-
-
Approximating Similarity Search
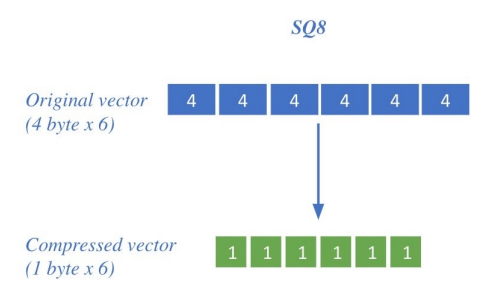
Compression - Scalar Quantization (SQ)
-
vector를 압축해 하나의 vector가 적은 용량을 차지
- 압축량↑ → 메모리↓, 정보 손실↑
-
e.g., Scalar Quantization: 4-byte floating point ⇒ 1-byte (8bit) unsigned integer로 압축
Pruning - Inverted File (IVF)
-
Search Space를 줄여 search 속도 개선
-
Clustering + Inverted File을 활용한 search
-
Clustering
- 전체 vector space를 k개로 cluster로 나눔 (k-means clustering)
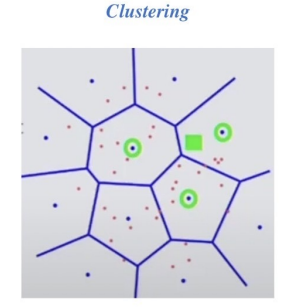
- 초록색 네모가 query vector일 때, 가장 근접한 3개의 cluster만 방문
- 전체 vector space를 k개로 cluster로 나눔 (k-means clustering)
-
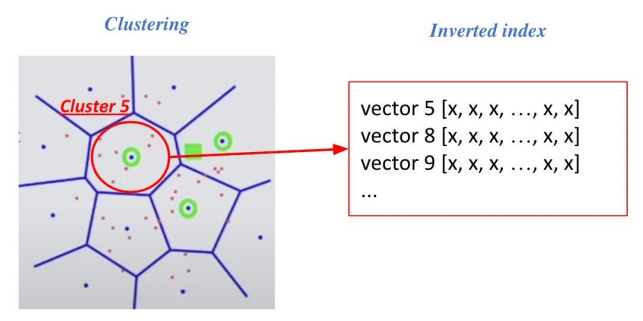
Inverted File
-
vector의 index = inverted list structure
-
각 cluster의 centroid id와 해당 cluster의 vector들이 연결되어있는 형태
-
각 vector마다 연결되어 있는 centroid들이 list 형식으로
-
-
Searching with clustering and IVF
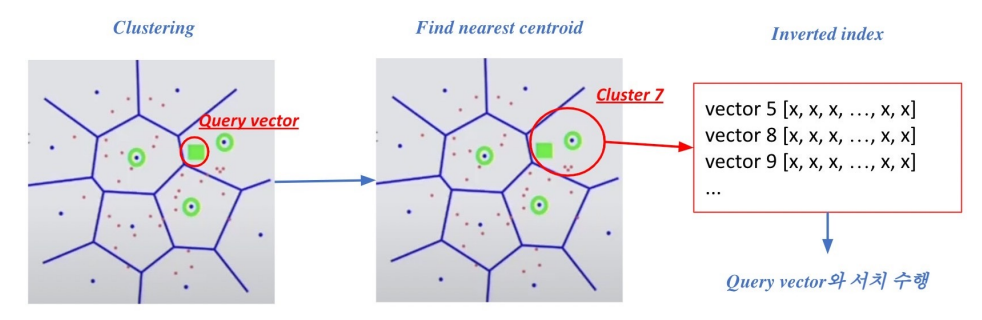
1) 주어진 query vector에 대해 근접한 centroid vector를 찾음
2) 찾은 cluster의 inverted list 내 vector들에 대해 search 수행
Introduction to FAISS
FAISS
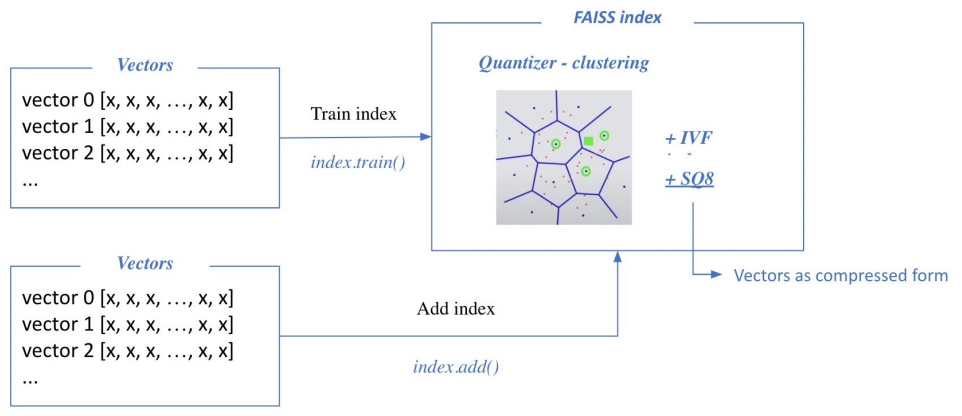
1) Train index and map vectors
-
적절한 cluster 지정을 위해 학습이 필요
-
Scalar Quantization을 위한 최대, 최솟값 계산
-
cluster와 SQ8이 학습이 마치고 나면 (Train Index), 그 후 SQ8 형태로 Add Index
-
add 데이터가 너무 많아 전체 학습이 비효율적일 때, 일부를 sample로 train에 사용
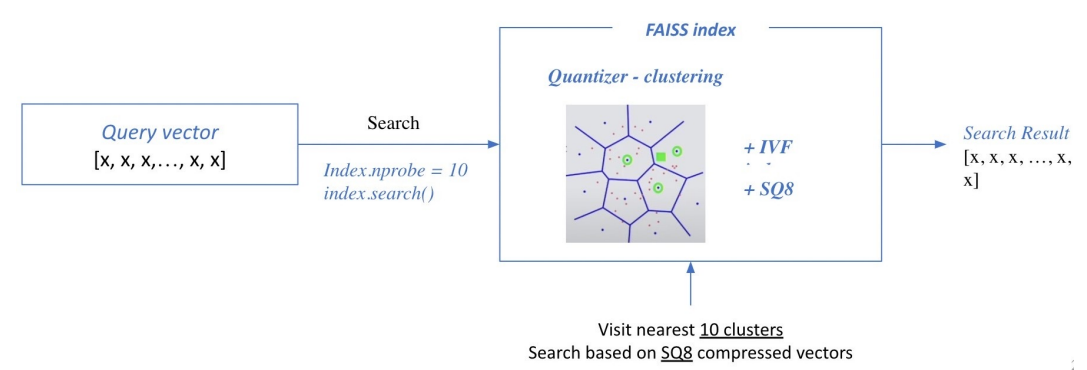
2) Search based on FAISS index
※ nprobe: 몇 개의 가장 가까운 cluster를 방문해 search 할 것인지
추가 학습
당최 이해가 되지 않아 전체적으로 재정리
용어 정리
Scalar Quantization 8
-
벡터의 최솟값과 최댓값을 사용해 분할 경계를 결정하고, 8개 구간으로 분할
-
e.g., 특정 차원 값이 분할 경계 [0, 1/8, 1/4, 1/2, … , 1]에 속한다면,
해당 차원은 0부터 7까지 정수 값 중 하나로 양자화양자화된 각 차원의 값들을 결합해 양자화된 벡터 생성
→ 원본 벡터가 8개의 discrete 값을 갖는 SQ8 quantizaed vector로 표현됨
centroid vector (중심 벡터)
-
clustering 과정에서 각 군집의 중심을 나타내는 벡터
-
해당 군집에 속하는 모든 벡터의 평균을 계산하여 얻어짐
Inverted File Index (역파일 색인)
-
군집에서 중심 벡터와 해당 군집의 벡터들이 역파일 색인으로 연결되어 있음
-
즉 중심 벡터를 찾으면, 그와 연결 된 해당 군집의 다른 벡터들과 search 가능
양자화와 중심 벡터
- 중심 벡터는 양자화 과정에서 사용되며 원본 벡터를 가장 가까운 중심 벡터로 매핑
-
벡터 공간을 여러 군집으로 분할
- 각 군집은 해당 군집에 가장 가까운 중심 벡터로 할당
-
양자화 단계: 각 벡터는 해당 벡터와 가장 가까운 중심 벡터로 매핑
- 일반적으로 유클리디안 거리를 측정
-
매핑된 인덱스 저장: 양자화된 벡터는 중심 벡터와의 거리를 나타내는 인덱스로 변환
- 이 인덱스는 양자화된 벡터를 나타내는 식별자 역할을 수행
→ 양자화는 군집화 과정을 통해 가장 가까운 군집의 중심 벡터로 표현이 변경되는 과정
- 이 인덱스는 양자화된 벡터를 나타내는 식별자 역할을 수행
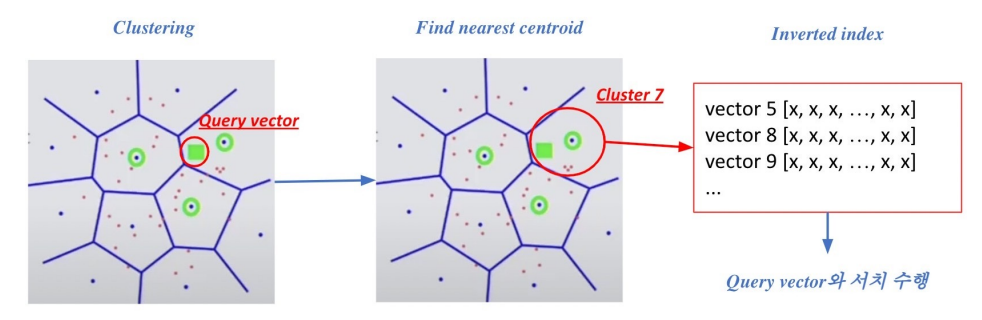
Clustering and IVF
-
Query vector와 유사한 3개의 centroid vector search
-
IVF를 이용해 centroid vector와 연결된 cluster 내의 vector들과 search
Faiss
문서 벡터뿐만 아니라 어떤 종류의 벡터든 유사성을 계산할 수 있음
FlatIndexL2
-
L2 유클리드 거리를 기반으로 벡터 간 거리를 계산
-
학습이 필요없음
-
모든 벡터를 메모리에 로드하고 유사도 검색을 수행하므로, 메모리 사용량이 많음
IndexIVFFlat
-
IVF(역파일 색인)을 이용해 인덱싱
-
학습이 필요하며, 군집 수와 분할 방법이 정의되어야 함
-
군집 내에서 Flat Index를 사용하므로 메모리 사용량 최적화
-
Flat Index는 양자화 과정 없이 벡터 그대로 저장하고 유사도 검색 수행
IndexIVFPQ를 이용하면 전체 벡터를 저장하지 않고 압축된 벡터만을 저장하며, 메모리 사용량을 줄일 수 있음
Using GPU

Scaling up with FAISS & 실습
brute-force
import faiss
d = 64 # 벡터 차원
nb = 100000 # 데이터베이스 크기
nq = 10000 # 쿼리 개수
xb = np.random.random((nb, d)).astype('float32') # 데이터베이스 예시
xq = np.random.random((nq, d)).astype('float32') # 쿼리 예시
### 인덱스 만들기 ###
index = faiss.IndexFlatL2(d) # 인덱스 빌드
index.add(xb) # 인덱스에 벡터 추가- 전체 db를 다 탐색하므로 학습이 필요 없음
### 검색하기 ###
k = 4 # 가장 가까운 4개의 벡터
D, I = index.search(xq, k) # D: 쿼리와의 거리, I: 검색된 vector indexIVF with FAISS
nlist = 100 # 클러스터 개수
quantizer = faiss.IndexFlatL2(d)
index = faiss.IndexIVFFlat(quantizer, d, nlist) # Inverted File 생성
index.train(xb) # 클러스터 학습
index.add(xb) # 학습된 클러스터에 벡터 추가
D, I = index.search(xq, k)-
quantizer
-
군집화 수행을 위해 사용되는 인덱스 타입 지정
-
quantizer로 IndexFlatL2를 지정한다면, IndexIVFFlat은 IndexFlatL2를 사용하여 군집의 중심 벡터를 저장하고, 검색 작업을 수행
-
-
nlist 개수만큼 cluster을 생성
IVF-PQ with FAISS
메모리 사용량을 SQ보다 줄일 수 있음
Using faiss.Clustering
import faiss
num_clusters = 16
niter = 5
k = 5
# 1. Clustering
emb_dim = p_embs.shape[-1]
index_flat = faiss.IndexFlatL2(emb_dim)
clus = faiss.Clustering(emb_dim, num_clusters)
clus.verbose = True
clus.niter = niter
clus.train(p_embs, index_flat)
centroids = faiss.vector_float_to_array(clus.centroids)
centroids = centroids.reshape(num_clusters, emb_dim)
quantizer = faiss.IndexFlatL2(emb_dim)
quantizer.add(centroids)
# 2. SQ8 + IVF indexer (IndexIVFScalarQuantizer)
indexer = faiss.IndexIVFScalarQuantizer(quantizer, quantizer.d, quantizer.ntotal, faiss.METRIC_L2)
indexer.train(p_embs) #p_embs : embedding된 passage vectors
indexer.add(p_embs)
# 3. Search using indexer
start_time = time.time()
D, I = indexer.search(q_embs, k)
print("--- %s seconds ---" % (time.time() - start_time))- 바로
index_flat.train()을 해도 되고, 다음처럼 풀어 쓸 수 있음
※ 모든 이미지 및 코드 출처는 네이버 커넥트재단 부스트캠프 AI Tech 5기입니다. ※