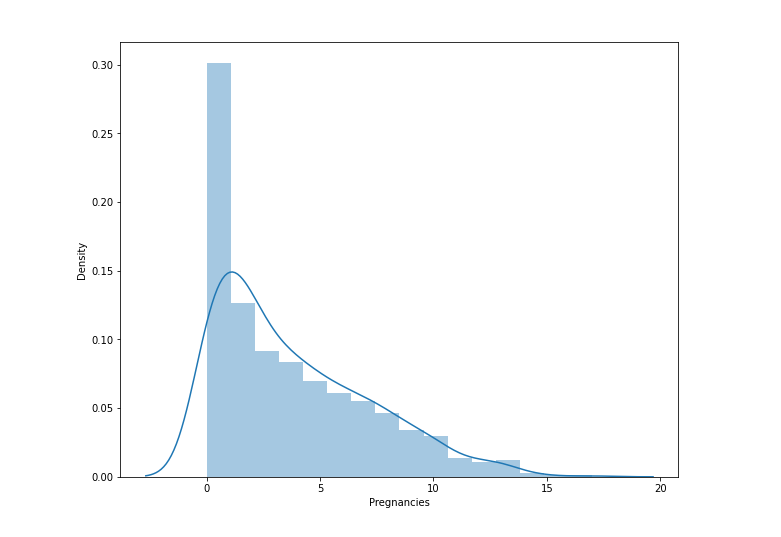
2.1.5 수치형 변수의 분포를 정답값에 따라 시각화
distplot
- 수치형 변수 1개의 분포를 파악할 때 사용
- histogram과 pdf도 함께 그려줌
plt.figure(figsize = (10, 8))
sns.distplot(df['Pregnancies'])
plt.show()
- 임신횟수의 분포를 보면 왼쪽으로 skewed된 형태를 보임.
- 횟수가 늘수록 상대적으로 적은 빈도
- histogram의 경우 bin의 수를 늘려주면 더 세밀하게 표현할 수 있음.
df_0 = df[df.Outcome == 0]
df_1 = df[df.Outcome != 0]
df_0.shape, df_1.shape

plt.figure(figsize = (10, 8))
sns.distplot(df_0.Pregnancies)
sns.distplot(df_1.Pregnancies)
plt.show()
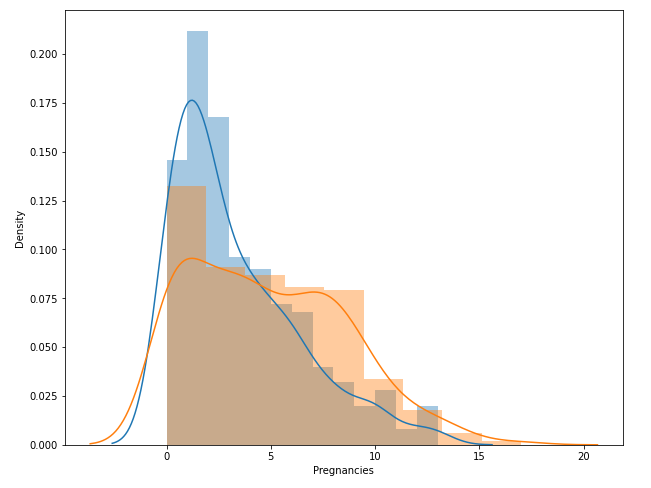
- 두 개의 변수의 distplot을 겹치게 그렸음
- 한 figure 내에 여러개도 그릴 수 있음
- 발병하지 않은 사람의 임신 횟수가 더 왼쪽으로 skewed된 형태임을 보임
- 임신 횟수가 5가 넘어가면 발병하는 경우의 pdf가 더 높은 부분에 위치 -> 임신 횟수가 높아지면 발병할 확률이 더 크다는 얘기
- pdf를 적분하면 확률 값
plt.figure(figsize = (10, 8))
sns.distplot(df_0.Age)
sns.distplot(df_1.Age)
plt.show()
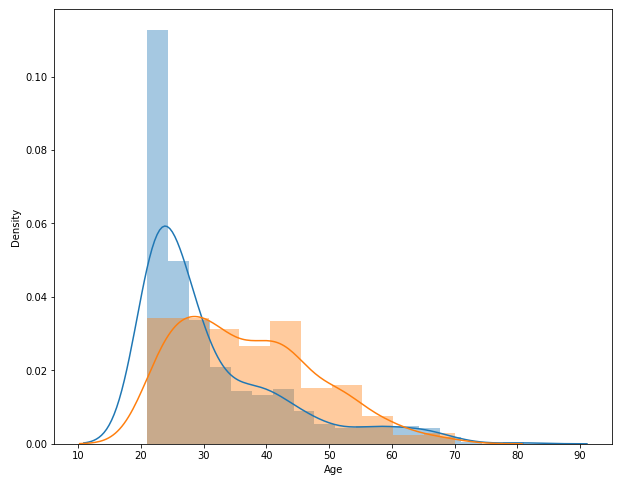
- 발병 여부에 따른 Age의 분포
- 30세가 넘어간 이후 발병 확률이 더 높아지므로 주의가 필요.
plt.figure(figsize = (10, 8))
sns.distplot(df_0.Age, hist = False)
sns.distplot(df_1.Age, hist = False)
plt.show()
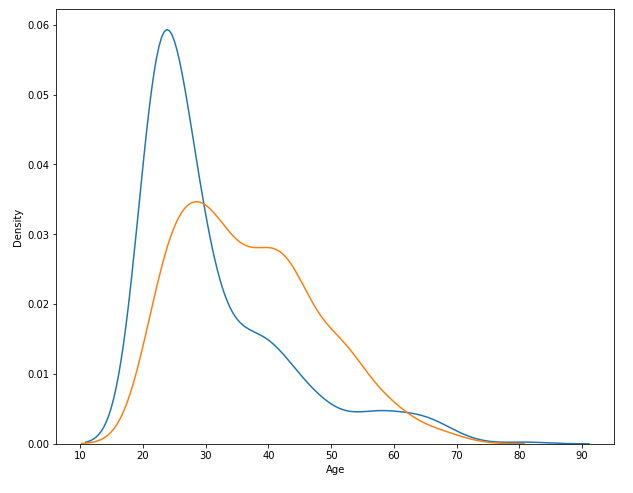
- hist = False를 해주면 histogram을 제외하고 pdf만 그려준다.
- 발병하지 않은 사람들의 나이 분포가 더 skewed -> 그만큼 젊은 나이대의 사람이 더 많다는 것
plt.figure(figsize = (10, 8))
sns.distplot(df_0.Age, hist = False, rug = True)
sns.distplot(df_1.Age, hist = False, rug = True)
plt.show()
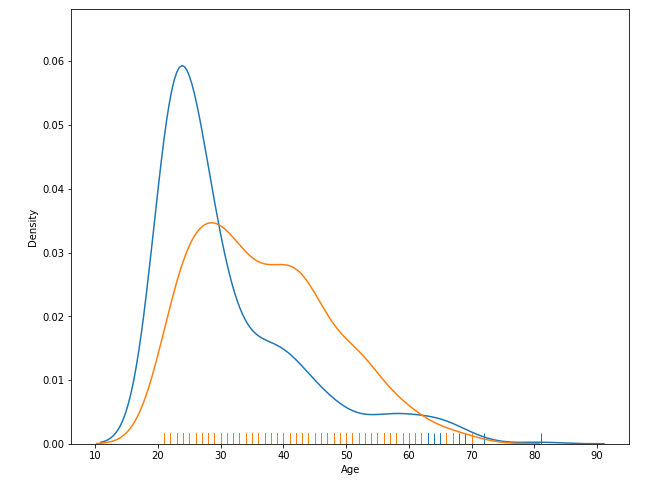
- 카펫같은 rug를 만들어 줌
- rug는 support하는 axis의 데이터를 1차원적으로 표현해주는 것.
plt.figure(figsize = (10, 8))
sns.distplot(df_0.Age, hist = False, rug = True, label = 'X')
sns.distplot(df_1.Age, hist = False, rug = True, label = 'O')
plt.legend()
plt.show()
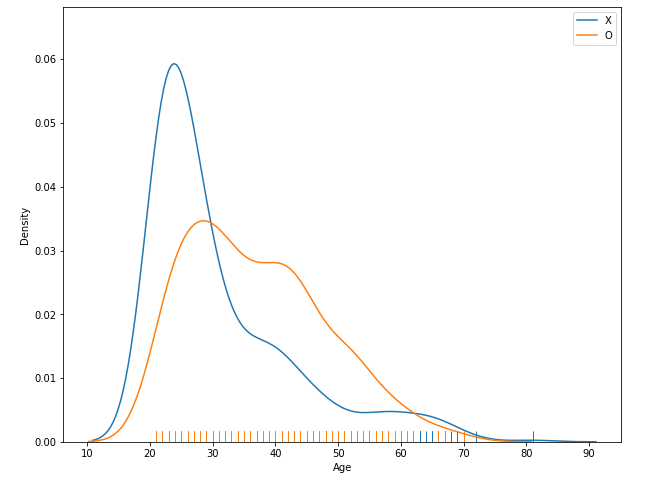
- label이 안보였으므로 legend를 추가해 줌. distplot의 인자로 label 명을 원하는 대로 줄 수 있고 입력된 라벨정보를
plt.legend()를 통해 범례를 추가해 줌
2.1.6 서브플롯으로 모든 변수 한번에 시각화 하기
subplot
- subplot은 다양한 그래프를 하나의 figure로 나타내고 grid의 형태는 알아서 지정
- 지정된 행/열 별로 그래프 정보를 입력해주면 됨.
df['Pregnancies_high'] = df['Pregnancies_high'].astype('int')
h = df.hist(figsize = (15, 15), bins = 20)
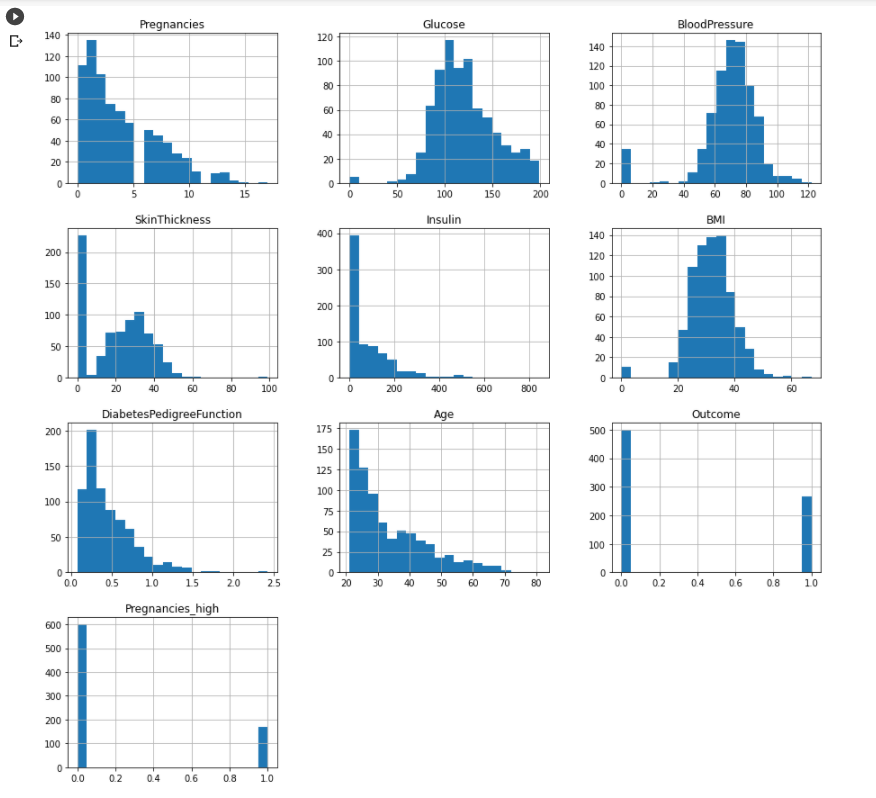
- df 내의 모든 변수들을 알아서 subplot을 이용해서 시각화 해줌
- 원래는 subplot을 이용해서 일일이 입력해줘야 하지만 상당히 편리!
- figsize와 막대의 개수를 지정 가능
- Outcome과 Pregnancies_high는 카테고리 변수였기 때문에 저런식으로 그려짐.
위처럼 편리하지 않고 직접 그려주기. 얼마나 위의 방법이 편리한지 알게될 것
col_num = df.columns.shape
col_num

반복문을 통해 column의 개수만큼 서브플롯을 그려줌
- plt.subplots를 이용해 row는3, col은3이고 figsize는 (15, 15)인 틀을 만들어 줌
fig, axes = plt.subplots(nrows = 3, ncols = 3, figsize = (15, 15))
- 이제 이 비어있는 틀에 하나씩 채워주는 것 with for문
fig, axes = plt.subplots(nrows = 3, ncols = 3, figsize = (15, 15))
sns.distplot(df.Outcome, ax = axes[0][0])
- 하나만 채워준 예시. 이렇게 하나씩 채워주긴 해야함
- 일일이 채우는 것 바보같은 짓이므로 for문을 이용해 채워줌!
cols = df.columns[:-1].tolist()
fig, axes = plt.subplots(nrows = 3, ncols = 3, figsize = (15, 15))
for i, col_name in enumerate(cols):
row = i // 3
col = i % 3
sns.distplot(df[col_name], ax = axes[row][col])
- axes 별로 graph를 채워줌!
- enumerate을 이용하면 list와 list size 만큼의 index 배열이 생기고 이를 동시에 return한다
- 그래서 i에는 각 컬럼의 인덱스, col_name엔 컬럼명이 할당!
- row엔 i를 3으로 나눈 몫이 col엔 나머지가 할당
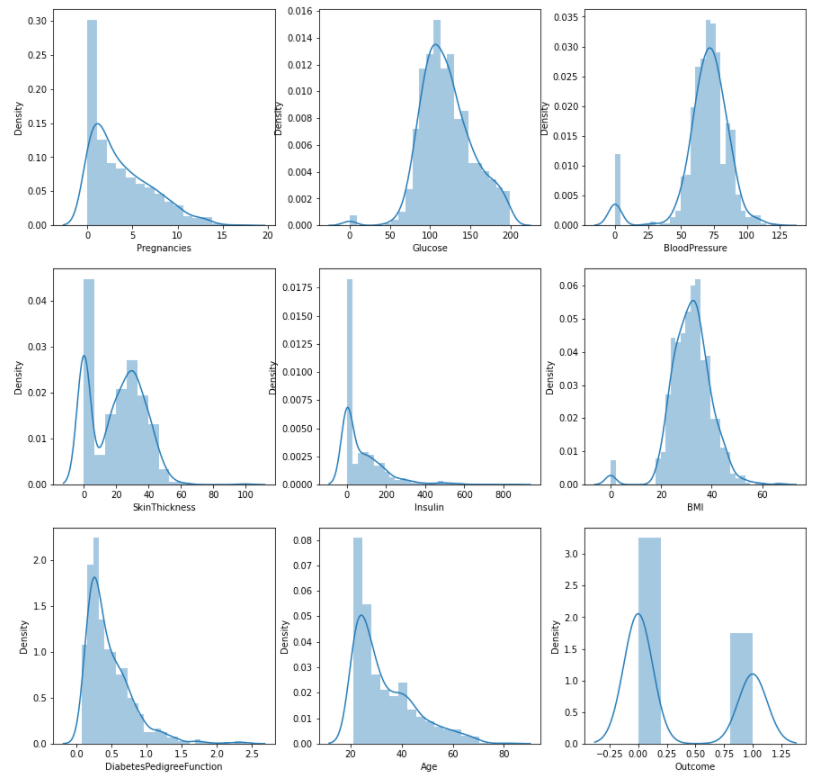
위와 같은 결과물을 반환
fig, axes = plt.subplots(nrows = 3, ncols = 3, figsize = (15, 15))
for i in range(3):
for j in range(3):
sns.distplot(df[df.columns[3*i + j]], ax = axes[i][j])
- subplots을 이용해 하나씩 그려주는 것의 장점은 각 그래프별로 customizing이 가능하다.
- bins의 수를 다 다르게 할 수도 있고, 막대의 색등 조정이 가능하지만 위에서 한 번에 그려준 것은 일률적이므로 커스터마이징이 어렵다.
- for문을 작성하기도 귀찮다면 그냥 위에서 사용한 방법을 쓰길.
fig, axes = plt.subplots(nrows = 4, ncols = 2, figsize = (15, 15))
for i, col_name in enumerate(cols[:-1]):
row = i // 2
col = i % 2
sns.distplot(df_0[col_name], ax = axes[row][col])
sns.distplot(df_1[col_name], ax = axes[row][col])
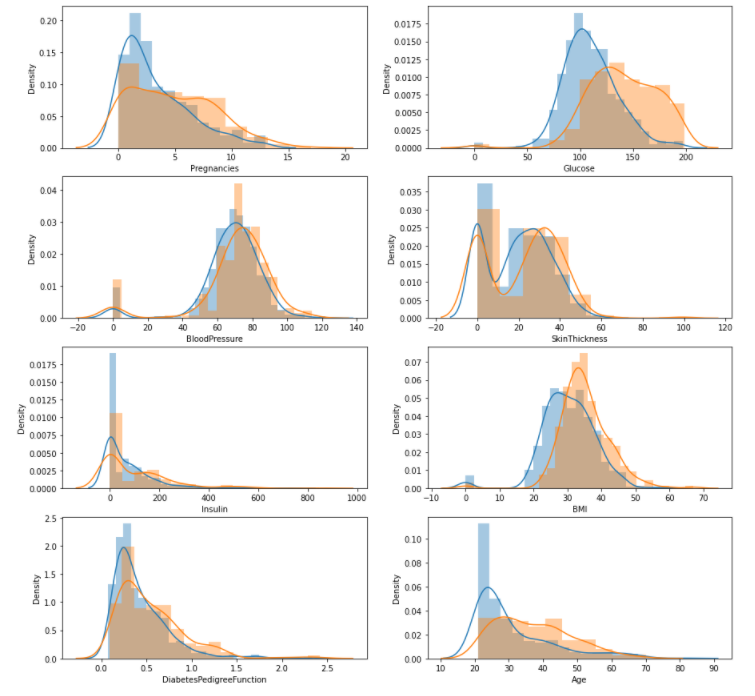
- 한 axes 별로 두 개의 변수를 넣을수도 있음!
- 발병 여부에 따른 각 변수들의 분포를 비교.
- 혈압과 피부 두께는 발병 여부에 따른 차이가 별로 없어보이고 글루코스, 임신횟수, BMI, AGE는 차이가 있어보인다.
2.1.7 시각화를 통한 변수간 차이 이해하기
Violin plot
- violin plot으로 subplot 그리기
fig, axes = plt.subplots(nrows = 4, ncols = 2, figsize = (15, 15))
for i, col_name in enumerate(cols[:-1]):
row = i // 2
col = i % 2
sns.violinplot(data = df, x = 'Outcome', y = col_name, ax = axes[row][col])
plt.show()
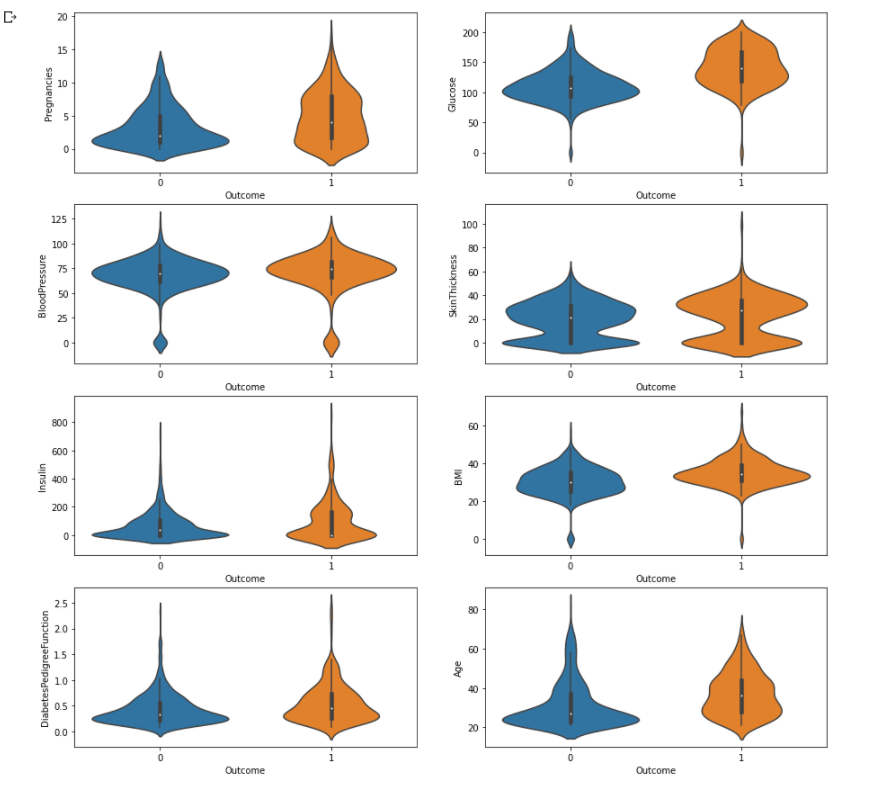
- Outcome 별로 각 변수들의 violin plot을 그려줌
- 대체적으로 당뇨병 환자들의 각 수치들이 조금 높게 형성되어있다고 생각된다.
- 포도당과 인슐린에서 0으로 몰린 것은 결측치 때문!!
plt.figure(figsize = (8, 6))
sns.regplot(data = df, x = 'Glucose', y = 'Insulin')
plt.show()
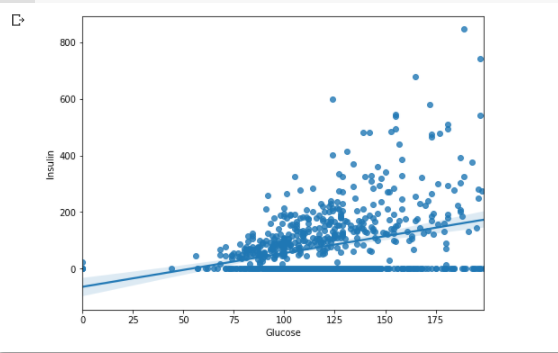
- regplot을 그려보면 인슐린이 0인 값들이 엄청 많음
- regplot을 통해 두 변수의 scatter plot을 그려주고 파란색 선이 회귀직선!
- 그 선을 감싸고 있는 투명한 부분은 신뢰구간? 신뢰영역?을 뜻함
- regplot엔 hue 옵션이 없으므로 두 개의 변수까지만 시각화가 가능.
- 세 변수의 시각화를 원하면 lmplot 이용하기
plt.figure(figsize = (10, 8))
sns.lmplot(data = df, x = 'Glucose', y = 'Insulin', hue = 'Outcome', ci = False)
plt.show()
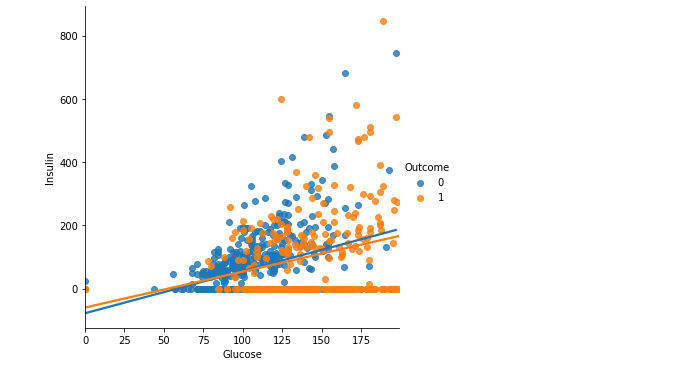
- ci = False로 주어 신뢰영역이 보이지 않게 함
- 발병 여부에 따라 색을 다르게 해서 각각의 산점도와 회귀직선을 시각화
- 당뇨병에 걸리지 않은 사람의 직선의 기울기가 좀 더 가파르다 -> 더 큰 영향을 미침
- why? 글루코스 한 단위의 변화에 기울기 만큼 인슐린이 변한는데 기울기가 더 가파르단 얘기는 더 크게 값이 변하므로.
plt.figure(figsize = (10, 8))
sns.lmplot(data = df[df.Insulin > 0], x = 'Glucose', y = 'Insulin', hue = 'Outcome', ci = False)
plt.show()
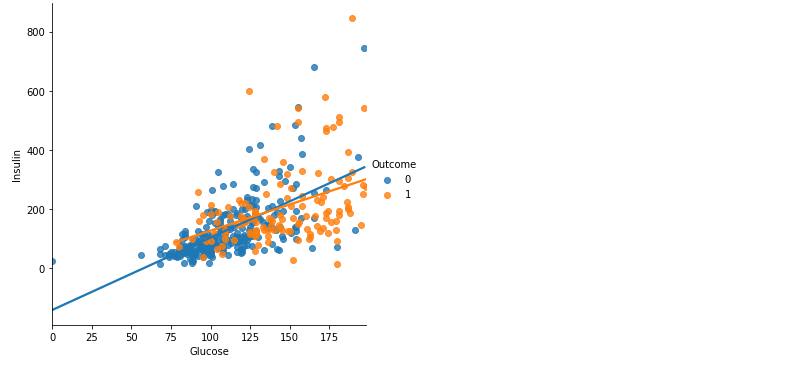
- 0보다 큰 값들로만 시각화 => 결측치를 제외하고 시각화!
- 발병 여부에 관계 없이 비슷한 경향을 보인다고 생각됨
Pairplot
- 데이터 프레임 내 모든 변수들의 산점도를 그려주고 diagonal 위치엔 각 변수의 histogram을 그려줌
- 변수의 수를 n이라 한다면 n*n개의 그래프를 그려주는 것이라 시간이 꽤 걸림
sns.pairplot(df)
plt.savefig('pairplot.png')
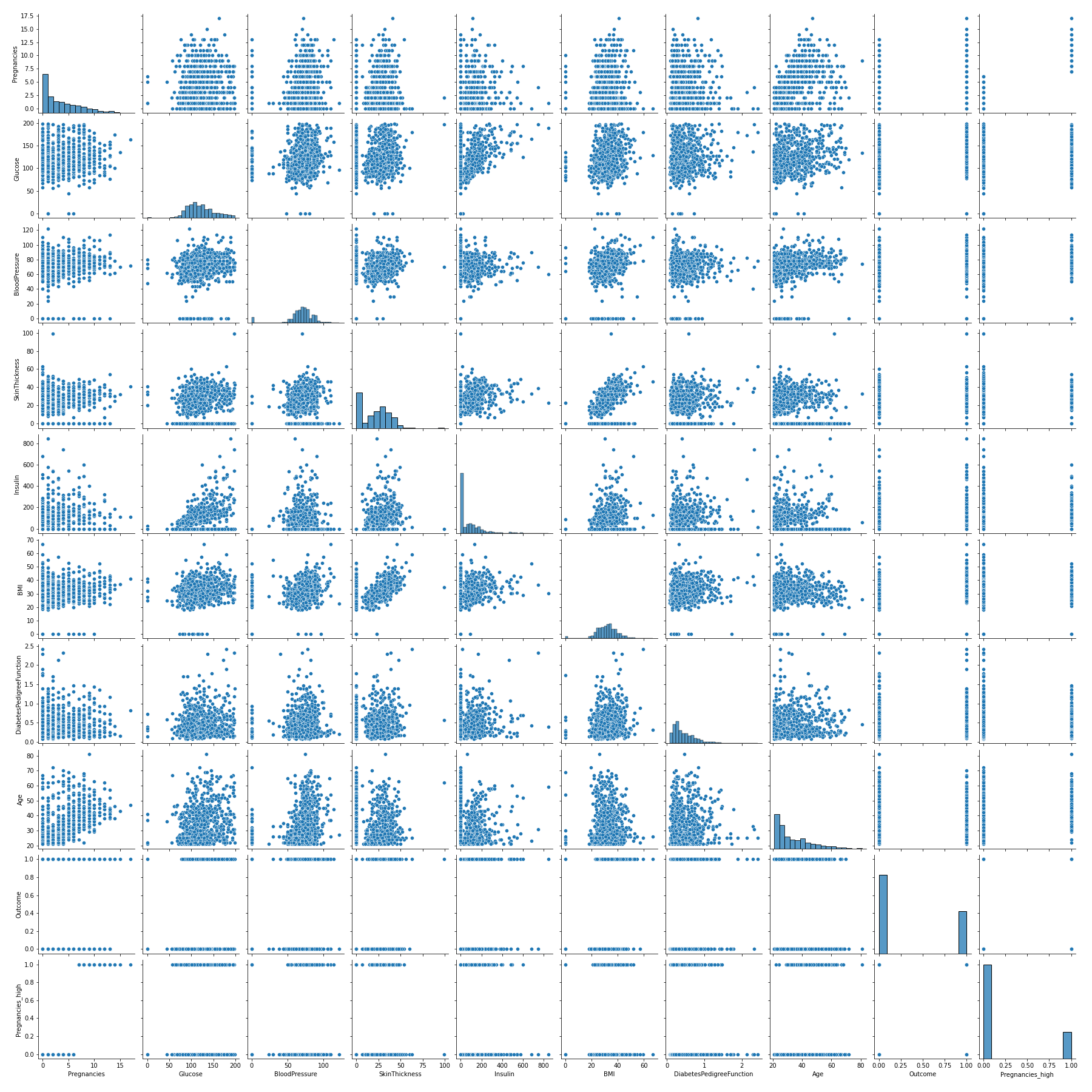
- 각 변수의 분포를 볼 뿐만 아니라 변수 별로 산점도를 보며 관계 파악이 가능
- 독립변수간의 상관관계가 존재한다면 다중공선성이 발생할 수도 있고 이는 불필요한 변수의 중복이 되기 때문에 성능 차이를 확인해보고 별 차이 없다면 빼주는게 맞음
- 이상적인 모델은 간결하되 정확도가 높은 모델!
g = sns.PairGrid(df, hue = 'Outcome')
g.map(plt.scatter)
plt.savefig('PariGrid.png')
- PairGrid를 이용해 hue 옵션을 넣어주고 map을 이용해서 산점도를 그려줌!
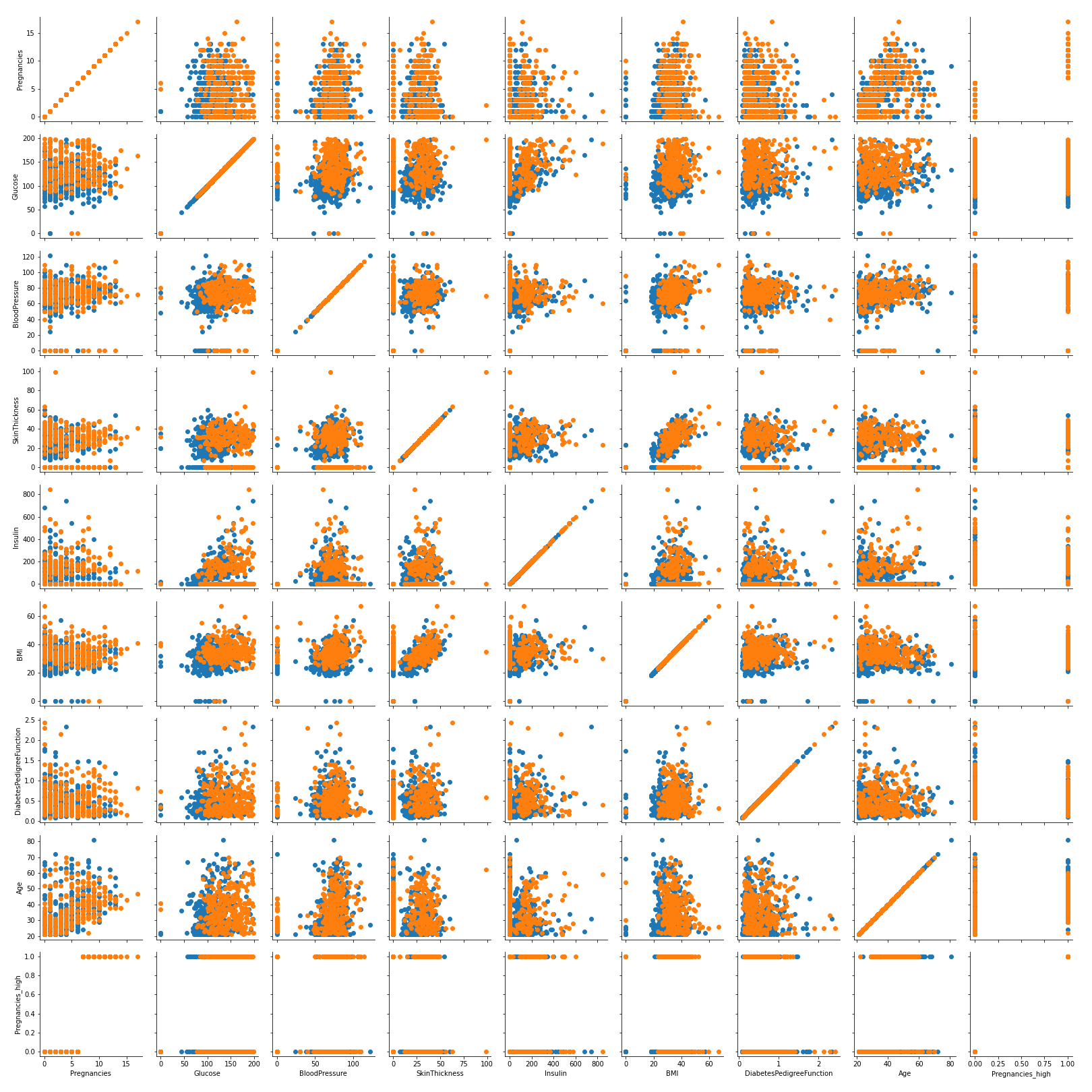
- 발병여부 별로 각 변수들의 산점도를 그려보았다.
- 이러한 부분들은 EDA에서 많이 하고, 각 변수들의 분포는 어떤지, 변수간 관계는 어떤지등을 파악해서 불필요한 부분들은 제거하고, 이상치가 있는지 등을 눈으로 파악하기 좋음.
2.1.8 피처엔지니어링을 위한 상관 계수 분석
상관분석
- 두 변수 간의 선형/비선형 관계를 갖고 있는 지를 분석
- 1에 가까우면 강한 양의 상관, -1에 가까우면 강한 음의 상관, 0은 두 변수가 독립적 => 상관X
df_corr = df.corr()
df_corr.style.background_gradient()
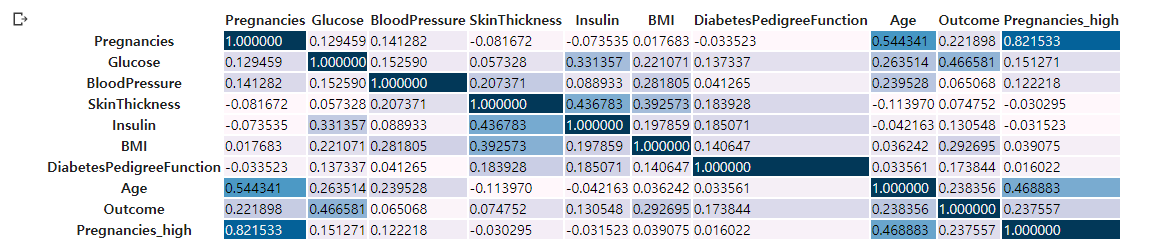
.corr()을 이용해 컬럼과 인덱스에 각 변수가 들어가고 이 둘의 상관계수가 담긴 행렬을 생성한다.- 생선된 행렬에
.style.background_gradient() 을 이용하면 위처럼 히트맵 느낌으로 상관계수 행렬을 시각화 해준다.
sns.heatmap(df.corr())
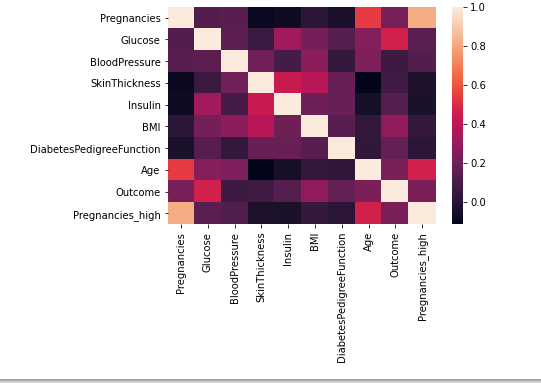
- 히트맵의 특징은 두 변수의 각 범주 별 비교를 할 때 유용
- 시간대/요일별 승객의 수와 같은 것을 시각화 할때도 유용.
- 그냥 전체 격자 내에 값에 따라 색을 다르게 해서 격자 별로 비교하기에 좋음.
- Pregnancies_high는 Pregnancies의 파생변수라 당엲 높은 상관관계를 가질 수 밖에 없음
- 좀 눈에 띄는게 Age와 Pregnancies, Outcome과 Glucose 등이 상대적으로 강한 양의 상관관계를 가진다고 생각된다.
sns.heatmap(df.corr(), vmax = 1, vmin = -1)
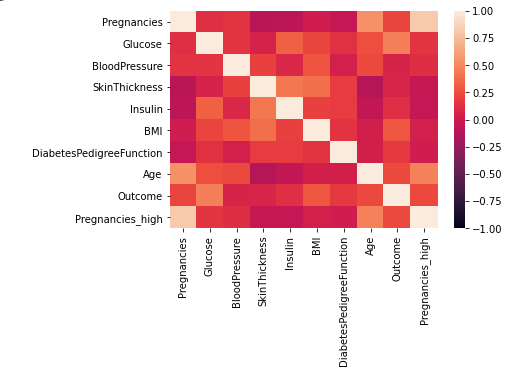
- vmin을 -1로 지정해줘 음의 상관도 파악. Age와 피부 두께는 상관계수 값이 음으로 파악됨.
plt.figure(figsize = (15, 8))
sns.heatmap(df.corr(), annot = True, vmax = 1, vmin = -1, cmap = 'coolwarm')
plt.show()
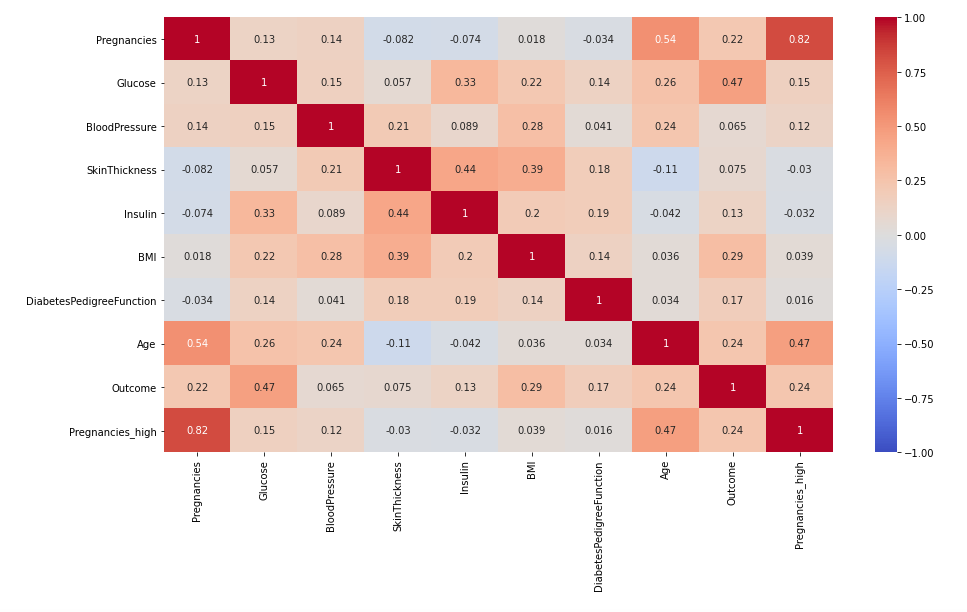
- annot = True로 하면 각 그리드의 값들을 같이 그려줌 -> 상관 계수의 값도 붙여주는 것
- cmap을 이용해서 다양한 색으로 표현 가능!!
- Outcome에 글루코스, BMI, Age가 상대적으로 높은 상관계수를 가짐.
- 절대적으로 높고 낮음의 기준은 없음. 해당 산업군에서의 통용되는 기준을 쓰거나 분석자의 주관에 따름.
df.iloc[:, :-2].replace(0, np.nan)
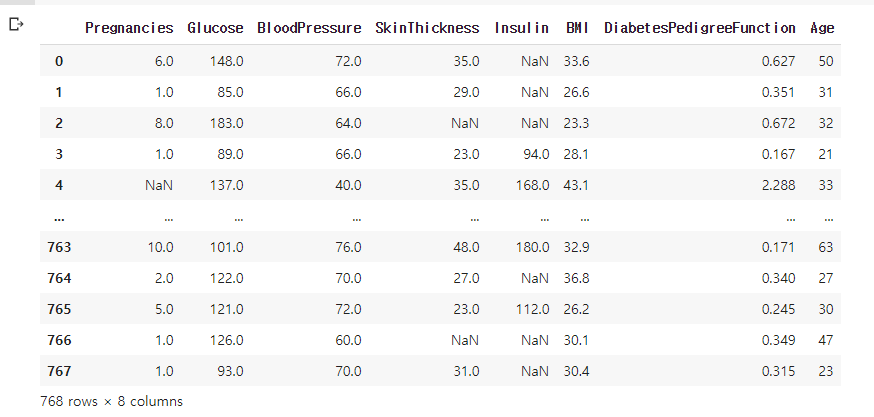
- iloc을 이용해 마지막 두 컬럼을 제외하고 selecting. 0인 것들은 결측치로 처리해 줌.
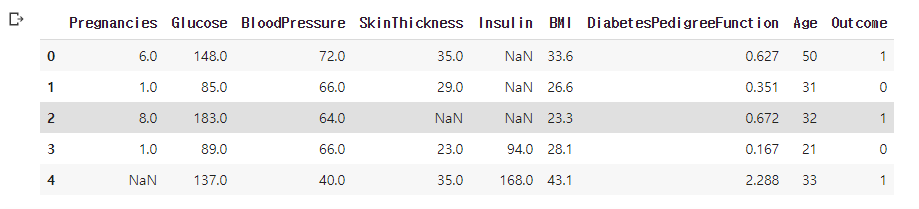
df_matrix = df.iloc[:, :-2].replace(0, np.nan)
df_matrix['Outcome'] = df['Outcome']
df_matrix.head()
plt.figure(figsize = (15, 8))
sns.heatmap(df_matrix.corr(), annot = True, vmax = 1, vmin = -1, cmap = 'coolwarm')
plt.show()
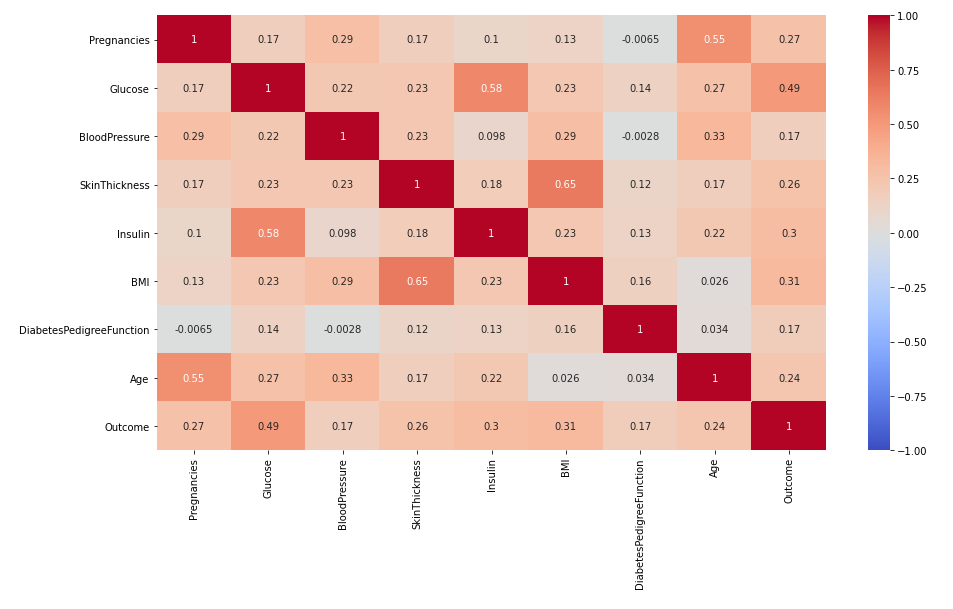
- nan 값을 제외하고 평균등을 계산하므로 corr은 결측치를 제외하고 계산된다.
- 0이라는 값들은 결측치였지만 0으로 있었기 때문에 계산될 때 포함되어 제대로 된 수치 계산이 안되었음
- nan 처리를 해주고 난 후 상관 계수를 보니 혈압과 인슐린의 상관계수가 상대적으로 높아졌음
df_matrix.corr()[['Outcome']]

plt.figure(figsize = (15, 8))
sns.regplot(data = df_matrix, x = 'Insulin', y = 'Glucose')
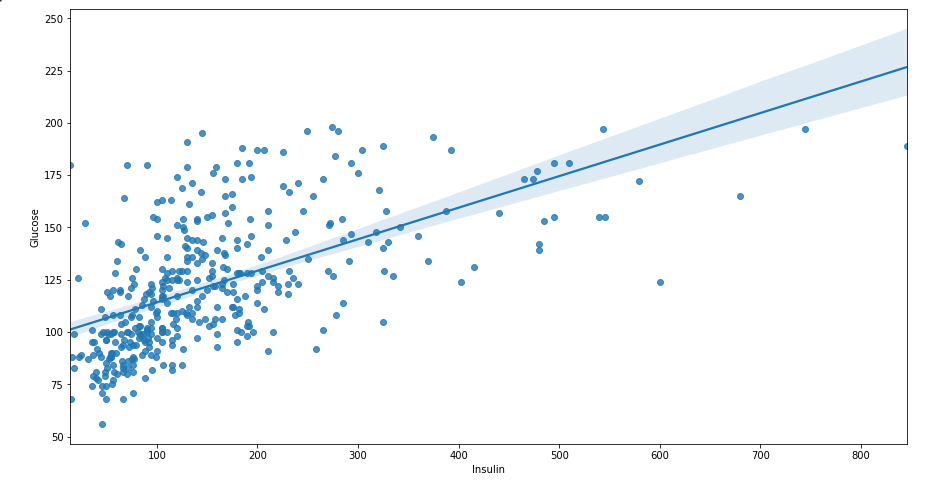
- Beta = Cov(x,y)/var(x)∗var(y) 이고 r=Cov(x,y)/Sx∗Sy
- B=r∗Sy/Sx로 표현 가능.
- B는 회귀계수(상수항 제외), r은 상관계수
- r이 커지면 B도 커질 수 밖에 없음.
- 기울기가 가파르다면 => 그만큼 높은 상관계수를 가진다는 의미
plt.figure(figsize = (15, 8))
sns.regplot(data = df, x = 'Age', y = 'Pregnancies')
plt.show()
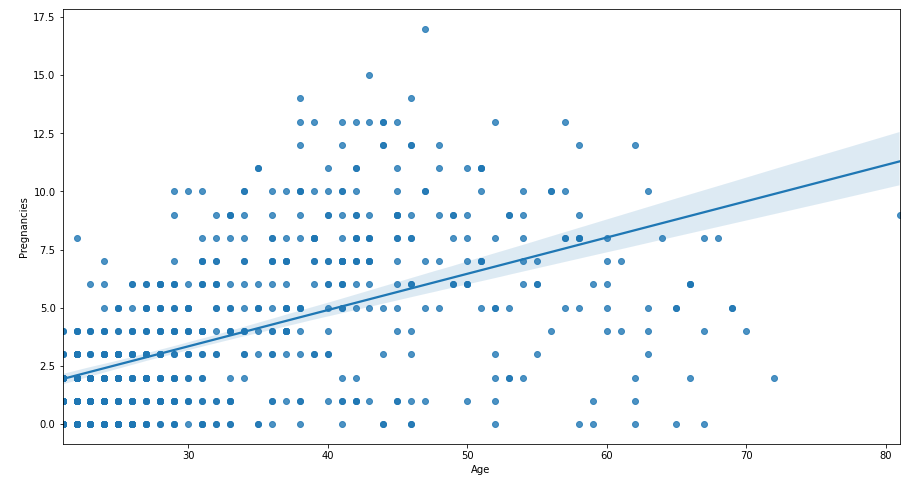
- 임신 횟수와 Age의 산점도 + 회귀선
- 임신 횟수는 이산형이므로 단순 linear를 적용할 것이 아닌 Logistic Regression을 이용해야 함.
plt.figure(figsize = (15, 8))
sns.lmplot(data = df, x = 'Age', y = 'Pregnancies', hue = 'Outcome')
plt.show()
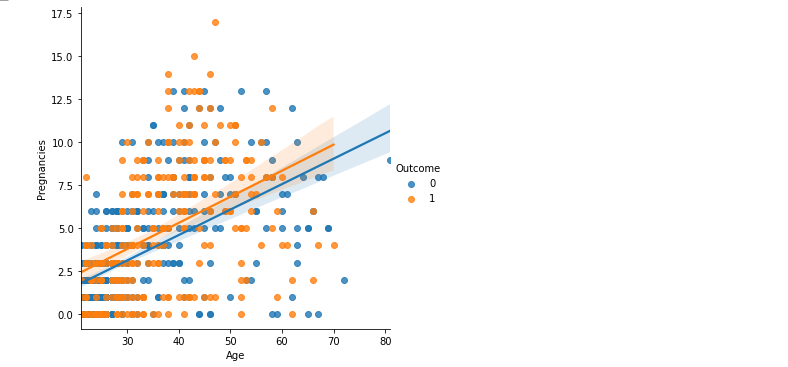
- 회귀선이 조금 다르게 그려졌음 -> 절편의 값만 다르고 기울기는 유사하다 생각
- 마찬가지로 LinearRegression이 아닌 LogisticRegression을 적용해야 한다 생각
plt.figure(figsize = (15, 8))
sns.lmplot(data = df, x = 'Age', y = 'Pregnancies', hue = 'Outcome', col = 'Outcome')
plt.show()
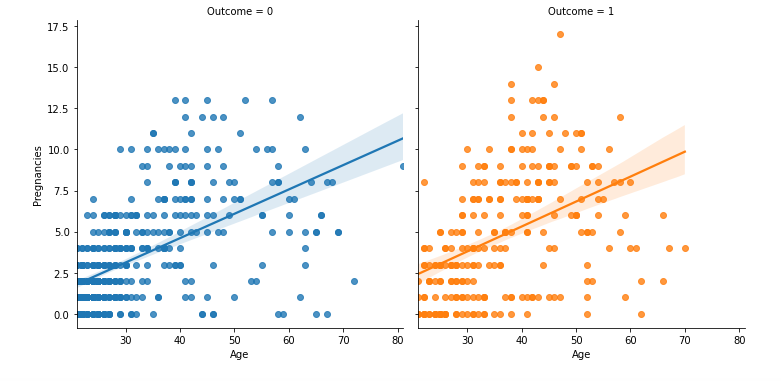
- x, y, hue, col, size, alpha 등을 활용해서 다양한 변수를 한 번에 시각화가 가능
- 너무 많은 걸 시각화 하면 해석이 어려울 수 있으므로 주의
- col을 이용해서 발병 여부에 따라 두 개의 그래프를 그림