2.1.1 당뇨병 데이터셋 미리보기
데이터 구성
- Pregnancies : 임신 횟수
- Glucose : 2시간 동안의 경구 포도당 내성 검사에서 혈장 포도당 농도
- BloodPressure : 이완기 혈압 (mm Hg)
- SkinThickness : 삼두근 피부 주름 두께 (mm), 체지방을 추정하는데 사용되는 값
- Insulin : 2시간 혈청 인슐린 (mu U / ml)
- BMI : 체질량 지수 (체중kg / 키(m)^2)
- DiabetesPedigreeFunction : 당뇨병 혈통 기능
- Age : 나이
- Outcome : 768개 중에 268개의 결과 클래스 변수(0 또는 1)는 1이고 나머지는 0입니다.
EDA 진행
import pandas as pd
import numpy as np
import seaborn as sns
import matplotlib.pyplot as plt
data shape 확인
df = pd.read_csv('/content/drive/MyDrive/edwith/프로젝트로 배우는 데이터 사이언스/diabetes.csv')
df.shape

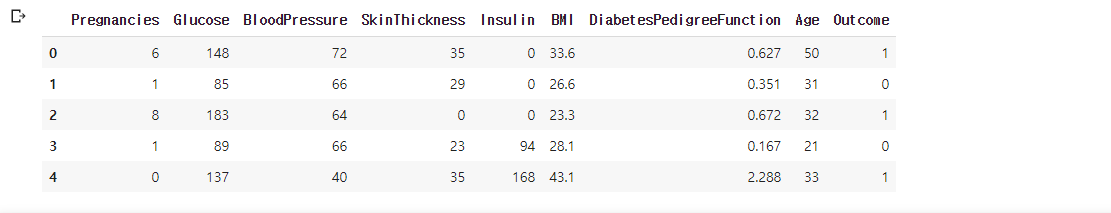
data 상위 5개 관측치 미리보기
df.head()
data 내 변수들의 타입과 결측치, 메모리 사용량 확인
df.info()
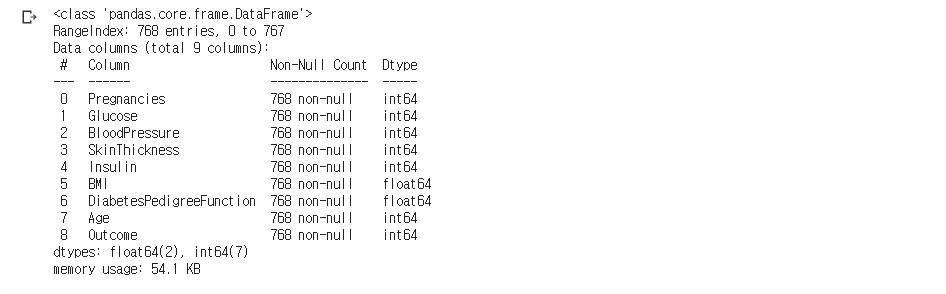
- 모든 컬럼에 결측치가 하나도 없고, 수치형 변수들이다. float이냐 int냐의 차이는 있지만.
결측치 확인
df.isna()
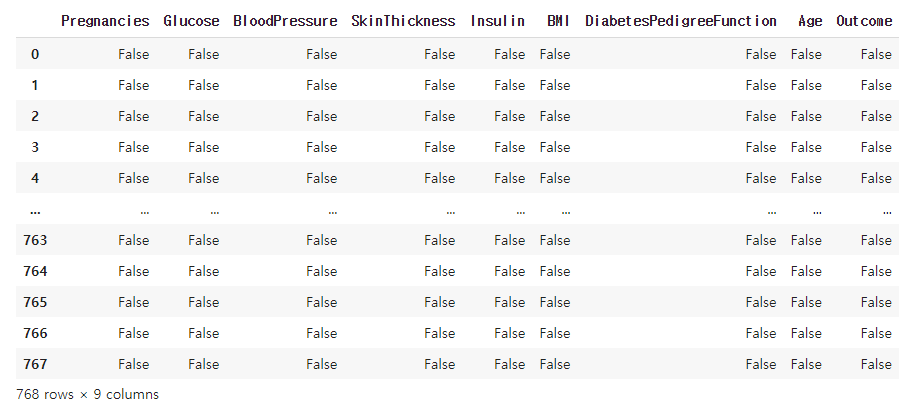
- 결측되었다면 True, 아니면 False를 반환
df.isna().sum()

- sum을 해줘 컬럼별로 결측치의 수를 세어줌. data frame에 사용할 수 잇는 매서드들은 대부분 컬럼을 기준의 apply임.
- row별 관측치의 수를 구하고 싶다면 아래처럼.
df.isna().apply(sum, axis = 1)
기술통계량 확인
df.describe()
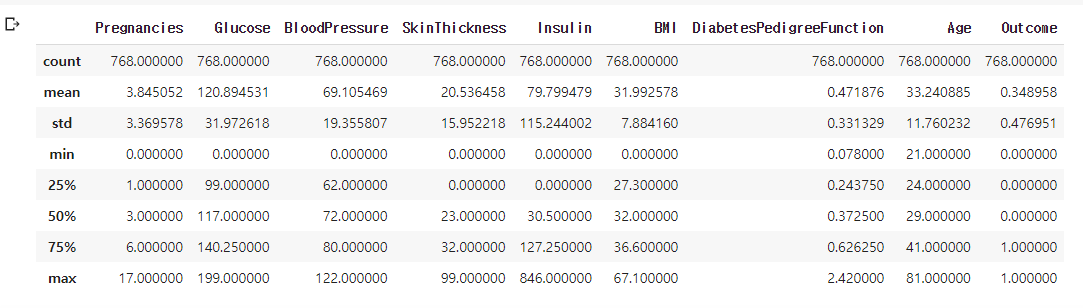
- 수치형 변수들이므로 평균, 표준편차, 최소, 최대, 사분위수들을 return 해줌
- Object type 이라면 count와 최빈값 등을 return
feature_columns = df.columns[:-1].tolist()
feature_columns

- df.columns는 index 형태로 return하고 인덱싱을 통해 마지막 값인 타겟을 제외하고 tolist()를 이용해 리스트로 만들어 줌.
2.1.2 결측치 보기
cols = feature_columns[1:]
- features에서 첫 번째 컬럼만 제외하고 cols에 담아줌
df_null = df[cols].replace(0, np.nan)
df_null = df_null.isnull()
df_null.sum()

- pregnancies를 제외한 나머지 컬럼을 cols에 넣어줬었고 cols에 해당되는 모든 컬럼에 대해 0인 경우는 np.nan으로 임의로 null값으로 대체해 줌.
- 결측치의 수는 0의 수라고 생각하면 됨.
plt.figure(figsize = (10, 8))
df_null.sum().plot.barh()
plt.show()
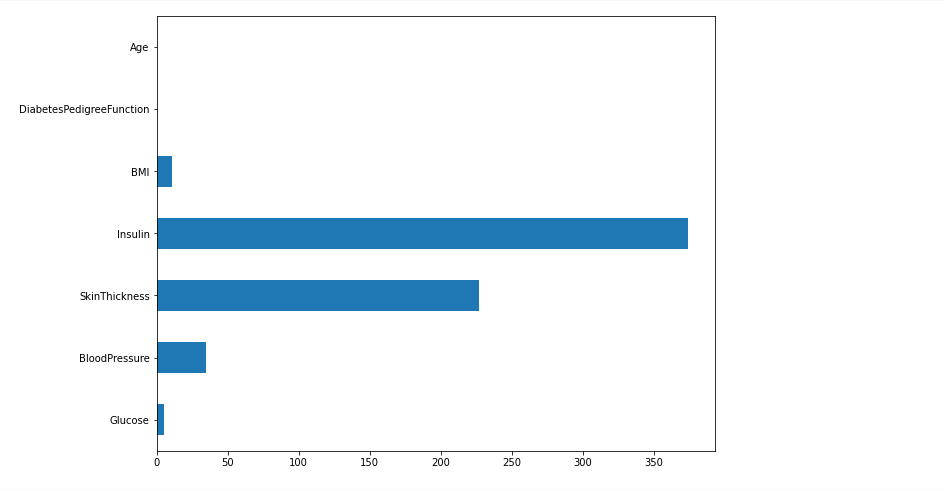
- pandas의 plot를 이용해서 편리하게 결측치 수 시각화!
- 인슐린이 0인 값이 가장 많음
- 이전 트리 모델의 중요도에서 인슐린 수치가 중요하지 않다고 나왔었는데 0인 값들이 너무 많았기 때문이 아닐까란 생각.
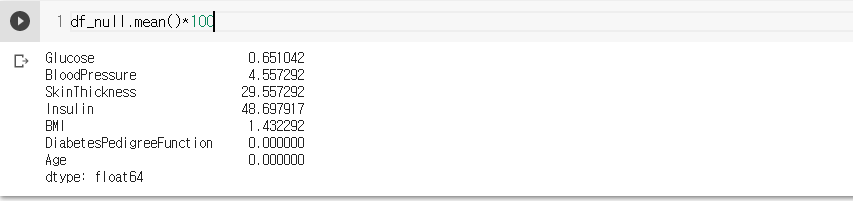
- 결측치의 비율(%)을 계산
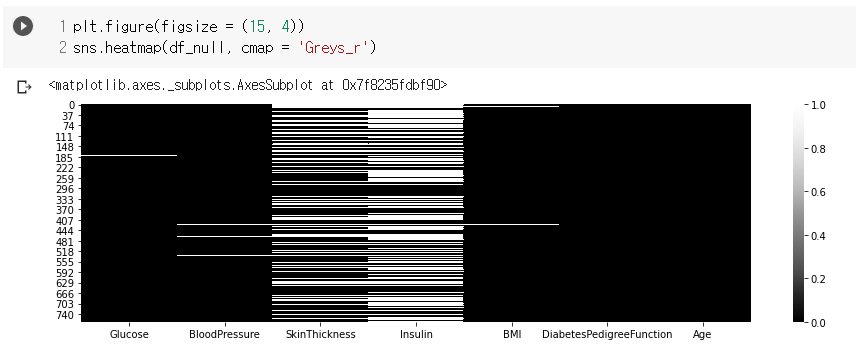
- y축을 idnex로 x축을 columns로 히트맵을 그려 결측치를 시각화 함.
- df_null의 값이 1이라면 결측치, 0이라면 결측치X. 따라서 흰색이 결측치에 해당
- 시각화를 통해 어느 위치에 결측치가 많은지 파악.
2.1.3 훈련과 예측에 사용할 정답값을 시각화
정답값
df['Outcome']

df['Outcome'].value_counts()

df['Outcome'].value_counts(normalize = True)

- normalize를 True로 주어 비율을 파악.
- 두 클래스의 비율이 어느정도 맞는게 좋음. Why? 하나의 클래스에 대해서만 학습을 하게돼 다른 클래스를 제대로 맞추지 못하는 경우가 발생.
- 이런 경우엔 부족한 쪽을 오버샘플링 하거나 많은 쪽을 언더 샘플링.
- 샘플의 불균형도 해결해줘야 하는 문제임.
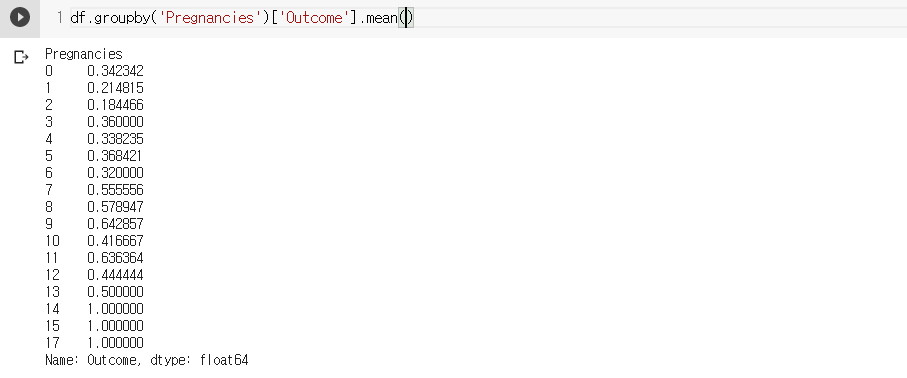
- 임신 횟수 기준으로 groupby해서 Outcome의 평균을 계산해 줌.
- Outcome은 0, 1 이므로 평균을 계산해주면 1의 비율이 됨.
- 임신의 횟수에 따라 당뇨에 환자의 수가 들쭉날쭉함.
- 14, 15, 17인 경우 무조건 당뇨라고 할 수는 없음. 왜냐 빈도수 자체가 너무 적을 것이기 때문. 빈도수도 보면서
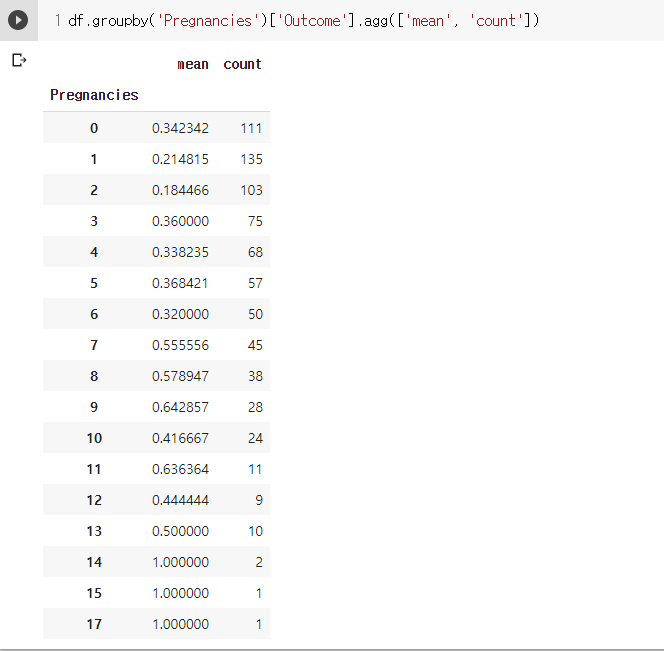
- 임신의 횟수가 많은 경우의 빈도가 당연히 적을 것이라 생각.
- 임신의 횟수가 많아지면 당뇨에 걸린 환자가 많다라는 결론을 도출하기는 애매한듯.
df_po = df.groupby('Pregnancies')['Outcome'].agg(['mean', 'count']).reset_index()
df_po
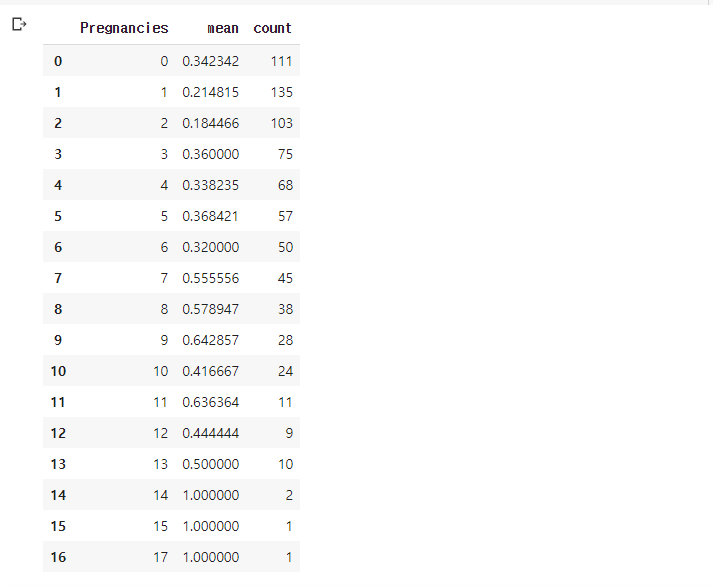
- index였던 Pregnancies를 컬럼으로 사용하기 위해 reset_index()!
- agg를 이용해서 다양한 집계함수를 추가 가능함. sum, min, max....등등
df_po.plot()
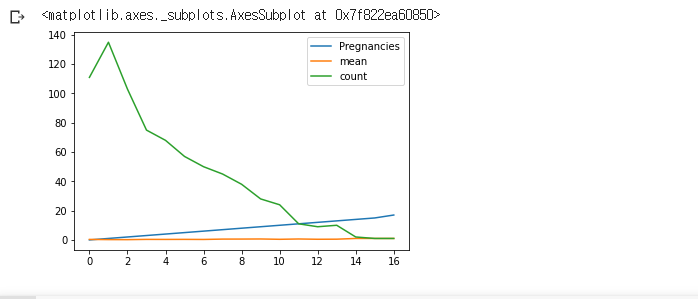
- df_po의 인덱스를 기준으로 컬럼별 lineplot을 그림.
- 임신횟수는 서서히 증가하고, Count는 급격한 감소.
- mean은 별다른 차이가 존재하지 않는다.
Count의 Scale이 상대적으로 많이 크기 때문에 다르 변수들 line의 모양이 제대로 안 보이는 것!!
df_po['mean'].plot()
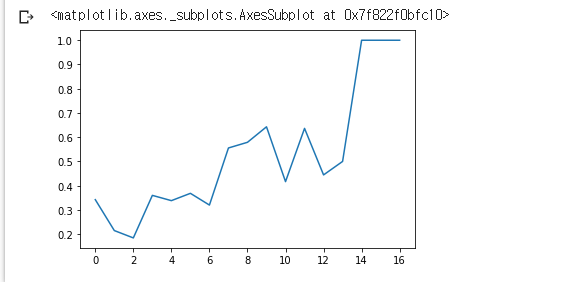
df_po['mean'].plot.bar()
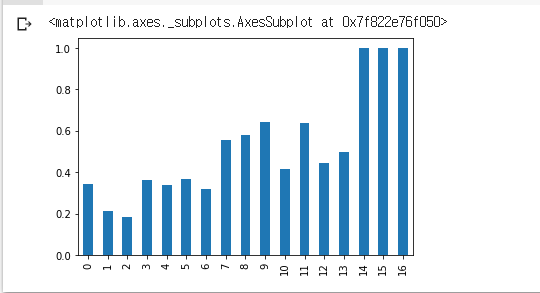
- 인덱스별 mean의 값. 전체적으로 본다면 우상향 하는 경향을 보인다고 생각됨.
df_po['mean'].plot.bar(rot = 0)
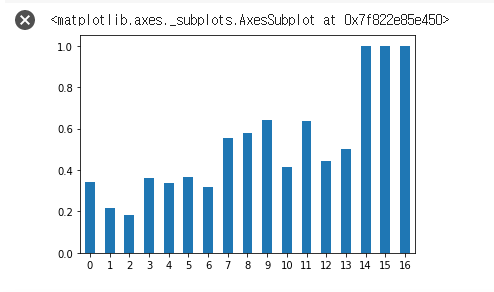
- 원래는
plt.xticks(rot = 0) 이런식으로 지정해줘야 하는데 판다스의 plot을 이용해서 바로 시각화 해주면 훨씬 편리
countplot
sns.countplot(data = df, x = 'Outcome')
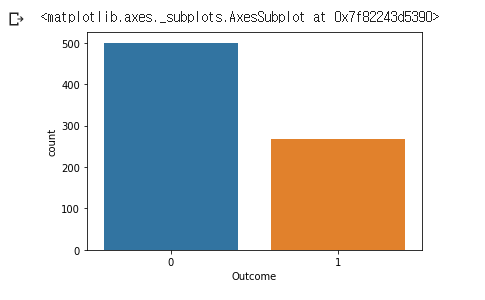
- seaborn의 countplot을 이용해 클래스별 빈도를 막대그래프로 시각화!
df['Outcome'].value_counts().plot.bar(rot = 0)와 동일. countplot이 훨씬 간결. 클래스별 색도 구분, x축과 y축의 label까지 자동이므로
plt.figure(figsize = (10, 8))
sns.countplot(data = df, x = 'Pregnancies')
plt.show()
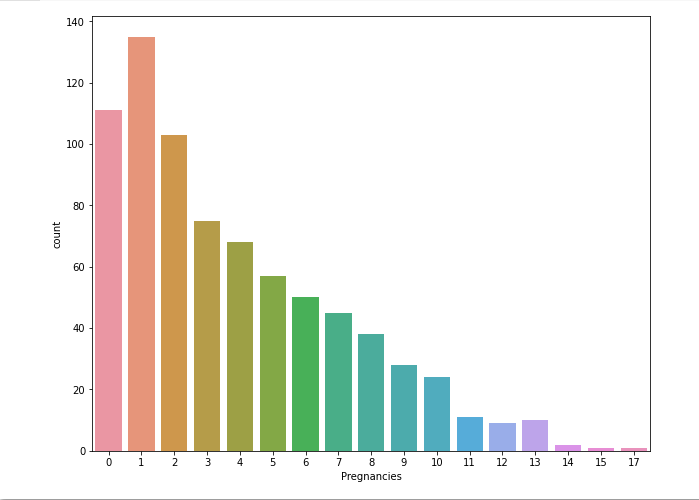
- 임신 횟수의 시각화. 14, 15, 17 이런 값들은 이상치가 아닐까...? 가능한 부분인가 의심된다.
plt.figure(figsize = (10, 8))
sns.countplot(data = df, x = 'Pregnancies', hue = 'Outcome')
plt.show()
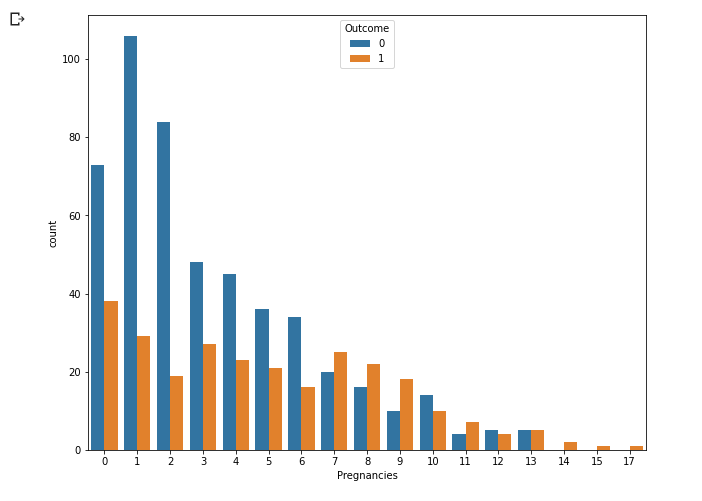
- 임신횟수에 따른 당뇨병 발병 빈도수를 확인
- 6회 까지는 발병하지 않은 경우가 많지만 7, 8, 9는 발병이 더 많음.
- 임신의 범주는 총 16개로 가지가 너무 많이 갈라지게 되고 이는 곧 오버피팅을 초래할 수 있으므로 2개의 범주로 묶어줌.
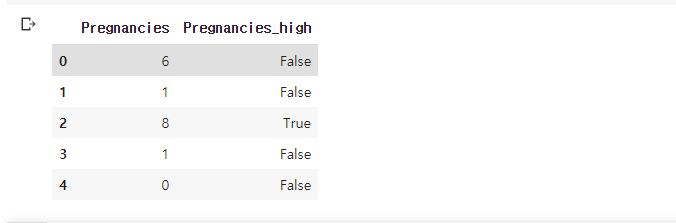
df['Pregnancies_high'] = df['Pregnancies'] > 6
df[['Pregnancies', 'Pregnancies_high']].head()
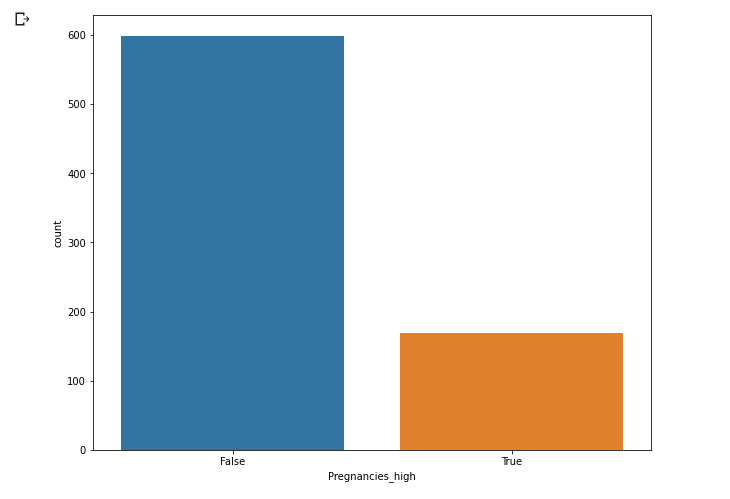
plt.figure(figsize = (10, 8))
sns.countplot(data = df, x = 'Pregnancies_high')
plt.show()
plt.figure(figsize = (10, 8))
sns.countplot(data = df, x = 'Pregnancies_high', hue = 'Outcome')
plt.show()
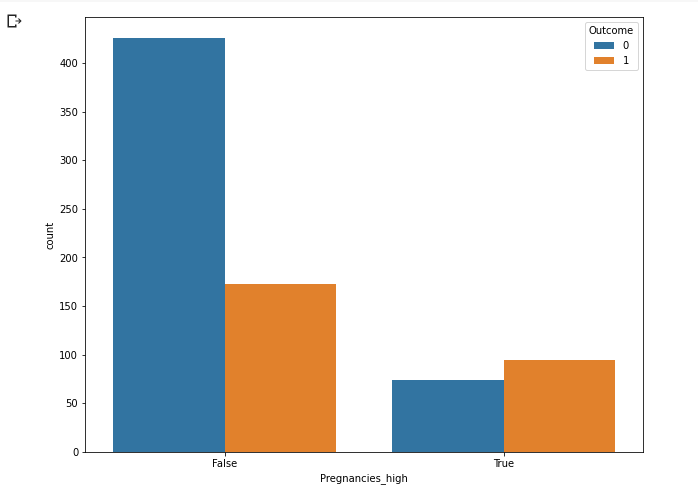
- High인 경우와 아닌 경우의 당뇨병 발생 빈도수를 확인
- High가 아닌 경우엔 발병하지 않은 빈도가 높지만 High인 경우엔 발병의 빈도가 더 높음.
2.1.4 두 개의 변수를 정답값에 따라 시각화 해보기
barplot
plt.figure(figsize = (10, 8))
sns.barplot(data = df, x = 'Outcome', y = 'BMI')
plt.show()
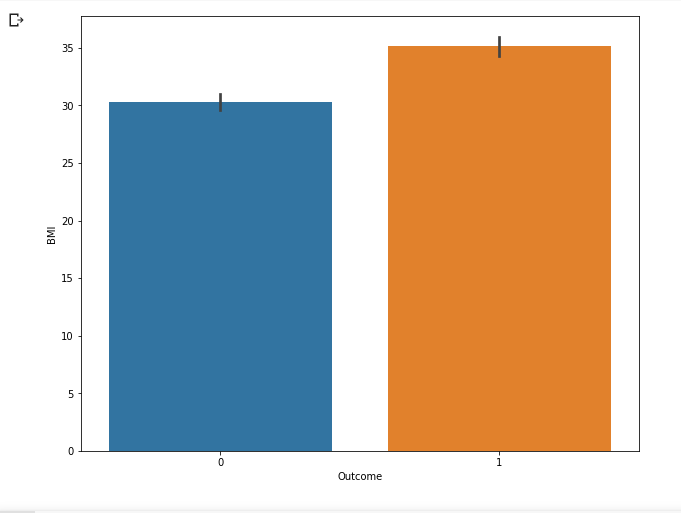
- 발병한 경우가 BMI 수치가 좀 더 높음. 원래 barplot은 범주형 변수에 대해 빈도수 시각화 하는 경우에 사용됨.
- x축을 범주형으로, y축을 수치형 변수로 넣어주면 수치형 변수의 평균값을 높이로 가지는 막대그래프를 그려줌.
- 즉, 발병여부에 따른 BMI 평균값의 차이를 시각화 한 것.
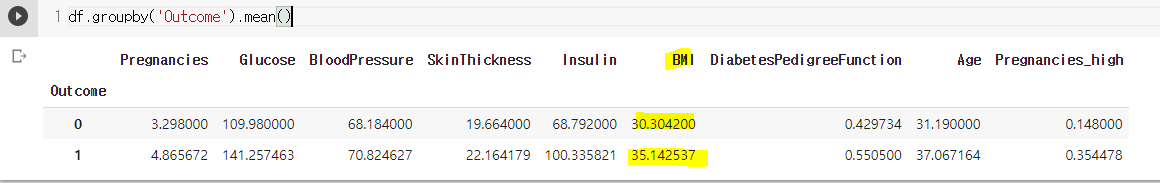
plt.figure(figsize = (10, 8))
sns.barplot(data = df, x = 'Outcome', y = 'Glucose')
plt.show()
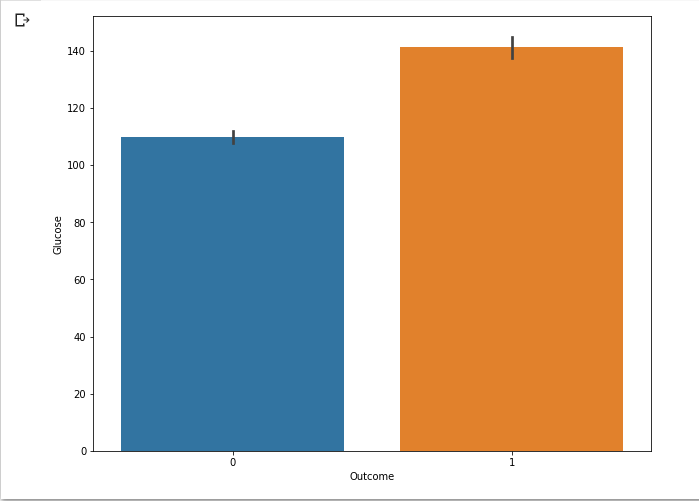
- 당뇨병 환자일수록 글루코스의 평균값이 더 높음
plt.figure(figsize = (10, 8))
sns.barplot(data = df, x = 'Outcome', y = 'Insulin')
plt.show()
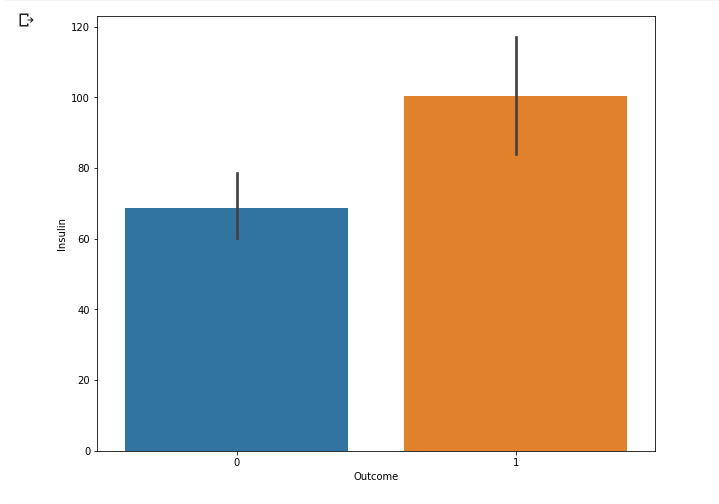
- 막대 위에 검정색 선은 데이터 일부를 샘플링해 95%의 신뢰구간을 나타내는 것임.
- 막대의 끝을 기준으로 형성된 것으로 보아 평균값의 95% 신뢰구간을 의미하는 듯.
- 같은 유의수준이라 했을 때 신뢰구간이 짧은 것이 보다 정확한 것. 같은 유의수준일 때 신뢰구간이 길다면 신뢰를 할 수 없다고 보면 됨. 왜냐 -inf, inf로 두면 100% 이므로.
- 여담으로 95%의 신뢰구간 할 때 이 95%는 95%의 확률로 이 구간안에 포함된다가 아니라, 신뢰구간을 100번 구했을 때 신뢰구간 안에 모수가 있는 경우가 95번이라는 의미임.
plt.figure(figsize = (10, 8))
sns.barplot(data = df, x = 'Pregnancies', y = 'Outcome')
plt.show()
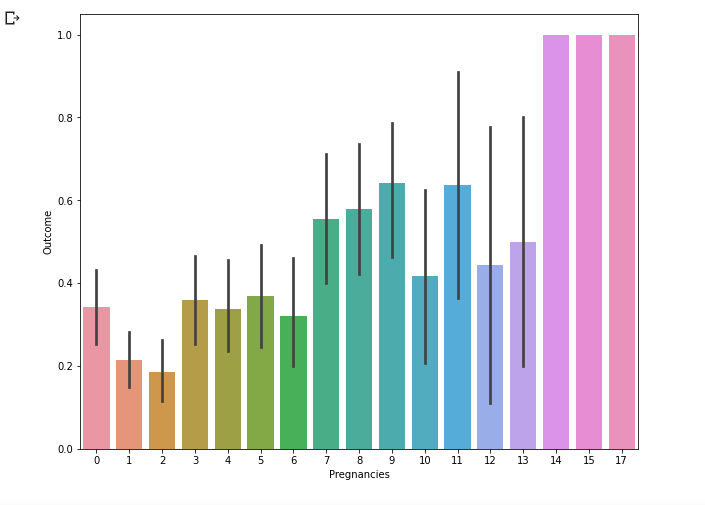
- 임신 횟수에 따른 발병율 시각화. 평균 값을 시각화 한 것 이지만 0, 1 이므로 비율이 됨.
plt.figure(figsize = (10, 8))
sns.barplot(data = df, x = 'Pregnancies', y = 'Glucose', hue = 'Outcome')
plt.show()
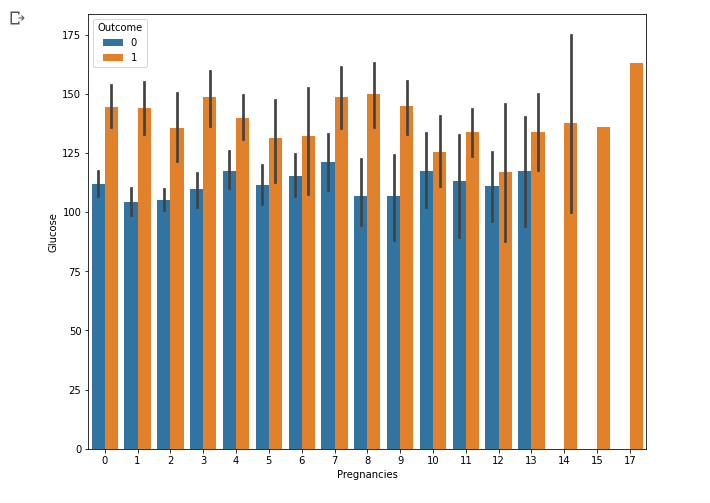
- x, y뿐만 아니라 hue도 이용해 총 세가지의 변수를 시각화 한 것.
- 임신 횟수에 따른 글루코스 수치를 보는데 이 때 당뇨병 여부까지 포함시킨 것.
- 당뇨환자가 글루코스 수치가 더 높음.
plt.figure(figsize = (10, 8))
sns.barplot(data = df, x = 'Pregnancies', y = 'BMI', hue = 'Outcome')
plt.show()
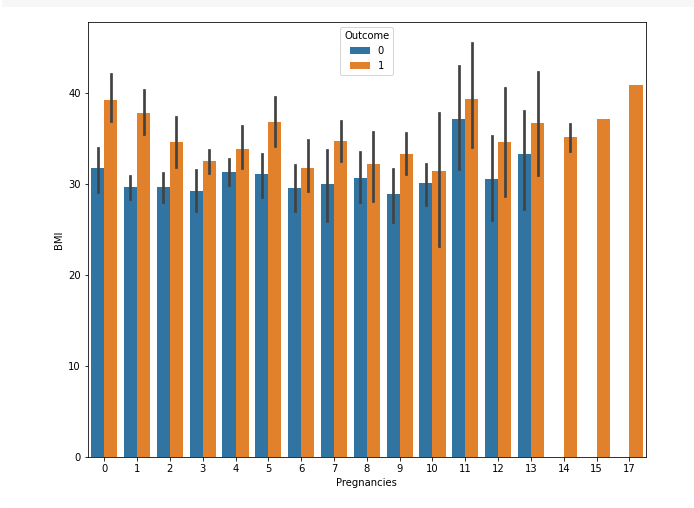
- 임신 횟수, BMI, 발병여부 총 세가지를 시각화.
- 우선 당뇨병 환자가 BMI가 더 높고 임신 횟수에 따른 BMI의 차이는 파악하기가 쉽지 않다.
- 그리고 저 범례는 따로 위치를 지정하지 않으면 알아서 빈 공간에 plotting
plt.figure(figsize = (10, 8))
sns.barplot(data = df, x = 'Pregnancies', y = 'Insulin', hue = 'Outcome')
plt.show()
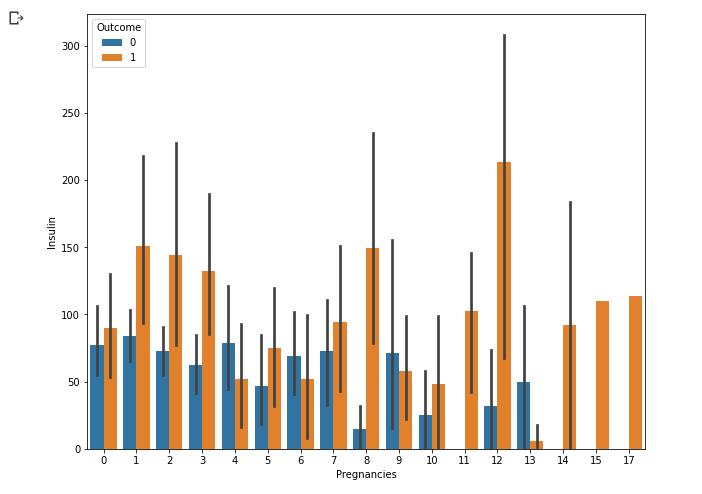
- 임신 횟수, 인슐린 수치, 발병여부를 시각화
- 당뇨병 환자의 평균 인슐린 수치가 더 높음. 실제로 당뇨 환자들은 인슐린 수치를 낮추기 위해 주사를 맞음
- 신뢰구간이 유독 긴 부분이 보이는데 95%의 신뢰구간을 구할 때 평균 +- t(n, 0.025)*sd.
- +- 뒷 부분의 값이 크다면 구간의 길이가 길어짐. t값은 그렇게 커지진 않으므로 sd 값이 중요.
- n이 커질수록 sd는 0에 가깝게 안정화 됨. 임신 횟수는 커질수록 그 빈도수가 적음. 편차 자체가 원래 클 수도 있지만 표본의 개수 자체가 너무 적어서 이렇게 긴 신뢰구간이 형성된 것이라 생각.
plt.figure(figsize = (10, 8))
sns.barplot(data = df[df['Insulin'] >0], x = 'Pregnancies', y = 'Insulin', hue = 'Outcome')
plt.show()
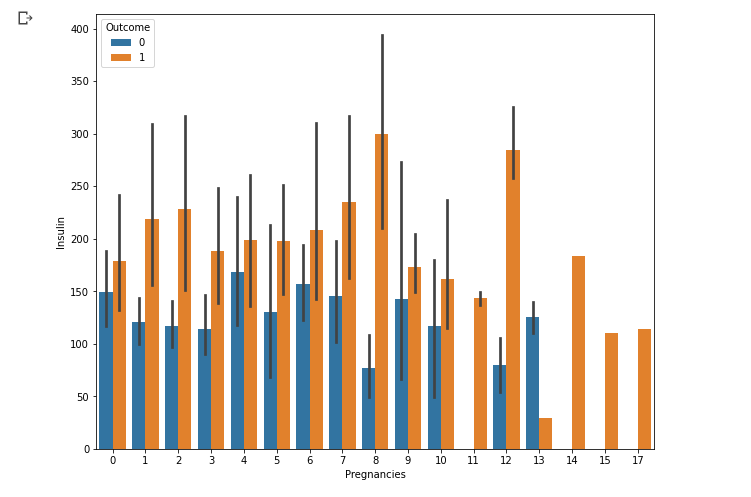
- 인슐린 수치가 0 이상인 것들에 대해서만 임신 횟수, 인슐린 수치, 발병여부를 시각화
- 신뢰구간의 길이가 대체적으로 줄어들었는데 범위를 제한했으므로 편차가 줄어들 수 밖에 없음.
- 0보다 작은 값들이 있었던 것 같은데 이는 이상치가 아닐지...?
- 신뢰구간이 형성되지 않은 막대들의 경우에는 빈도수가 1일 것으로 파악. 표본이 하나 뿐이라면 sd = mean = x 이므로.
boxplot
- 명목형 변수와 수치형 변수간의 시각화를 할 때도 사용
plt.figure(figsize = (10, 8))
sns.boxplot(data = df, x = 'Pregnancies', y = 'Insulin', hue = 'Outcome')
plt.show()
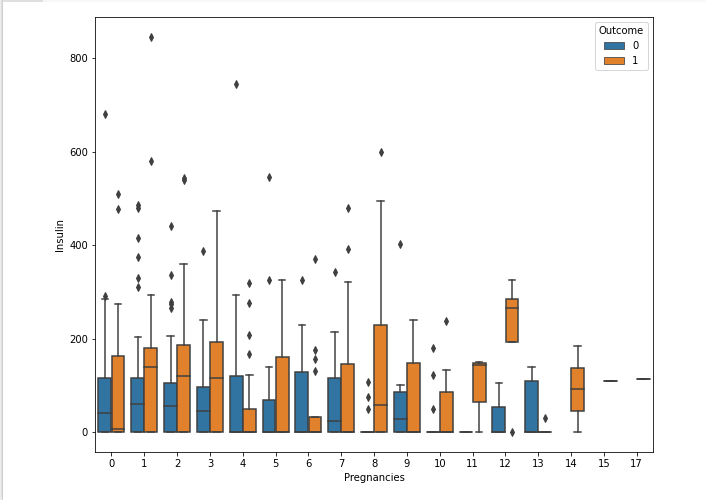
- 임신 횟수, 인슐린 수치, 발병 여부 세 가지 시각화
- 당뇨병 환자일수록 인슐린 수치가 더 높아지는 것은 확인
- boxplot은 q3-q1을 높이로 하는 막대를 그리고 위 아래로 whisker가 있음
- 위 아래 whisker를 벗어나는 값들은 이상치로 판단.
- 다이아몬드 모양의 점이 이상치가 되는 것
- 위에서 그렸던 barplot은 인슐린 수치의 평균값을 높이로 갖는 막대를 그렸다면
boxplot은 임신 횟수별 인슐린 수치의 모든 값들을 다 나타낸 것
- 연속형 변수가 어떤 형태의 분포일지 파악이 얼추 가능
- 막대가 길다면 -> q3-q1이 길다는 것이고 전체적으로 완만한 분포를 띈다고 생각.
plt.figure(figsize = (10, 8))
sns.boxplot(data = df[df['Insulin'] > 0], x = 'Pregnancies', y = 'Insulin', hue = 'Outcome')
plt.show()
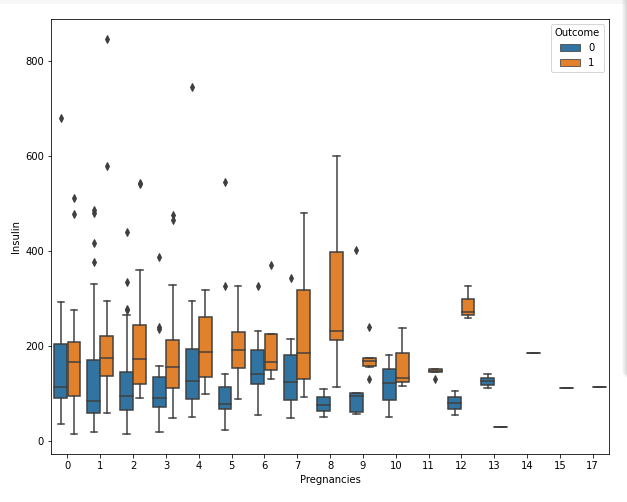
- 0보다 작은 값들을 제외하고 box plot을 그려보니 ylim이 0이상이었는지 아래 whisker가 제대로 보이지 않았던게 제대로 보임
- 당뇨병 환자의 경우 임신 횟수가 8까지는 인슐린 수치의 증가하는 형태로 파악
- 근데 인슐린 수치의 증가가 임신 횟수에 의한 영향인지, 당뇨병 때문인지는 파악 못함.
- 회귀 분석을 통해 coef을 보고 영향도를 파악하거나 분산 분석을 통해 파악
vilolinplot
- boxplot과 유사한데 boxplot은 특정 구간의 분포의 형태 -> 많이 몰려있는지 아닌지는 파악이 어려움
- vilolin plot은 분포를 파악가능하게 굴곡있는 형태로 시각화 해줌
plt.figure(figsize = (10, 8))
sns.violinplot(data = df[df['Insulin'] > 0], x = 'Pregnancies', y = 'Insulin', hue = 'Outcome', split = True)
plt.show()
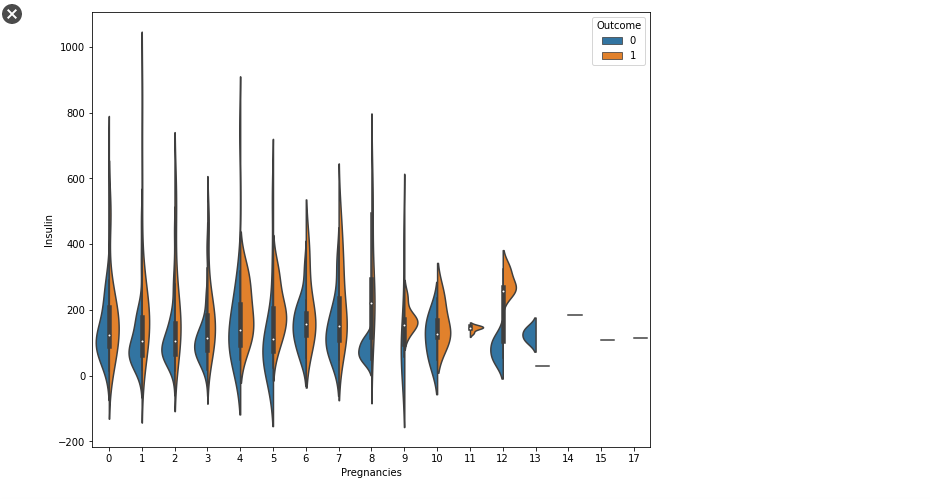
- hue의 카테고리에 따라 pdf를 그려주고 두 개를 붙여놔 발병 여부에 따른 분포 비교도 가능하고 임신 횟수에 따른 분포 비교도 가능
- split을 True로 주어 좌우로 데이터를 나눠서 그려준 것.
swarmplot
- 카테고리 변수에 따른 연속형 변수의 분포를 파악할 수 있고
- 각각의 값들을 점으로 시각화. 카테고리별 산점도라고 생각
plt.figure(figsize = (10, 8))
sns.swarmplot(data = df[df['Insulin'] > 0], x = 'Pregnancies', y = 'Insulin', hue = 'Outcome')
plt.show()
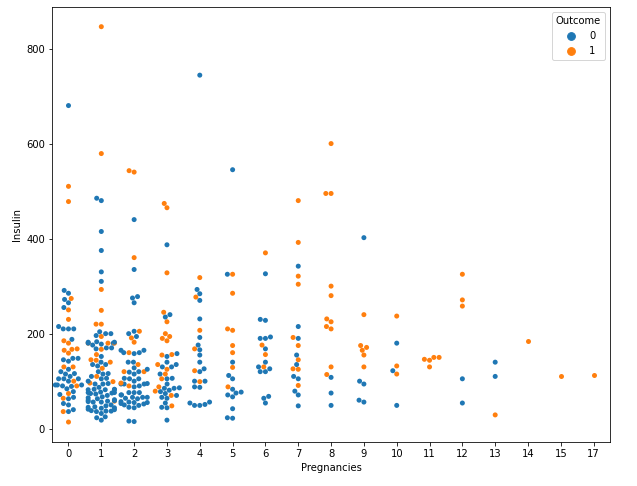
- 특정 부분에서 많이 분포하고 있으면 횡으로 더 넓게 포인팅 해줌.
- 발병 여부에 따라 임신횟수별로 인슐린 수치들을 비교
- violin plot은 이 점들을 pdf로 표현한 것이라 생각.









































