Gaussian Process는 함수들의 사전분포에 대한 것이다. 이때, 함수들의 사전분포를 정하는 이유는 주어진 데이터로부터 함수를 추정하는 과정에서 특정 조건을 부여하여 추론 혹은 예측 과정을 더 용이하게 하기 위함이다. 이 과정에서 Gaussian, especially Multivariate Gaussian distribution을 사용하는 이유는 조건부 확률분포의 정규성 등 Gaussian distribution의 좋은 성질이 있기 때문이다.
Bayesian Approach for Linear Regression
Weight-Space View
다음과 같은 Linear Model을 고려하자.
베이지안적 관점에서, parameter 에 대한 사전분포를 다음과 같이 정규분포 형태로 줄 수 있다.
그러면, 베이즈 규칙
에 의해 posterior distribution을 다음과 같이 구할 수 있다.
즉, posterior distribution 역시 정규분포를 따르는 것을 확인할 수 있고, 이로부터 posterior distribution의 평균이 의 MAP(maximum a posteriori) estimate임을 알 수 있다.
Prediction
앞서 구한 사후분포를 바탕으로 새로운 test input data 에 대한 predictive distribution을 구하기 위해서는 다음과 같이 posterior에 대해 가능한 모든 선형모형의 평균을 취하는 방식을 사용한다.
Predictive distribution 역시 마찬가지로 정규분포를 따른다는 것을 확인할 수 있다.
Projection into Feature Space
일반적인 선형모형은 설명변수와 반응변수와의 관계를 오직 선형관계로만 파악해내기 때문에, 복잡한 구조를 갖는 데이터 형태에 대해서는 잘 적용되지 못한다는 한계점이 있다. 따라서 이를 극복하기 위해 Polynomial regression과 같은 분석방법을 채택하기도 하는데, 이러한 방법들의 공통점은 선형모형의 사용과정에서 우선 Input data를 high-dimensional feature space에 project한다는 것이다. 예를 들면, polynomial regression의 경우 데이터를 에 사영시킨 것이다. 이 경우 새로운 선형모형은 다음과 같다.
이를 이용하면 predictive distribution은 다음과 같이 주어진다.
이 과정에서 등장하는 내적 형태의 함수 를 kernel이라 정의한다.
Function-space View
앞서 살펴본 방식은 선형모형의 모수 에 대한 사전분포를 바탕으로 사후분포를 구하고, 이를 이용한 예측 과정이었다. 반면, 다음과 같이 모형을 가정하는 대신에, 함수의 (사전)확률분포인 Gaussian Process를 고려하여 새로운 데이터에 대한 예측 모형을 만들 수 있다(는 일반적으로 0).
Gaussian process는 정의에 의해 임의의 input point들의 finite subset 에 대한 evaluation 이 Multivariate gaussian distribution을 따르므로, 다음과 같이 주어진 Gaussian process로 부터 random Gaussian vector를 생성할 수 있다.
여기서 공분산행렬은 임의의 input data의 부분집합 에 대한 kernel matrix를 의미한다(input data가 feature space보다 높은 차원을 가지면 singular matrix가 됨).
Prediction
주어진 사전분포 로부터 random function을 생성하는 것에 그쳐서는 회귀분석을 진행할 수 없다. 추가적으로 실제 관측된 데이터가 주는 정보들을 해당 사전분포에 결합시키는 과정이 필요하다. 관측치 이 주어질 때, 모형
에 대해 를 추정하고 이를 바탕으로 새로운 test data 에 대한 output vector 를 예측해야 하는 문제를 생각해보자. 우선 함수 는 Gaussian process 를 따른다고 설정하면 관측치 에 대한 evaluation은 random gaussian vector
가 된다. 이로부터 관측치 의 분포는
이 된다. 그러면 와 prediction output vector 의 결합분포는
와 같다. 정규분포의 조건부확률분포 공식을 이용하면 다음과 같이 Gaussian process regression equation을 얻을 수 있다.
Prediction for single point
앞서 구한 prediction equation을 하나의 에 대한 예측값을 구하는 과정에 사용해보면 다음과 같다.
여기서 는 test point 와 n개의 training point 간의 커널함수값으로 이루어진 n-dimensional 벡터이다. 이때 예측치의 평균함수가 의 선형결합으로 이루어져있음을 확인할 수 있는데, 실제로는 다음과 같은 n개의 kernel function의 선형결합 꼴로 나타낼 수 있다.
Algorithm
# RBF kernel
def kernel(a, b, param):
sqdist = np.sum(a**2, 1).reshape(-1, 1) + np.sum(b**2, 1) - 2*np.dot(a, b.T)
return np.exp(-0.5 * (1/param) * sqdist)
# noise level
noise = 0.1
# Computing Algorithm
L = np.linalg.cholesky(K + noise*np.eye(N))
alpha = np.linalg.solve(L.T, np.linalg.solve(L, y))
# test points
Xtest = np.linspace(-5, 15, 100).reshape(-1, 1)
Ktest = kernel(X.reshape(-1, 1), Xtest, param)
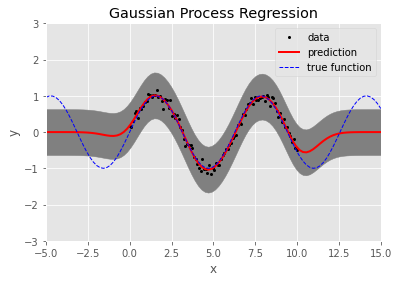
ytest = np.dot(Ktest.T, alpha) # prediction values100개의 임의 생성 데이터에 대한 결과는 다음과 같다.
References
- C. E. Rasmussen - Gaussian Process for Machine Learning
- Code on GIthub : https://github.com/ddangchani/Velog/blob/main/Statistical%20Learning/GP%20Regression.ipynb