MOLI : Multi-omics Late Interation with deep neural networks for drug response prediction
논문리뷰

Hossein Sharifi-Noghabi, Olga Zolotareva, Colin C. Collins,* and Martin Ester
ISBM/ECCB 2019
🐶 https://academic.oup.com/bioinformatics/article/35/14/i501/5529255
Multi-omics 란?
- 유전체(Genome), 전사체(Transcriptome), 단백체(Proteome), 대사체(Metabolome) 등
-ome으로 끝나는 분야들을 의미 - 하나의 분야에 대해 연구하고 모델링 하는 것은 Single-omics 연구라고 한다면 동시에 다양한 omics data를 활용하는 연구를 Multi-omics 연구라 함
- Multi-omics data를 한 번에 분석하면서 보다 정확하고 포괄적인 생물학적 정보를 얻을 수 있음
Abstract
motivation
- gene expression은 drug response prediction에 가장 유용한 data
- 최근 연구에 따르면 omics를 통합하면 예측 정확도가 향상될 수 있다는 것을 보이고 있음
- Multi-omics 접근법이 drug response와 clinical relevance를 향상시킬 것이라 예상
Introduction
-
Drug response 연구에서 중요한 challenge는 clinical utility. 즉, Translatability
-
Translatability 달성을 위해서는 in vivo 에 대해 학습해야 하지만 in vivo(체내) dataset에는 drug response가 기록된 환자의 정보가 작음
-
in silico(실험실 데이터) drug response prediction의 경우, *in vitro(체외) data에 대해 우수한 성능을 가진 모델이 in vivo data에도 우수한 성능을 가져야 함
*in vitro(체외) data 의 경우 쥐 데이터(PDX) 등이 있다. (사람에게 직접 실험하지 못하니 쥐에게 실험하여 결과를 정리한 데이터 셋)
-
Multi-omics data는 동일한 샘플에서 다른 관점을 제공하며 생물학적 과정을 더 잘 특성화 함을 보장
-
어떻게 서로 다른 data types를 통합?
Multi-omics data 통합 방법 2가지
-
Early intergration
- 샘플에 대해 사용할 수 있는 모든 data type을 연결
- Auto-Encoder와 같은 feature learning method를 적용하여 integrated representation 생성
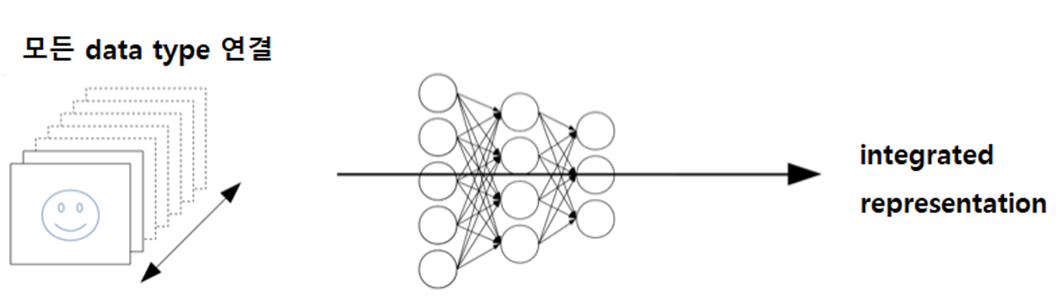
- 단점 :
각 omics type의 고유한 분포 무시로 인한 정보 손실
적절한 normalize 필요
input data의 dimension 증가
- 단점 :
-
Late integration
- 각 omics data type에 대해 별도로 학습
- 해당 feature는 하나의 unified representation으로 통합되어 classifier 또는 regressor의 입력으로 사용
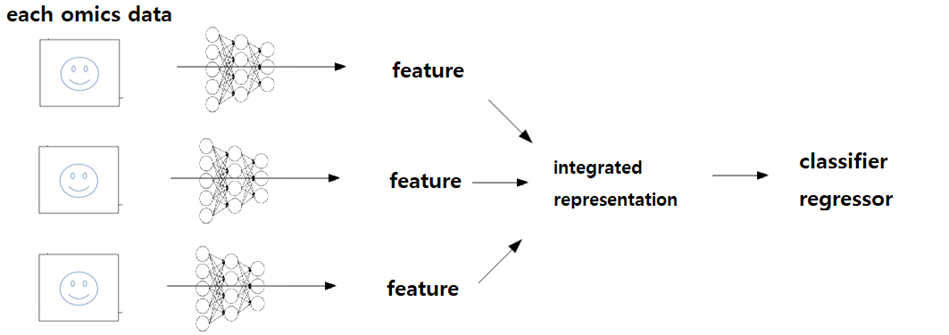
- 장점
- 각omics type의 고유한 분포 유지
- 각 omics type에 single-normalization 적용 가능
- input data의 dimentionality 증가 X
- 장점
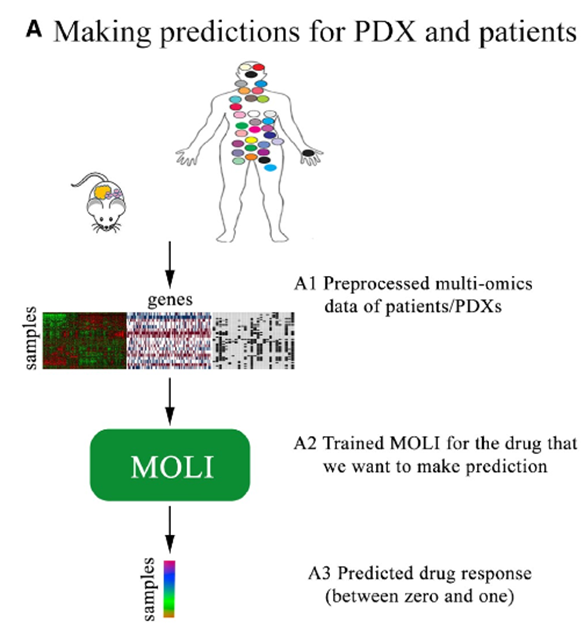
- MOLI : somatic mutation, copy number abberation, gene expression data를 입력
- 주어진 약물에 대한 반응 예측
- Triplet loss + BCE loss = cost function을 사용한 최초의 end-to-end late integration method with deep neural networks
Materials and methods
structure
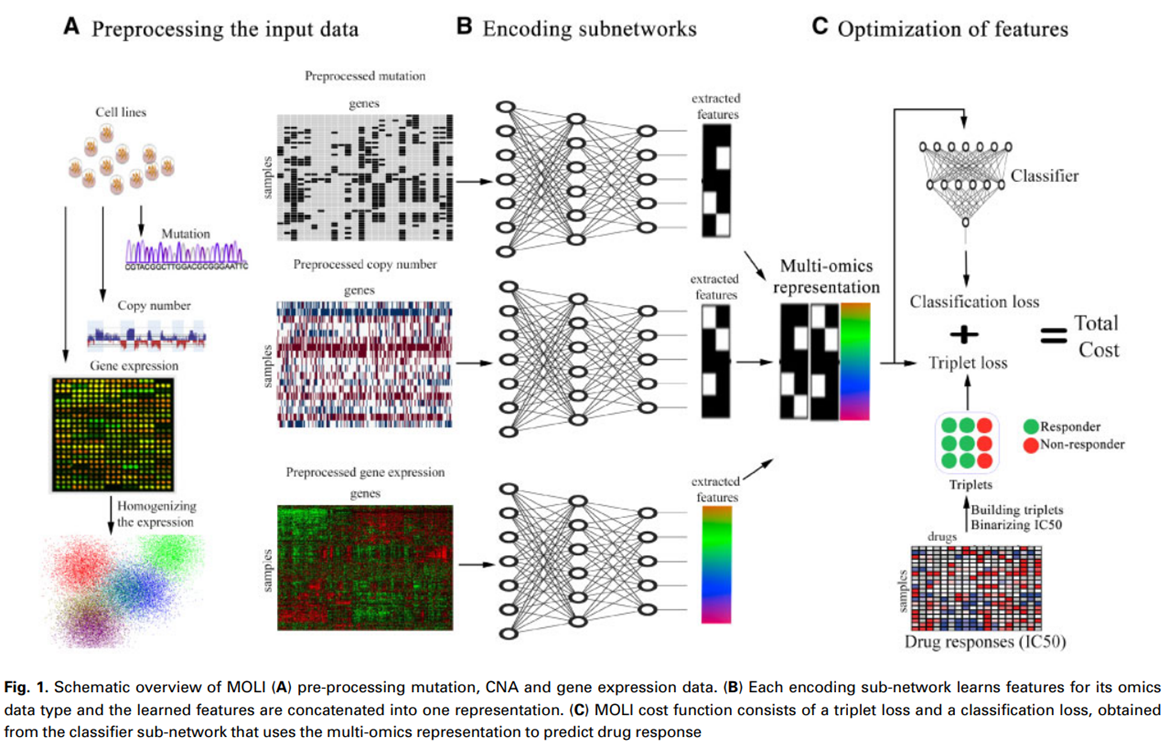
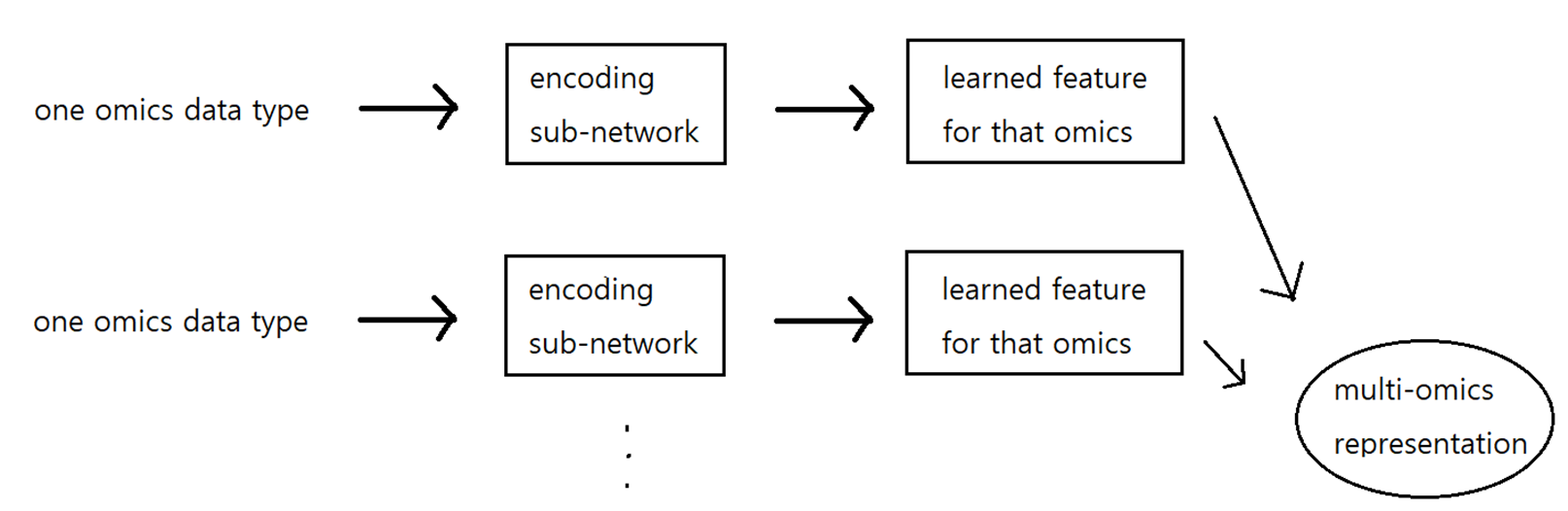
A : 데이터 전처리 후 MOLI의 입력으로 사용
B : 각 입력 (mutation, copy number, gene expression)에 대해 sub-network를 만들고 각각의 feature 추출 -> multi-omics representation으로 integration
C : 통합된 representation을 Classifier로 입력 후 cost function을 이용해 drug response
Learning features by encodeing sub-network
- 각 omics data의 feature를 학습하기 위해 input space -> feature space로 mapping하는 encoding feed forward sub-network를 가짐
- 각 encoding sub-network는 ReLU를 사용하는 FC network
- Dropout, bath normalization 사용
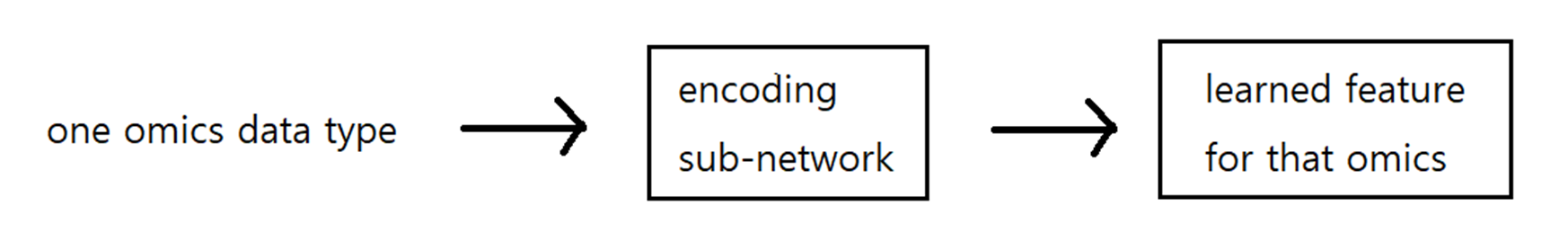
Integrating learned features by late integration
- integrated representation은 l2 normalization layer를 통해 모든 가중치를 모델에 고르게 반영
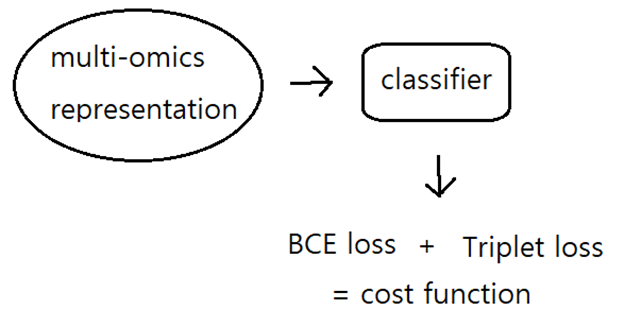
Optimizing the learend features by the combined cost function
- 학습된 feature는 drug response predict classifier에 사용
- 따라서 MOLI의 마지막 layer는 sigmoid 사용
- Dropout을 사용하여 normalization, weight decay를 사용하여 모델 복잡도 감소
- 여기에 Triplet loss를 추가하여 cost function 재구성
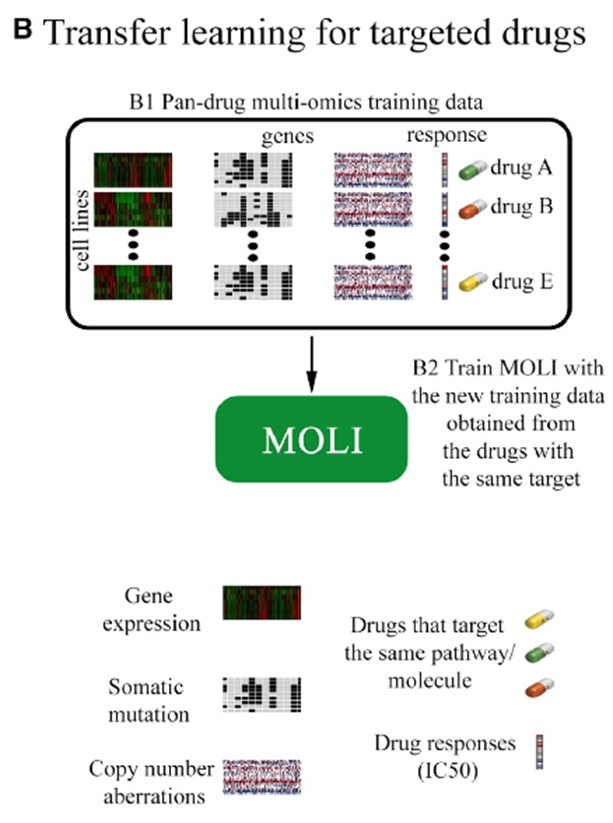
Transfer learning for targeted drugs
- MOLI는 drug-specific data 대신 pan-drug data에 대해 학습
- 해당 접근법은 선별된 cell line과 얻어진 반응이 pan-drug data와 유사하지만 동일하지 않기에 dataset의 크기를 증가시킴
Predicting drug response for TCGA patients
-
Pan-drug input에서 학습된 모델을 사용하여 EGFR 억제제에 대한 약물 반응이 기록되지 않은 여러 TCGA dataset 환자에 대한 약물 반응 예측
-
EGFR 억제제는 EGFR 경로를 타겟으로 하기에 해당 경로의 gene expression 상태가 예측된 약물 반응과 상관관계가 있을 것으로 기대
-
상관관계를 연구하기 위해 예측된 반응과 발현 수준 사이의 multiple linear regression을 사용
-
각 유전자에 대한 p-value를 얻고 Bonferroni 보정 사용하여 다중 비교
datasets
-
GDSC cell lines dataset (Iorio et al., 2016) (실험실 데이터)
-
PDX Encyclopedia dataset (Gao et al., 2015) (쥐 데이터)
-
TCGA patients with the drug response available in their records (Ding et al., 2016) (실제 환자 데이터)
-
TCGA patients without the drug response (Weinstein et al., 2013) (실제 환자 데이터, 약물 반응 X)
Gene expression profile
- 서로 다른 플랫폼에서 발현 프로파일을 비교하기 위해 유전자 발현을 표준화하고 pairwise homogenization 절차를 수행 : gene expression 측정 방법이 달라서 표준화 하는 작업 필요
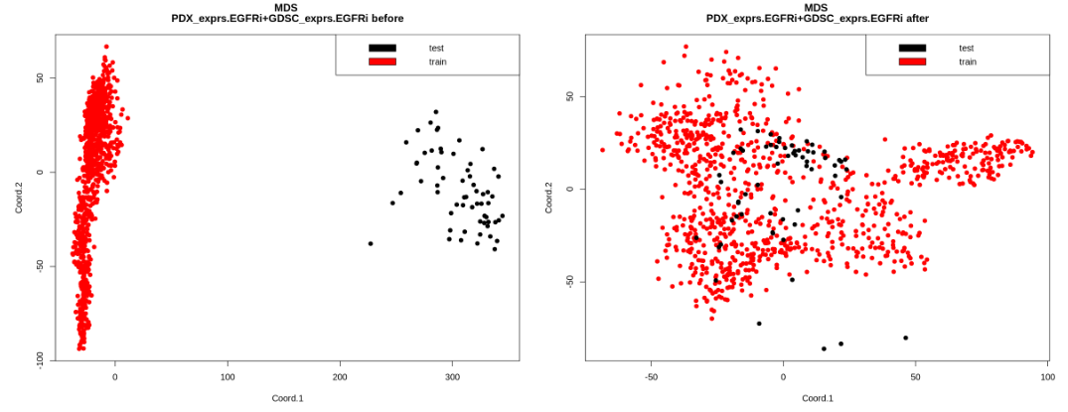
- 위 사진처럼 진행
Somatic copy number profile
- TCGA 데이터셋에서 신뢰성이 낮은 세그먼트를 제거하고 유전자가 겹치는 세그먼트의 강도 로그 비율에 해당하는 값을 할당
- 유전자가 둘 이상의 세그먼트와 겹치는 경우 가장 extreme한 log-ratio값을 유지
- TCGA와 달리, GDSC와 PDX 데이터셋은 total copy number의 추정치 제공
- 이러한 data를 TCGA와 비교하기 위해 각 유전자에 대해 해당 샘플에서 복사 중성 상태의 ploidy로 나눈 복사수의 로그를 계산합니다. 마지막으로, 모든 데이터셋에서 유전자 수준 복사수 추정치를 바이너 라이즈하고 복사 중성 유전자에는 0을, 삭제 또는 증폭이 겹치는 모든 유전자에는 1을 할당합니다.
Somatic point mutation
- Somatic mutation을 가진 유전자에게는 1 할당, 다른 모든 유전자에 0 할당
잘 모르겠음
이런 mutation을 어떻게 처리했다.. 그런 내용
Result
Experimental design
-
MOLI가 PDX 및 환자 데이터에서 예측 AUC 에서 단일 omics 및 초기 통합 기준선을 능가하는가?
-
표적 약물에 대해서도 transfer learning이 잘 작동하는가?
즉, MOLI가 pan-drug data로 학습되었을 때 drug-specific data로 학습된 MOLI를 능가하는가? -
표적 약물에 대해 MOLI의 예측이 해당 약물의 표적과 연관이 있는가?
-
Docetaxel, Cisplatin, Gemcitabine, Paclitaxel, Erlotinib 및 Cetuximab에 대한 PDX/환자 multi-omics data가 필요하기에 해당 약물로 검사된 GDSC cell line을 대상으로 MOLI 학습
-
모든 baseline을 동일 약물에 대해 학습하고 MOLI와 예측 AUC 비교
MOLI
VS
1. Early integration via deep neural networks, Ding et al. (2018)
-
Early integration via non-negative matrix factorization (NMF), (Cichocki and Phan, 2009; Févotte and Idier, 2011)
-
The single-omics (gene expression) ridge regression method, Geeleher et al. (2014)
-
An ordinary feed forward network with classification loss trained on the expression data
-
A version of MOLI trained only on the gene expression data
-
MOLI - only using classification loss (To test whether the triplet loss contributes to improve the performance)
-
외부 검증에 사용된 PDX dataset 에서 EGFR 경로를 타겟으로 하는 약물에 초점을 맞춰 *Transfer learning 연구
-
이러한 약물에 대한 multi-omics data를 사용하여 대규모 train set 구축
-
Pan-drug data로 학습한 MOLI와 specific-drug로 학습한 MOLI 비교
*Transfer learning
- Transfer learning은 하나의 문제를 해결하고 이와 다르면서 관련된 문제에 적용하는 동안 얻은 지식을 저장하는데 집중하는 기계 학습의 연구 문제이다. 예를 들어 자동차를 인식하기 위해 학습하는 동안 얻은 지식은 트럭 인식을 시도할 때 적용할 수 있다.
- MOLI에서의 Transfer learning은 in vitro -> in vivo 로의 Transfer
-
AUC를 기준으로 DNN의 hyper parameter를 조정하기 위해 5-fold 교차 검증을 사용
-
hyper parameter : hidden layers, learning rates, mini-batxh size, weight decay, dropout rate,epochs, margin, regularization (only for the triplet loss)
-
해당 약물에 대한 전체 dataset에서 얻은 hyper parameter로 재학습
-
모든 DNN에 Adagrad를 사용하여 hyper parameter 최적화
Multi-omics interation by MOLI improves the drug response performance
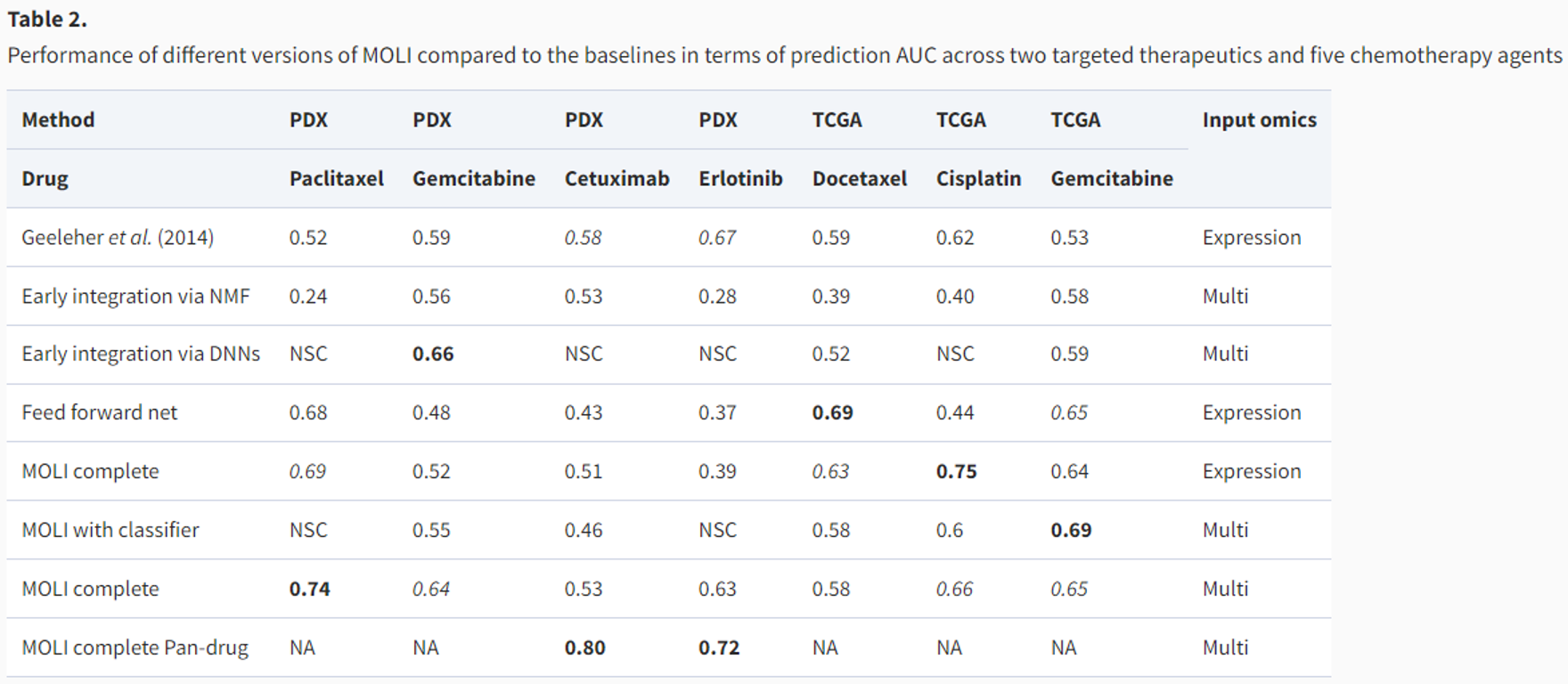
Transfer learning for targeted grugs improves performance significantly
-
Target drug의 경우, pan-drug multi-omics 에 대해 학습된 MOLI가 specific-drug 에 대해 학습한 MOLI 보다 나은 performance
-
Pan-drug MOLI : Cetuximab의 경우 0.8, Erlotinib의 경우 0.72의 AUC를 달성
--> Transfer learning 이 표적 약물에 대한 예측 성능을 향상시킬 수 있음을 시사
Predictions for TCGA patients by MOLI have associations with EGFR genes
-
TCGA에서 약물 반응이 없는 multi-omics data에 MOLI를 적용하고 반응 예측
-
Multiple linear regression에서 얻어진 p-values에 따르면 EGFR 유전자와 MOLI에 의해 예측된 반응 사이에는 강한 연관성이 있음을 보여줌
-
유방암의 경우, AP2A1(P = 0.007), CALM2(P = 0.01), CLTA(P = 0.0002), EGFR(P = 1×10-510-5), PIK3CA(P = 0.007), UBA52(P = 3×10-6-6)
-
전립선암의 경우, AKT1(P = 0.02), CDK1(P = 0.01), RICTOR(P = 0.0002), CREB1(P = 0.02) 및 CSK(P = 0.01)
-
신장암에서 EGFR(P = 0.04), 폐암에서 CDC42(P = 0.04), EGFR(P = 3×10-5) 및 PRKAR2A(P = 0.01) 유전자의 발현은 예측 반응과 유의미한 연관성을 관찰
-
그러나 방광암과 췌장암의 경우 유의한 연관성은 관찰되지 않았다.
Discussion
-
Drug response prediction을 위해 DNN 기반의 MOLI 제안
-
Somatic mutation, CNA, gene expression data를 통합하고 drug response prediction
-
Cost function을 사용하는 DNN, 최초의 end-to-end 방식
-
Single-omics와 DNN 기반 early integration multi-omics method 보다 높은 성능
-
표적 약물에 대한 transfer learning이 specific-drug 에서 학습보다 높은 성능
-
MOLI의 예측이 EGFR 경로의 일부 유전자 발현과 유의미한 연관성을 가짐
Conclusion
-
MOLI는 AUC및 PR curve에서 single-omics 예측 성능 능가
-
Early integration을 사용한 DNN 능가
-
Cost function(Triplet loss + BCE loss)이 결합된 MOLI가 single and multi-omics baseline 능가
-
Transfer learning을 사용하여 pan-drug에서 학습된 MOLI는 drug-specific에서 학습된 MOLI 능가
-
MOLI의 생물학적 중요성; MOLI가 예측한 반응이 TCGA 환자에 대한 EGFR 경로의 수 많은 유전자 발현 레벨과 유의미한 연관성을 가짐
논문을 읽고 나서
대학원 입학 후 처음으로 읽은 논문이라 애정이 깊다. (사실 뻥임) 해당 논문은 multi-omics를 처음으로 통합한 논문으로 인용수가 높지만 drug 마다 prediction을 해야 되어서 코드를 뜯어보면 알겠지만 약물 별로 코드가 구성되어 있다. 게보린 반응, 타이레놀 반응 처럼... 하지만 처음으로 omics data 를 통합하였다는 것이 의미가 많다고 한다.
현재 나온 다른 논문들은 pan-drug를 사용한 논문도 많고 moli가 기초를 잡았다,, 뭐 그런 넉힘.. 그리고 transger learning 이라는 뜻을 해석하기가 어려웠다. CV에서는 대량 데이터로 학습 후 원하는 데이터로 학습하면 굳굳이다 이런 의미였는데 moli에서는 A drug 를 통해 B drug에도 적용하고 .. 체외 데이터로 학습 후 체내 데이터에 적용하고 .. 이런 복잡한 의미를 가지고 있었다. 마치 너구리로 짜빠구리를 만드는 것이 transfer .. 이런 ... 느낌....

