1. Volcano Plot 이란 ?
Volcano Plot이란 수많은 변수들 중 통계적으로 유의미한 변화를 보이는 데이터를 직관적으로 식별하기 위해 사용하는 산점도(Scatter plot)의 일종이다. 이름처럼 화산이 폭발하는 듯한 모양을 띄고있다.
주로 두 그룹(ex. 실험군 vs 대조군)간의 유전자 발현량, 단백질 수치 등의 차이를 분석할 때 사용된다.
수천, 수만 개에 달하는 데이터 포인트 중에서 어떤 유전자가 의미 있는 변화를 보였는지 한눈에 알려주는 아주 강력한 도구이다.
- X축 : Log2FoldChange
- X축에는Log2FoldChange를 사용하는데, 데이터의 분포를 대칭으로 만들어주고, 변화량이 큰 값들의 차이를 시각적으로 잘 표현할 수 있다.
- 결과적으로 X축의 절댓값이 클수록 두 그룹 간의 변화량 차이가 크다는 것을 의미한다.
- Y축 : -Log10P-value
- 일반적으로 P-value는 0에 가까운 아주 작은 값을 가지기 때문에, -log10을 취해 값의 분포를 넓혀준다.- 결과적으로 Y축의 값이 클수록 두 그룹 간의 차이가 통계적으로 유의미하다는 것을 의미한다.
2. R로 구현해보자
library(DESeq2)
library(airway)
library(ggplot2)
library(ggrepel)
library(dplyr)
# 2. 데이터 준비 및 차등 발현 분석 (Analysis)
data(airway)
# DESeq2 분석을 위한 데이터 객체를 생성
dds <- DESeqDataSet(airway, design = ~ cell + dex)
# 차등 발현 분석을 실행
dds <- DESeq(dds)
# 분석 결과를 'trt'(처리군) vs 'untrt'(대조군) 기준으로 추출
res <- results(dds, contrast = c("dex", "trt", "untrt"))
# 3. 결과 데이터프레임 가공 (Data Wrangling)
res_df <- as.data.frame(res)
res_df <- na.omit(res_df)
res_df$gene <- rownames(res_df)
# 기준: |log2FoldChange| > 1 그리고 padj < 0.05
res_df <- res_df %>%
mutate(change = case_when(
log2FoldChange > 1 & padj < 0.05 ~ "Upregulated",
log2FoldChange < -1 & padj < 0.05 ~ "Downregulated",
TRUE ~ "Not Significant"
))
# 4. 최종 시각화 (Visualization)
# 플롯에 라벨링할 상위 10개 유의 유전자를 선택
top_genes <- res_df %>%
filter(change != "Not Significant") %>%
arrange(padj) %>%
head(10)
# ggplot2를 사용하여 Volcano Plot을 생성
ggplot(data = res_df, aes(x = log2FoldChange, y = -log10(padj), color = change)) +
# 점 그래프 (Scatter plot)
geom_point(alpha = 0.6, size = 2) +
# 색상 직접 지정
scale_color_manual(name = "Expression Change",
values = c("Upregulated" = "#E64B35FF", "Downregulated" = "#0073C2FF", "Not Significant" = "grey")) +
# 유의성 기준선 추가 (점선)
geom_vline(xintercept = c(-1, 1), linetype = "dashed") +
geom_hline(yintercept = -log10(0.05), linetype = "dashed") +
# ggrepel을 이용해 겹치지 않게 유전자 라벨 추가
geom_text_repel(data = top_genes,
aes(label = gene),
box.padding = 0.5,
max.overlaps = Inf) +
# 전체적인 테마 및 제목, 축 이름 설정
theme_bw(base_size = 14) +
labs(title = "Differential Gene Expression",
subtitle = "Dexamethasone Treatment vs. Control in Airway Cells",
x = expression(paste("Log"[2], " Fold Change")),
y = expression(paste("-Log"[10], " Adjusted P-value")))3. 결과 분석
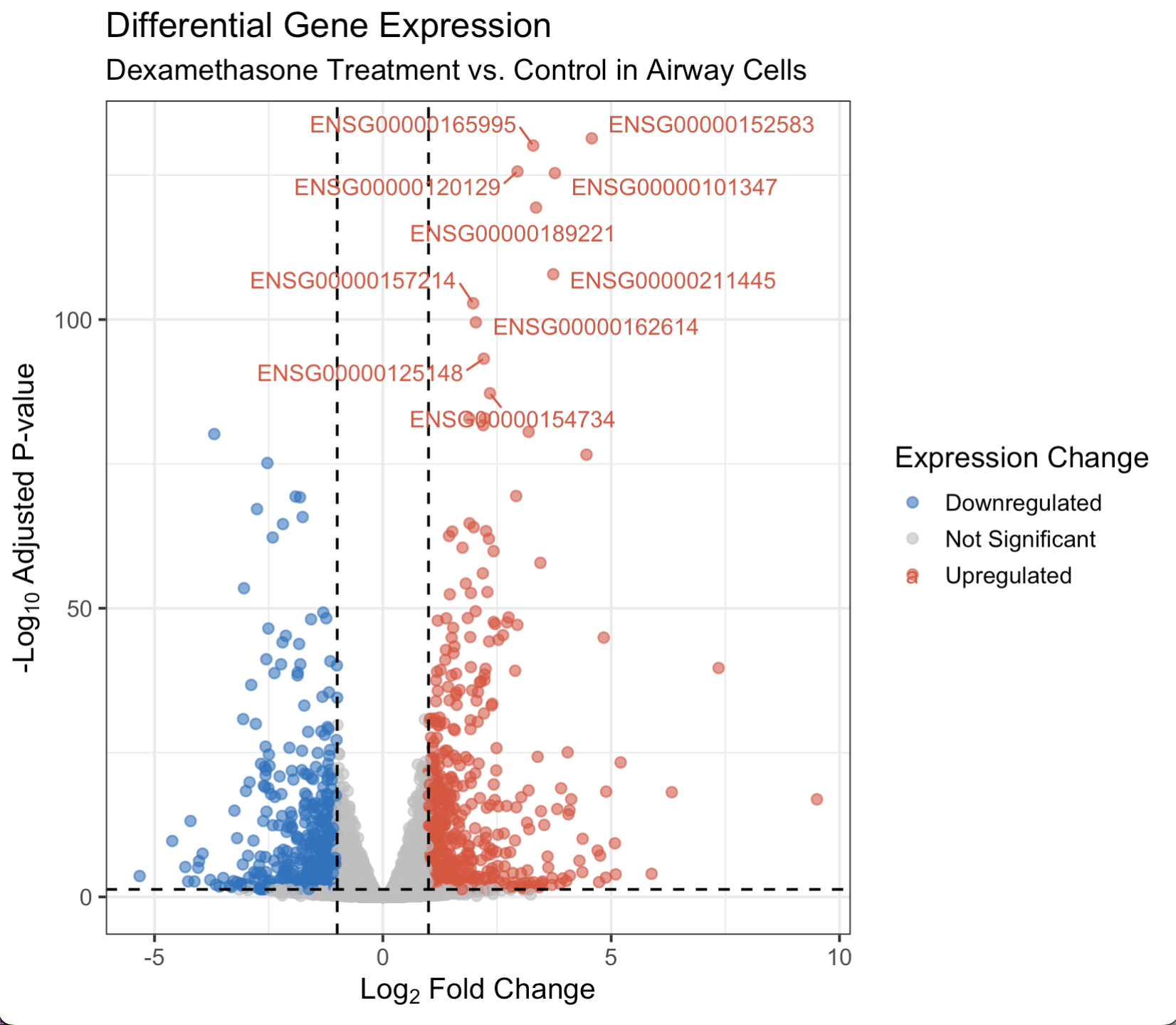
- 빨간점 : 통계적으로 유의미하게 발현이 증가한 유전자들. 이들은 약물 처리의 직접적인 타겟이거나 그로 인해 활성화된 중요한 바이어 마커 후보가 될 수 있다.
- 파란점 : 통계적으로 유의미하게 발현이 감소한 유전자들. 약물에 의해 억제된 유전자들로, 질병과 관련된 경로를 차단하는 역할을 할 수 있다.
- 회색점 : 발현량 변화가 통계적으로 유의미하지 않거나, 변화 자체가 미미한 유전자들이다.
