1. TP53
TP53 유전자는 DNA 손상을 복구하거나, 복구가 불가능할 경우 세포 사멸을 유도하여 암을 억제하는 '게놈의 수호자'라고 불리는 중요한 유전자 이다. 이 유전자에 돌연변이가 발생하면 DNA 손상을 입은 세포가 계속 증식하여 다양한 종류의 암 이 발생할 수 있으며 , 리-프라우메니 증후군(Li-fraumeni like syndrome)과 같은 유전성 암 발생 위험을 높이기도 한다.
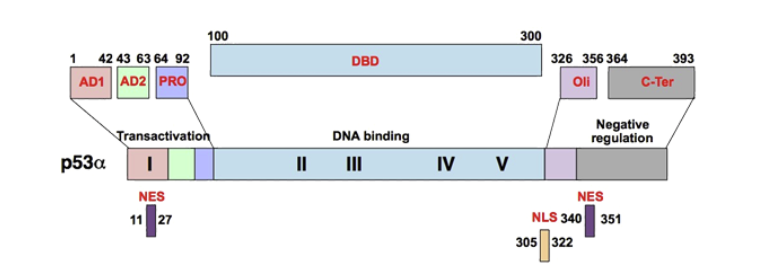
특히 폐암, 유방암, 대장암 등을 포함한 많은 종류의 암에서 50 ~ 60% 정도로 높은 빈도의 변이를 보이는데, 이는 인체 종양에서 발견되는 발암 또는 종양 억제 유전자들의 돌연변이 빈도중에서도 가장 높다.
1-1. TP53(p53 단백질)의 주요 기능
- 세포 주기 정지 : DNA가 손상된 것을 감지하면, p53 단백질은 세포 분열 주기를 일시적으로 멈춘다.
- DNA 복구 : p53 단백질은 손상된 DNA를 수리하는 다른 유전자들을 활성화시켜 DNA를 원상태로 복구하도록 돕는다.
- 세포 사멸 유도(Apoptosis) : 만약 DNA 손상이 너무 심각해 복구가 불가능하다면, p53 단백질은 세포 자살 프로그램 을 가동한다.
1-2. 임상적 중요성
- 진단 및 예후 예측 : 암 조직에서의 TP53 돌연변이의 유무와 종류를 분석하여 암의 악성도를 추적할 수 있다. 일반적으로 TP53 돌연변이가 있는 경우 암은 공격적인 성향을 보인다.
- 치료 반응 예측 : 특정 항암 치료에 대한 반응성을 예측하는 지표로도 사용된다. 일부 항암제는 p53의 정상적인 기능에 의존하여 암세포를 사멸시키므로, TP53에 돌연변이가 있는 경우 치료 효과가 떨어질 수 있다.
1-3. 생화학 경로
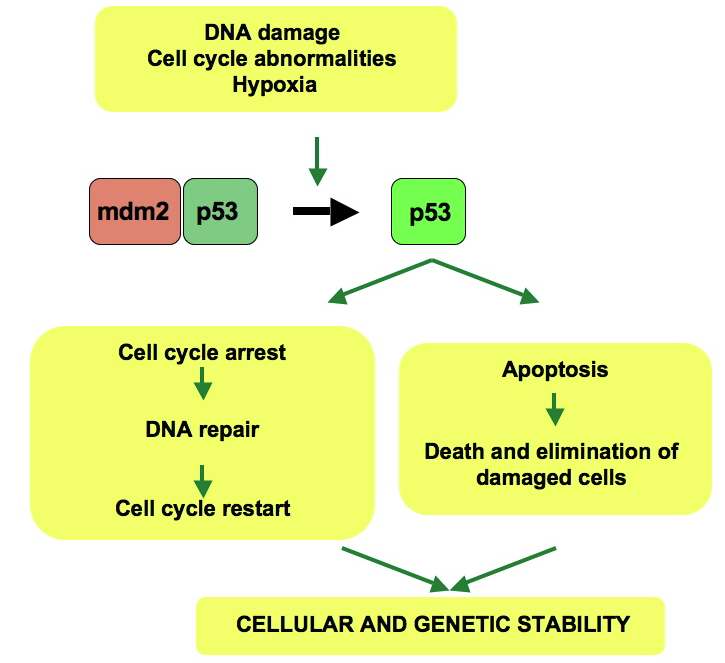
정상 세포에서 p53은 음성조절자 mdm2 에 의해 불활성화 된다. 그러나 DNA손상이나 다른 스트레스가 있을시 다양한 경로들이 p53과 mdm2를 분리시켜 활성화 된다. p53은 세포주기를 멈추게 하고 손상을 고치거나, 세포를 없앨 것을 결정하는데 어떤 원리로 p53이 결정을 내리는지는 아직 알 수 없다.
이는 현재 분자생물학에서 활발히 연구되고 있는 중요한 질문 중 하나이다.
2. FASTA 형식과 BLAST
2-1. FASTA
FASTA 형식은 유전자를 구성하는 염기서열이나 단백질을 구성하는 아미노산 서열을 텍스트로 저장하는 표준 방식이다.
-
Description Line
- 반드시 '>'로 시작한다.- '>' 뒤에는 해당 서열의 이름, 아이디, 출처 등을 식별할 수 있는 정보를 자유롭게 기입한다.
-
Sequence Data
- 설명 줄 다음 줄부터 실제 염기 또는 아미노산 서열을 입력한다.
서열은 한 줄로 길게 쓰고나 여러 줄로 나누어 쓸 수 있으며 줄바꿈, 공백, 숫자 등은 프로그램이 알아서 무시한다.
>gi|568815597|ref|NM_000546.5| Homo sapiens tumor protein p53 (TP53), transcript variant 1, mRNA
AGCTTCGACAGCAGCTC ..............2-2. BLAST (Basic Local Alignment Search Tool)
BLAS는 특정 염기 서열이나 아미노산 서열이 어떤 생물체의 유전자/단백질과 유사한지를 빠르게 검색해준다. 전 세계의 방대한 데이터베이스(ex. NCBI의 GenBank)를 대상으로 내가 가진 서열(Query Sequence)과 비슷한 부분을 찾아준다.
- BLAST 역할
- 유전자 / 단백질 동정 : 미지의 서열이 어떤 기능을 하는 유전자 혹은 단백질인지 정체를 밝힌다.- 종간 유사성 비교 : 비슷한 서열을 가진 다른 종을 찾아내어 진화적인 관계(유연관계)를 추정할 수 있다.
- BLAST 원리와 결과
BLAST는 전체 서열을 하나하나 비교하는 대신, 서열을 짧은 조각(word)으로 나눈 뒤 데이터 베이스에서 이 조각과 일치하는 부분을 먼저 찾아낸다.
그리고 그 주변으로 정렬(alignment)을 확장해 나가며 국소적으로 유사도가 높은 영역(Local Alignment)을 찾아내는 방식이다.
검색이 종료되면 유사도가 높은 순서대로 결과 목록을 보여주는데, 이때 쓰이는 지표가 E-value(Expectation Value) 이다. - E-value : 현재 검색 결과와 같은 유사도(Score)가, 우연히 발견될 수 있는 기대 횟수를 의미한다. 0에 가까울 수록 우연의 일치일 가능성이 희박하며, 두 서열이 통계적으로 유의미한 관계에 있음 을 시사한다.
3. BioPython을 활용한 BLAST
3-1. BioPython
BioPython 은 생물학 데이터를 위한 거대한 패키지이며, 안에는 다양한 목적을 가진 여러 라이브러리가 있다.
이번에 쓰일 라이브러리인 Entrez와 Blast에 대해서 알아보도록 하자.
3-2. Bio.Entrez
Entrez는 미국 국립생물정보센터(NCBI)가 운영하는 방대한 생물학 데이터베이스에 접속하게 해주는 인터페이스 이다.
Entrez.esearch(): 특정 키워드로 지정된 데이터베이스를 검색해서, 조건에 맞는 데이터들의 고유 ID 목록을 찾아준다. 이때, 데이터를 가져오는것이 아님을 주의하자.Entrez.efetch(): 고유 ID를 입력하면 해당 ID의 실제 데이터를 FASTA, GenBank등 원하는 방식으로 가져온다.
3-3. Bio.Blast
Blast는 내가 가진 특정 서열(Query)을 거대한 서열 데이터베이스와 비교하여 유사서열을 찾아주는 프로그램이다.
NCBIWWW.qblast(): NCBI의 공식 BLAST 서버를 사용하여 검색한다.
3-4. Python에서 해보기
from Bio.Blast import NCBIWWW
from Bio.Blast import NCBIXML
from Bio import Entrez
# NCBI 데이터베이스에 접속하기 위해 자신의 이메일을 명시합니다. (NCBI 정책)
Entrez.email = "bublman375@knu.ac.kr"
# 1. 데이터 가져오기 (Entrez)
# protein DB에서 인간 TP53(ID: NP_000537.3)의 서열을 FASTA 형식으로 다운로드
handle = Entrez.efetch(db="protein", id="NP_000537.3", rettype="fasta", retmode="text")
fasta_sequence = handle.read()
handle.close()
print("--- 가져온 서열 ---")
print(fasta_sequence)
# 2. BLAST 실행
print("TP53 - NCBI BLAST Starting...")
# blastp (단백질 vs 단백질)로 nr DB에 검색, 상위 200개 결과를 요청
result_handle = NCBIWWW.qblast("blastp", "nr", fasta_sequence, hitlist_size=200)
print("TP53 - NCBI BLAST Ended")
# 3. 결과 파싱 및 필터링
# XML 형식의 BLAST 결과를 파이썬 객체로 변환
blast_record = NCBIXML.read(result_handle)
print("\n--- BLAST 검색 결과 (인간 & 인공 제작물 제외) ---")
count = 0
for alignment in blast_record.alignments:
# 검색 결과에서 인간(Homo sapiens)과 인공 서열(synthetic construct)은 제외
if "Homo sapiens" not in alignment.title and "synthetic construct" not in alignment.title:
for hsp in alignment.hsps:
count += 1
print(f"순위 {count}: {alignment.title}")
print(f" E-value: {hsp.expect}\n")
# 상위 10개 결과만 출력 후 종료
if count >= 10:
break
result_handle.close()Entrez.efetch(db="protein", id="NP_000537.3", rettype="fasta", retmode="text"):protein데이터베이스에서 고유 ID가NP_000537.3인 데이터를 요청한다. 이는 인간(Homo sapiens)의 p53이다.NCBIWWW.qblast("blastp","nr",fasta_sequence,hitlist_size=200): 일반적으로qblast는 상위 50개 결과만을 우선적으로 가져오지만hitlist_size=200을 통해 상위 200개 결과를 검색하였다.NCBIXML.read를 이용해 BLAST 서버로 받은 XML형식의 데이터를 파이썬이 다루기 쉬운 객체로 변환한다.if "Homo sapiens" not in alignment.title and "synthetic construct" not in alignment.title: 인간(Homo sapiens)와 인공제작물(Synthetic construct)를 제외하여 검색한다.
--- 가져온 서열 ---
>NP_000537.3 cellular tumor antigen p53 isoform a [Homo sapiens]
MEEPQSDPSVEPPLSQETFSDLWKLLPENNVLSPLPSQAMDDLMLSPDDIEQWFTEDPGPDEAPRMPEAA
PPVAPAPAAPTPAAPAPAPSWPLSSSVPSQKTYQGSYGFRLGFLHSGTAKSVTCTYSPALNKMFCQLAKT
CPVQLWVDSTPPPGTRVRAMAIYKQSQHMTEVVRRCPHHERCSDSDGLAPPQHLIRVEGNLRVEYLDDRN
TFRHSVVVPYEPPEVGSDCTTIHYNYMCNSSCMGGMNRRPILTIITLEDSSGNLLGRNSFEVRVCACPGR
DRRTEEENLRKKGEPHHELPPGSTKRALPNNTSSSPQPKKKPLDGEYFTLQIRGRERFEMFRELNEALEL
KDAQAGKEPGGSRAHSSHLKSKKGQSTSRHKKLMFKTEGPDSD
TP53 - NCBI BLAST Starting...
TP53 - NCBI BLAST Ended
--- BLAST 검색 결과 (인간 & 인공 제작물 제외) --
순위 1: ref|XP_063556659.1| cellular tumor antigen p53 isoform X1 [Gorilla gorilla gorilla]
E-value: 0.0
순위 2: ref|XP_004058559.3| cellular tumor antigen p53 isoform X2 [Gorilla gorilla gorilla]
E-value: 0.0
순위 3: ref|XP_002827020.3| cellular tumor antigen p53 [Pongo abelii]
E-value: 0.0
순위 4: ref|XP_030656345.1| cellular tumor antigen p53 [Nomascus leucogenys]
E-value: 0.0
순위 5: ref|XP_054313311.2| cellular tumor antigen p53 [Pongo pygmaeus] >gb|PNJ12854.1| TP53 isoform 1 [Pongo abelii] >gb|PNJ12856.1| TP53 isoform 3 [Pongo abelii]
E-value: 0.0
순위 6: ref|XP_031995228.1| cellular tumor antigen p53 [Hylobates moloch]
E-value: 0.0
순위 7: ref|XP_055112571.1| cellular tumor antigen p53 isoform X1 [Symphalangus syndactylus]
E-value: 0.0
순위 8: ref|XP_017706033.1| PREDICTED: cellular tumor antigen p53 isoform X1 [Rhinopithecus bieti]
E-value: 0.0
순위 9: ref|XP_050621303.1| cellular tumor antigen p53 isoform X1 [Macaca thibetana thibetana]
E-value: 0.0
순위 10: ref|XP_010360689.1| cellular tumor antigen p53 [Rhinopithecus roxellana] >ref|XP_017706034.1| PREDICTED: cellular tumor antigen p53 isoform X2 [Rhinopithecus bieti]
E-value: 0.04. 결론
결과적으로 이 작성한 코드는 인간의 p53 단백질과 진화적으로 가까운 '사촌' 단백질을 다른 종에서 찾아내라 는 명령을 수행하는 파이프라인이다.
다음 결과에서 1,2위는 Gorilla, 3위는 Orangutan(Pongo abelii) ... 임을 알 수 있다.
E-value가 0.0으로 표시되어 있지만 실제로는 0에 무수히 가까운 값이기 때문에 영장류 들이 인간의 유전적 조상임을 알 수 있다.
