학습기반 이미지 압축 논문을 보면서 가장 많이 살펴보는데 아래 RD-Curve 일 것이다.
왜냐면 모델의 성능을 한 눈에 직관적으로 알 수 있는 방법이기 때문이다.
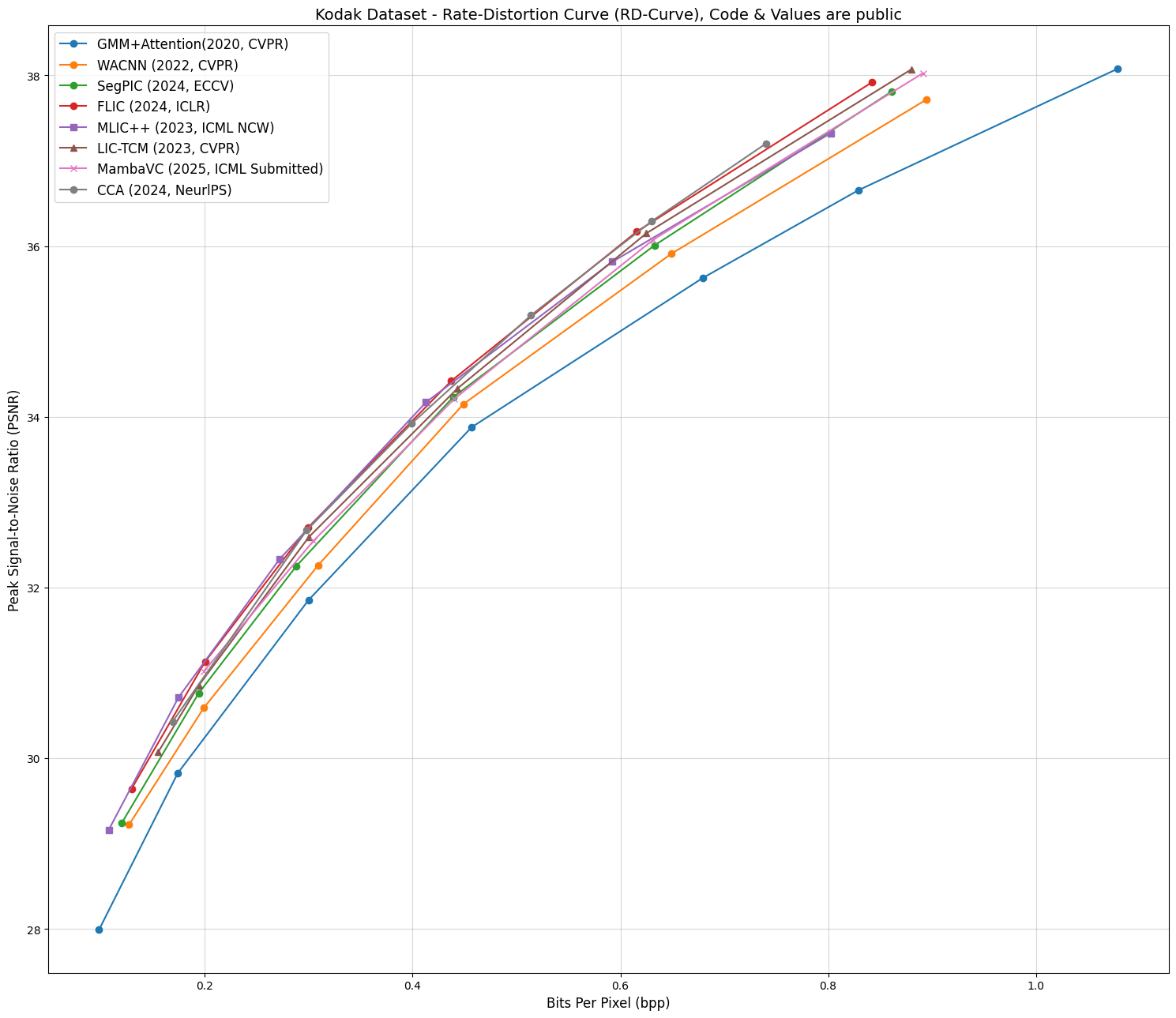
그래프를 보면 알 수 있듯이 제안 모델이 비교 모델보다 더 위에 있다 => 더 좋은 모델이다.
이걸 한 번에 알 수 있는 Figure이다.
하지만 근래 BD-Rate를 공부하면서 눈으로본 RD-Curve와는 다른 평가 결과가 나와서 코드와 같이 알아보고자 한다.
import matplotlib.pyplot as plt
cca_bpp = [0.740, 0.630, 0.514, 0.399, 0.298, 0.169]
cca_psnr = [37.20, 36.29, 35.19, 33.92, 32.67, 30.43]
stf_bpp = [0.127, 0.199, 0.309, 0.449, 0.649, 0.895]
stf_psnr = [29.22, 30.59, 32.26, 34.15, 35.91, 37.72]
# 데이터 입력
bpp_1 = [0.1294, 0.2003, 0.2993, 0.4372, 0.6158, 0.842]
psnr_1 = [29.64, 31.132, 32.702, 34.42, 36.17, 37.918]
bpp_2 = [0.1075, 0.1747, 0.2721, 0.4129, 0.5923, 0.8025]
psnr_2 = [29.161, 30.7137, 32.3345, 34.1702, 35.8161, 37.3229]
bpp_3 = [0.155, 0.194, 0.300, 0.443, 0.625, 0.880]
psnr_3 = [30.07, 30.85, 32.59, 34.33, 36.15, 38.07]
gmm_bpp = [0.098115338, 0.173912113,0.299725056, 0.456783226, 0.679351191, 0.829, 1.079]
gmm_psnr = [ 27.9897452, 29.8244, 31.851, 33.8773766, 35.62697935, 36.654, 38.08]
segpic_bpp = [0.120, 0.194, 0.288, 0.439, 0.633, 0.861]
segpic_psnr = [29.24, 30.76, 32.25, 34.23, 36.01, 37.81]
x_values = [0.891213389, 0.631520223, 0.440167364, 0.30446304, 0.19902371]
y_values = [38.02570573, 36.07381748, 34.20996183, 32.54554182, 31.01809218]
# 그래프 그리기
plt.figure(figsize=(15, 13))
plt.plot(gmm_bpp, gmm_psnr, marker='o', label='GMM+Attention(2020, CVPR)')
plt.plot(stf_bpp, stf_psnr, marker='o', label='WACNN (2022, CVPR)')
plt.plot(segpic_bpp, segpic_psnr, marker='o', label='SegPIC (2024, ECCV)')
plt.plot(bpp_1, psnr_1, marker='o', label='FLIC (2024, ICLR)')
plt.plot(bpp_2, psnr_2, marker='s', label='MLIC++ (2023, ICML NCW)')
plt.plot(bpp_3, psnr_3, marker='^', label='LIC-TCM (2023, CVPR)')
plt.plot(x_values, y_values, marker='x', label='MambaVC (2025, ICML Submitted)')
plt.plot(cca_bpp, cca_psnr, marker='o', label='CCA (2024, NeurlPS)')
# 그래프 설정
plt.xlabel('Bits Per Pixel (bpp)', fontsize=12)
plt.ylabel('Peak Signal-to-Noise Ratio (PSNR)', fontsize=12)
plt.title('Kodak Dataset - Rate-Distortion Curve (RD-Curve), Code & Values are public', fontsize=14)
plt.grid(alpha=0.5)
plt.legend(fontsize=12)
plt.tight_layout()
# 그래프 출력
plt.show()
위 그래프의 코드이며, SOTA라고 생각했던 모델들 중에 깃허브 레포와 RD data를 공개하는 모델만 정리했다.
공개를 하지않으면 내가 다 돌려서 확인해야하기에 너무 귀찮다,,,
특이사항으로 GMM+Attention 모델의 마지막 평가값은 임의로 추가한 값이다.
공식 레포에는 없는 값이다.
Bjontegaard metric
BD-Rate에 대해 알아야하는데 이론은 아래 블로그에서 친절하게 설명해준다.
코드는 위 깃허브 레포를 사용했다.
https://medium.com/innovation-labs-blog/bjontegaard-delta-rate-metric-c8c82c1bc42c
import numpy as np
import scipy.interpolate
def BD_PSNR(R1, PSNR1, R2, PSNR2, piecewise=0):
lR1 = np.log(R1)
lR2 = np.log(R2)
PSNR1 = np.array(PSNR1)
PSNR2 = np.array(PSNR2)
p1 = np.polyfit(lR1, PSNR1, 3)
p2 = np.polyfit(lR2, PSNR2, 3)
# integration interval
min_int = max(min(lR1), min(lR2))
max_int = min(max(lR1), max(lR2))
# find integral
if piecewise == 0:
p_int1 = np.polyint(p1)
p_int2 = np.polyint(p2)
int1 = np.polyval(p_int1, max_int) - np.polyval(p_int1, min_int)
int2 = np.polyval(p_int2, max_int) - np.polyval(p_int2, min_int)
else:
# See https://chromium.googlesource.com/webm/contributor-guide/+/master/scripts/visual_metrics.py
lin = np.linspace(min_int, max_int, num=100, retstep=True)
interval = lin[1]
samples = lin[0]
v1 = scipy.interpolate.pchip_interpolate(np.sort(lR1), PSNR1[np.argsort(lR1)], samples)
v2 = scipy.interpolate.pchip_interpolate(np.sort(lR2), PSNR2[np.argsort(lR2)], samples)
# Calculate the integral using the trapezoid method on the samples.
int1 = np.trapz(v1, dx=interval)
int2 = np.trapz(v2, dx=interval)
# find avg diff
avg_diff = (int2-int1)/(max_int-min_int)
# 소수 둘째자리까지 출력
avg_diff = round(avg_diff, 2)
return avg_diff
def BD_RATE(R1, PSNR1, R2, PSNR2, piecewise=0):
lR1 = np.log(R1)
lR2 = np.log(R2)
# rate method
p1 = np.polyfit(PSNR1, lR1, 3)
p2 = np.polyfit(PSNR2, lR2, 3)
# integration interval
min_int = max(min(PSNR1), min(PSNR2))
max_int = min(max(PSNR1), max(PSNR2))
# find integral
if piecewise == 0:
p_int1 = np.polyint(p1)
p_int2 = np.polyint(p2)
int1 = np.polyval(p_int1, max_int) - np.polyval(p_int1, min_int)
int2 = np.polyval(p_int2, max_int) - np.polyval(p_int2, min_int)
else:
lin = np.linspace(min_int, max_int, num=100, retstep=True)
interval = lin[1]
samples = lin[0]
v1 = scipy.interpolate.pchip_interpolate(np.sort(PSNR1), lR1[np.argsort(PSNR1)], samples)
v2 = scipy.interpolate.pchip_interpolate(np.sort(PSNR2), lR2[np.argsort(PSNR2)], samples)
# Calculate the integral using the trapezoid method on the samples.
int1 = np.trapz(v1, dx=interval)
int2 = np.trapz(v2, dx=interval)
# find avg diff
avg_exp_diff = (int2-int1)/(max_int-min_int)
avg_diff = (np.exp(avg_exp_diff)-1)*100
# 소수 둘째자리까지 출력
avg_diff = round(avg_diff, 2)
return avg_diff
코드는 위 코드를 사용했다. 깃허브 별이 100개 넘어서 공신력있는 코드라 생각해서 검증없이 사용했다.
결과
# BD-Rate 계산
models = {
"FLIC (2024)": (bpp_1, psnr_1),
"MLIC++ (2023)": (bpp_2, psnr_2),
"CCA (2024)": (cca_bpp, cca_psnr),
"LIC-TCM (2023)": (bpp_3, psnr_3),
"MambaVC (2025)": (x_values, y_values),
"SegPIC (2024)": (segpic_bpp, segpic_psnr),
"WACNN (2022)": (stf_bpp, stf_psnr),
}
bd_rate_results = {
model_name: BD_RATE(gmm_bpp, gmm_psnr, bpp, psnr)
for model_name, (bpp, psnr) in models.items()
}
# 결과 출력
bd_rate_results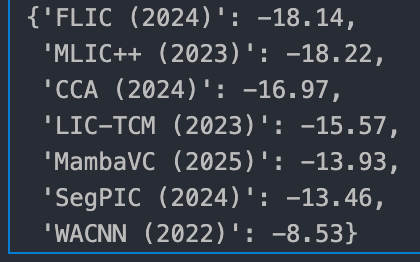
BD-rate는 가장 성능이 낮은 모델인 2020년 CVPR 논문을 기준으로 계산했다.
결과를 살펴보면
- MLIC++
- FLIC
- CCA
- LIC-TCM
- MambaVC
- SegPIC
- WACNN (아마 STF도 이쯤,,)
순으로 나타났고, paper with code에 랭크되어있는 MLIC++가 아직까진 미세한 차이로 SOTA를 달성했다.
VVC로 테스트하면 역전될지 의문이 생기기도한다.
왜냐하면 그래프로보면 값이 큰 람다는 FLIC나 LIC-TCM이 MLIC++보다 더 높은 지표를 가짐에도 BD-rate는 MLIC++가 더 좋게 나타났다. (물론 낮은 람다에선 MLIC++가 더 좋긴하다.)
결론
- MLIC++는 2023년 모델임에도 Kodak 데이터셋에서 SOTA 자리를 지켰다.
- 하지만 2024년에 좋은 모델들이 성능을 많이 끌어올렸다. 특히 FLIC..
- Transformer를 깨부수기위해 나온 Mamba 기반 모델도 압축에선 아직 최적화되지 못한듯하다.
졸업전까지만 안깨졌으면 좋겠다.
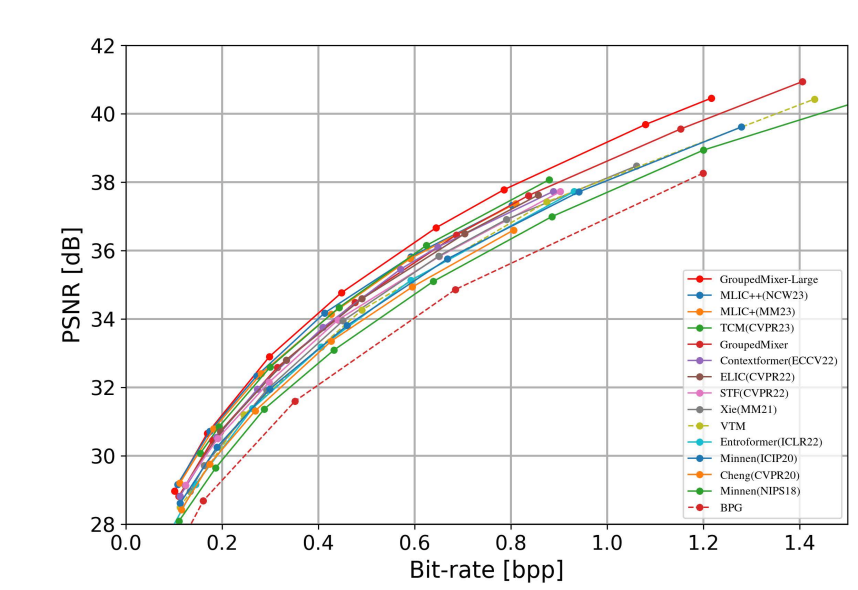
사실 비공식으로는 MLIC++는 1등에서 내려온 것 같다. 하지만 GroupedMixer 모델이 지금 깃허브 레포도 없고 RD-Data도 메일로 여쭤봐도 아직 답이 없으셔서,,, 포함은 시키지않았다.
-> 저널 Accept되면 공개하신다고 답장이 와서 추후 공개될 것 같다.
