교차 검증 개요
학습 데이터세트를 다시 학습 데이터세트 / 검증 데이터세트로 나눔
- 학습(train) 데이터세트: 학습을 위한 데이터
- 검증(validation) 데이터세트: 학습된 모델의 성능을 일차 평가
- 평가(test) 데이터세트: 모든 학습/검증 과정이 완료된 후 최종적으로 성능을 평가
검증 데이터세트가 왜 필요하지?
- train 데이터에 대해서 발생하는 과적합(Overfitting)을 방지하기 위해 검증이 필요
- train 데이터에만 열심히 학습 시켰는데, 막상 test 데이터에서는 맞지 않을 수 있기 때문에 우선 검증 데이터로 평가
- 교차검증은 검증을 여러번 해서 어느 데이터에서나 잘 맞는지 확인
K 폴드 교차 검증
K-Fold Cross Validation
K 개의 폴드 세트에 k 번의 학습과 검증 평가 반복 수행
k = 5 예를 들어보자
학습 데이터세트를 5등분해서 폴드 하나를 검증 데이터세트로, 나머지를 모두 학습 데이터세트로 해서 검증을 하는 것! 아래 표를 보자
| 분할1 | 분할2 | 분할3 | 분할4 | 분할5 |
|---|---|---|---|---|
| 검증 | 학습 | 학습 | 학습 | 학습 |
| 학습 | 검증 | 학습 | 학습 | 학습 |
| 학습 | 학습 | 검증 | 학습 | 학습 |
| 학습 | 학습 | 학습 | 검증 | 학습 |
| 학습 | 학습 | 학습 | 학습 | 검증 |
이렇게 분할해서 각 학습 데이터세트에 대해 학습을 하고, 각 검증 데이터세트에 대해 평가를 한다.
각 평가에 대한 평균이 교차 검증의 최종 평가이다.
scikit-learn과 반복문을 이용해서 구현해보자
sklearn.model_selection의 KFold를 사용함
from sklearn.tree import DecisionTreeClassifier
from sklearn.metrics import accuracy_score
from sklearn.model_selection import KFold
import numpy as np
iris = load_iris()
features = iris.data
label = iris.target
dt_clf = DecisionTreeClassifier(random_state=156)Kfold(n_splits=k) -> k개의 폴드 세트로 분리하는 KFold 객체
# 5개의 폴드 세트로 분리하는 KFold 객체와 폴드 세트별 정확도를 담을 리스트 객체 생성
kfold = KFold(n_splits=5)
cv_accuracy = []
print('붓꽃 데이터 세트 크기:',features.shape[0])붓꽃 데이터 세트 크기: 150현재 붓꽃 데이터 세트의 크기가 150이다.
k=5니까 5분할해서 학습 데이터세트 크기는 120, 검증 데이터세트 크기는 30이 되겠지?
kfold.split(features)로 학습용 / 검증용 index를 분리할 수 있다.
여기서 features는ndarray인데,DataFrame이 들어가도 무관하다!
총 k = 5 번의 반복이 돌면서 아래 과정이 진행된다.
1. 학습 / 검증 데이터세트 설정
2. 훈련 데이터에 대해 DecisionTreeClassifier로 학습
3. 학습한 것을 검증 데이터에 대해 예측
4. 정답(y_test)와 예측한 것(pred)를 비교해서 정확도 측정
반복이 모두 끝나면 마지막에 5개의 정확도에 대해 평균을 내서 평균 검증 정확도를 구한다.
n_iter = 0
# KFold객체의 split( ) 호출하면 폴드 별 학습용, 검증용 테스트의 로우 인덱스를 array로 반환
for train_index, test_index in kfold.split(features):
# kfold.split( )으로 반환된 인덱스를 이용하여 학습용, 검증용 테스트 데이터 추출
X_train, X_test = features[train_index], features[test_index]
y_train, y_test = label[train_index], label[test_index]
#학습 및 예측
dt_clf.fit(X_train , y_train)
pred = dt_clf.predict(X_test)
n_iter += 1
# 반복 시 마다 정확도 측정
accuracy = np.round(accuracy_score(y_test,pred), 4)
train_size = X_train.shape[0]
test_size = X_test.shape[0]
print('\n#{0} 교차 검증 정확도 :{1}, 학습 데이터 크기: {2}, 검증 데이터 크기: {3}'
.format(n_iter, accuracy, train_size, test_size))
print('#{0} 검증 세트 인덱스:{1}'.format(n_iter,test_index))
cv_accuracy.append(accuracy)
# 개별 iteration별 정확도를 합하여 평균 정확도 계산
print('\n 평균 검증 정확도:', np.mean(cv_accuracy)) #1 교차 검증 정확도 :1.0, 학습 데이터 크기: 120, 검증 데이터 크기: 30
#1 검증 세트 인덱스:[ 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29]
#2 교차 검증 정확도 :0.9667, 학습 데이터 크기: 120, 검증 데이터 크기: 30
#2 검증 세트 인덱스:[30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59]
#3 교차 검증 정확도 :0.8667, 학습 데이터 크기: 120, 검증 데이터 크기: 30
#3 검증 세트 인덱스:[60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 76 77 78 79 80 81 82 83 84 85 86 87 88 89]
#4 교차 검증 정확도 :0.9333, 학습 데이터 크기: 120, 검증 데이터 크기: 30
#4 검증 세트 인덱스:[ 90 91 92 93 94 95 96 97 98 99 100 101 102 103 104 105 106 107 108 109 110 111 112 113 114 115 116 117 118 119]
#5 교차 검증 정확도 :0.7333, 학습 데이터 크기: 120, 검증 데이터 크기: 30
#5 검증 세트 인덱스:[120 121 122 123 124 125 126 127 128 129 130 131 132 133 134 135 136 137 138 139 140 141 142 143 144 145 146 147 148 149]
평균 검증 정확도: 0.9이렇게 검증 데이터 세트는 앞에서부터 30개씩 쪼개서 순차적으로 설정된다.
각각의 정확도의 평균이 평균 검증 정확도가 된다.
위 결과에서 각 검증 정확도의 차이가 심한 것을 확인하자!
각 분할에 대해 Label의 분포가 불균형하게 들어갔기 때문에 그렇다.
iris 데이터세트는 0~49 Label이 0, 50~99 Label이 1, 100~149 Label이 2로 되어있는데 앞에서부터 쪼개니까 당연히 문제가 있겠지?
Stratified K 폴드가 이 문제를 해결한다.
Stratified K 폴드
Stratified K-Fold
불균형한 분포도를 가진 레이블 데이터 집합을 위한 K 폴드 방식
학습데이터와 검증 데이터를 나눌 때, 각 레이블의 비율을 맞춰서 나눈다.
예를들어 데이터세트의 크기가 150이고, Label이 [0, 1, 2] 3 종류, K = 3 이라고 하자
그럼 학습데이터 크기는 100, 검증 데이터 크기는 50이 되겠지?
여기서 각 Label의 갯수가 아래처럼 나눠지는 것
| Label | 학습 데이터 | 검증 데이터 |
|---|---|---|
| 0 | 33 | 16 |
| 1 | 33 | 17 |
| 2 | 34 | 17 |
| 합 | 100 | 50 |
scikit-learn으로 확인해보자
K=3인 경우이고, split(X, y)가 들어가는 것을 확인하자!
Label에 대한 정보가 있어야 Label의 비율을 맞춰서 넣어줄 수 있겠지?
그래서 y가 들어가야 됨!
from sklearn.model_selection import StratifiedKFold
skf = StratifiedKFold(n_splits=3)
n_iter=0
for train_index, test_index in skf.split(iris_df, iris_df['label']):
n_iter += 1
label_train= iris_df['label'].iloc[train_index]
label_test= iris_df['label'].iloc[test_index]
print('## 교차 검증: {0}'.format(n_iter))
print('학습 레이블 데이터 분포:\n', label_train.value_counts())
print('검증 레이블 데이터 분포:\n', label_test.value_counts())## 교차 검증: 1
학습 레이블 데이터 분포:
2 34
1 33
0 33
Name: label, dtype: int64
검증 레이블 데이터 분포:
1 17
0 17
2 16
Name: label, dtype: int64
## 교차 검증: 2
학습 레이블 데이터 분포:
1 34
2 33
0 33
Name: label, dtype: int64
검증 레이블 데이터 분포:
2 17
0 17
1 16
Name: label, dtype: int64
## 교차 검증: 3
학습 레이블 데이터 분포:
0 34
2 33
1 33
Name: label, dtype: int64
검증 레이블 데이터 분포:
2 17
1 17
0 16
Name: label, dtype: int64Label의 비율에 맞춰서 학습 / 검증 데이터가 분리된 것을 확인할 수 있음
정확도가 어떻게 나오는지 확인하고 일반 K-Fold 교차 검증과 비교해보자
split()은 위에서 말했던 것처럼DataFrame이 들아와도 무관하다!
dt_clf = DecisionTreeClassifier(random_state=156)
skfold = StratifiedKFold(n_splits=3)
n_iter=0
cv_accuracy=[]
# StratifiedKFold의 split( ) 호출시 반드시 레이블 데이터 셋도 추가 입력 필요
for train_index, test_index in skfold.split(features, label):
# split( )으로 반환된 인덱스를 이용하여 학습용, 검증용 테스트 데이터 추출
X_train, X_test = features[train_index], features[test_index]
y_train, y_test = label[train_index], label[test_index]
#학습 및 예측
dt_clf.fit(X_train , y_train)
pred = dt_clf.predict(X_test)
# 반복 시 마다 정확도 측정
n_iter += 1
accuracy = np.round(accuracy_score(y_test,pred), 4)
train_size = X_train.shape[0]
test_size = X_test.shape[0]
print('\n#{0} 교차 검증 정확도 :{1}, 학습 데이터 크기: {2}, 검증 데이터 크기: {3}'
.format(n_iter, accuracy, train_size, test_size))
print('#{0} 검증 세트 인덱스:{1}'.format(n_iter,test_index))
cv_accuracy.append(accuracy)
# 교차 검증별 정확도 및 평균 정확도 계산
print('\n## 교차 검증별 정확도:', np.round(cv_accuracy, 4))
print('## 평균 검증 정확도:', np.mean(cv_accuracy)) #1 교차 검증 정확도 :0.98, 학습 데이터 크기: 100, 검증 데이터 크기: 50
#1 검증 세트 인덱스:[ 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 100 101 102 103 104 105 106 107 108 109 110 111 112 113 114 115]
#2 교차 검증 정확도 :0.94, 학습 데이터 크기: 100, 검증 데이터 크기: 50
#2 검증 세트 인덱스:[ 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 67 68 69 70 71 72 73 74 75 76 77 78 79 80 81 82 116 117 118 119 120 121 122 123 124 125 126 127 128 129 130 131 132]
#3 교차 검증 정확도 :0.98, 학습 데이터 크기: 100, 검증 데이터 크기: 50
#3 검증 세트 인덱스:[ 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 83 84 85 86 87 88 89 90 91 92 93 94 95 96 97 98 99 133 134 135 136 137 138 139 140 141 142 143 144 145 146 147 148 149]
## 교차 검증별 정확도: [0.98 0.94 0.98]
## 평균 검증 정확도: 0.9666666666666667일반 K-Fold보다 정확도가 훨씬 균일하게 나오는 것을 확인할 수 있음
평균 검증 정확도도 더 높게 나온다!
cross_val_score()
사실 scikit-learn에는 교차 검증을 훨씬 쉽게 해주는 함수가 있음
그것이 cross_val_score()
이 함수는 기본적으로 Stratified K-Fold를 사용함
바로 예시 코드로 이해해보자
cross_val_score(Estimator, Data (X), Label (y), 평가 척도, K) 들어가 있는 것 확인
from sklearn.tree import DecisionTreeClassifier
from sklearn.model_selection import cross_val_score , cross_validate
from sklearn.datasets import load_iris
iris_data = load_iris()
dt_clf = DecisionTreeClassifier(random_state=156)
data = iris_data.data
label = iris_data.target
# 성능 지표는 정확도(accuracy) , 교차 검증 세트는 3개
scores = cross_val_score(dt_clf , data , label , scoring='accuracy',cv=3)
print('교차 검증별 정확도:',np.round(scores, 4))
print('평균 검증 정확도:', np.round(np.mean(scores), 4))교차 검증별 정확도: [0.98 0.94 0.98]
평균 검증 정확도: 0.9667Stratified K-Fold는 Label의 비율을 맞춰주는 거니까 분류에서만 사용 가능하고 회귀에선 안 되겠지?
그래서 회귀에서 cross_val_score()는 일반 K-Fold 교차 검증으로 동작한다!
GridSearchCV
교차 검증과 최적 하이퍼 파라미터 튜닝을 한 번에 하는 것
하이퍼 파라미터는 Decision Tree를 예를 들면 max_depth, min_samples_split 이런 것들, 자세한 설명은 여기서 안 다룸
하이퍼 파라미터를 순차적으로 변경하면서 각각 교차 검증을 실행해서 가장 높은 정확도를 가진 하이퍼 파라미터 조합을 찾는 것!
따라서 ( CV 세트 수 * 파라미터 순차 적용 횟수) 만큼 학습/검증을 수행!
parameters를 이렇게
Dictionary형태로 만들어줘야 함
Dict의 value가 1개여도 무조건list로 만들어야 함!
여기서는 파리미터 조합이 3 * 2 로 총 6개가 나온다.
from sklearn.datasets import load_iris
from sklearn.tree import DecisionTreeClassifier
from sklearn.model_selection import GridSearchCV
# 데이터를 로딩하고 학습데이타와 테스트 데이터 분리
iris = load_iris()
X_train, X_test, y_train, y_test = train_test_split(iris_data.data, iris_data.target,
test_size=0.2, random_state=121)
dtree = DecisionTreeClassifier()
### parameter 들을 dictionary 형태로 설정
parameters = {'max_depth':[1,2,3], 'min_samples_split':[2,3]}GridSearchCV(Estimator, Parameters, K, refit 여부) 확인
refit은 최적의 파라미터를 조합을 찾은 다음에 그 조합으로 다시 학습시켜놓을지를 정하는 것임
import pandas as pd
grid_dtree = GridSearchCV(dtree, param_grid=parameters, cv=3, refit=True)
# 붓꽃 Train 데이터로 param_grid의 하이퍼 파라미터들을 순차적으로 학습/평가
grid_dtree.fit(X_train, y_train)
# GridSearchCV 결과 추출하여 DataFrame으로 변환
scores_df = pd.DataFrame(grid_dtree.cv_results_)
scores_df[['params', 'mean_test_score', 'rank_test_score', \
'split0_test_score', 'split1_test_score', 'split2_test_score']]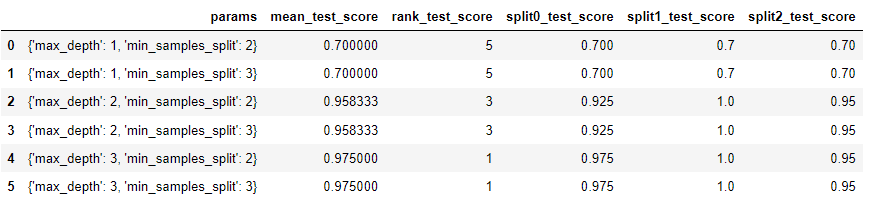
이렇게 최적의 파라미터 조합을 확인할 수 있다.
best_params_에 최적의 파라미터 조합을 저장하고,
best_score_에 최적의 파라미터 조합의 score를 저장한다.
refit=True설정해야 위에 값들이 각각 저장됨
print('GridSearchCV 최적 파라미터:', grid_dtree.best_params_)
print('GridSearchCV 최고 정확도: {0:.4f}'.format(grid_dtree.best_score_))
# refit으로 최적의 파라미터 세트가 들어있음
pred = grid_dtree.predict(X_test)
print('테스트 데이터 세트 정확도: {0:.4f}'.format(accuracy_score(y_test,pred)))GridSearchCV 최적 파라미터: {'max_depth': 3, 'min_samples_split': 2}
GridSearchCV 최고 정확도: 0.9750
테스트 데이터 세트 정확도: 0.9667
refit=True로 설정했기 때문에 best_estimator_에 이미 최적으로 학습된 estimator가 반환됨
예측하고 정확도를 구하면 위와 동일하게 나옴
# GridSearchCV의 refit으로 이미 학습이 된 estimator 반환
estimator = grid_dtree.best_estimator_
# GridSearchCV의 best_estimator_는 이미 최적 하이퍼 파라미터로 학습이 됨
pred = estimator.predict(X_test)
print('테스트 데이터 세트 정확도: {0:.4f}'.format(accuracy_score(y_test,pred)))테스트 데이터 세트 정확도: 0.9667