06-1 군집 알고리즘
[키워드]
비지도 학습
: 머신러닝의 한 종류, 훈련 데이터에 타깃X(= 입력 데이터만 있음) -> 외부의 도움없이 스스로 학습
즉, 알고리즘이 스스로 데이터 속의 패턴을 찾아냄
ex) 군집, 차원 축소 등
히스토그램
: 구간별로 값이 발생한 빈도를 그래프로 표시한 것
- 보통 x축이 값의 구간(계급)이고 y축이 발생 빈도(도수)
군집
: 비슷한 샘플끼리 하나의 그룹으로 모으는 대표적인 비지도 학습 작업
- 군집 알고리즘으로 모은 샘플 그룹을 클러스터라고 부름
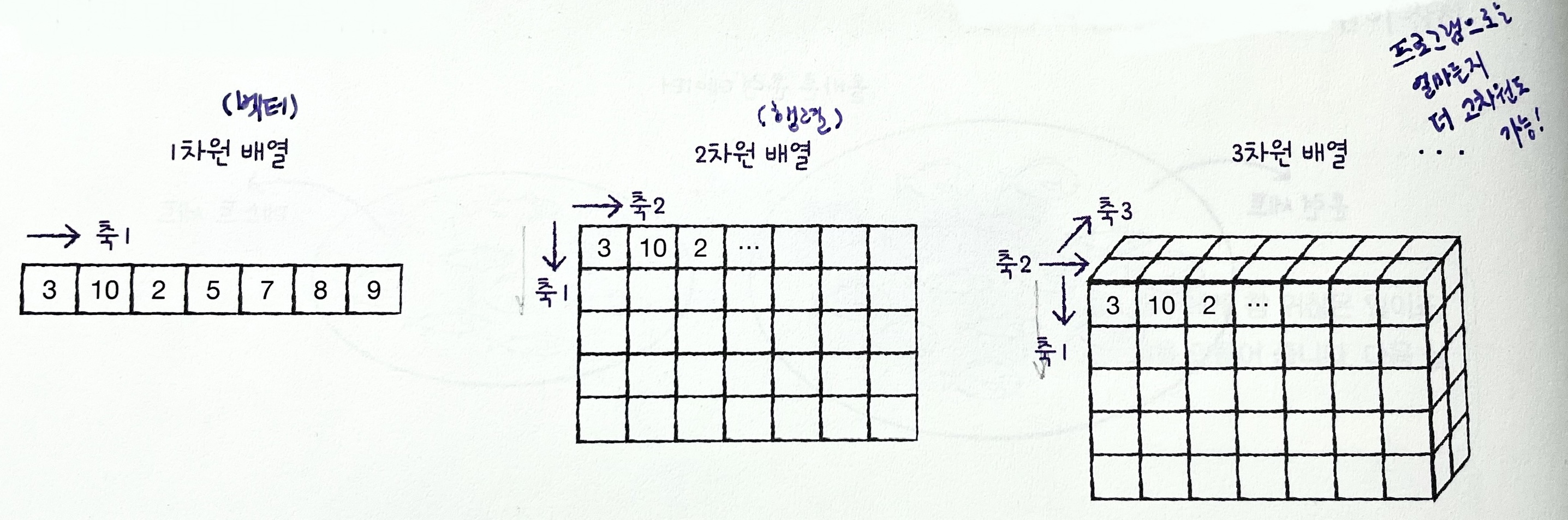
사진 데이터 다루기
데이터 가져오기
- 넘파이 배열로 저장된 파일 다운로드
!: 리눅스 shell 명령으로 바꿔줌wget: 웹에서 파일을 다운로드하여 저장하는 명령어(-0 뒤에는 파일 이름)
# 과일 데이터 준비
!wget https://bit.ly/fruits_300_data -O fruits_300.npy- 다운받은 파일 불러오고 배열의 크기 확인 -> 3차원 데이터
np.load(): npy파일 불러오는 메소드
import numpy as np
import matplotlib.pyplot as plt
# 다운로드 받은 파일 불러오기
fruits = np.load('fruits_300.npy')
print(fruits.shape)
# 가로세로 100x100, 300개
->
(300, 100, 100)- 첫번째 행(가로)에 있는 픽셀 100개 값 출력
- 이 배열은 흑백, 0~255 정수값 가짐(숫자 클수록 밝음)
(.shape결과와 동일한 순서로 인덱싱 = 샘플, 가로, 세로)
print(fruits[0, 0, :])
->
[ 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 2 1
2 2 2 2 2 2 1 1 1 1 1 1 1 1 2 3 2 1
2 1 1 1 1 2 1 3 2 1 3 1 4 1 2 5 5 5
19 148 192 117 28 1 1 2 1 4 1 1 3 1 1 1 1 1
2 2 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
1 1 1 1 1 1 1 1 1 1]데이터 그리기
plt.imshow(): 넘파이 배열에 저장된 이미지를 그림cmap: 이미지 색상 테마를 지정해줌
plt.imshow(fruits[0], cmap='gray')
plt.show()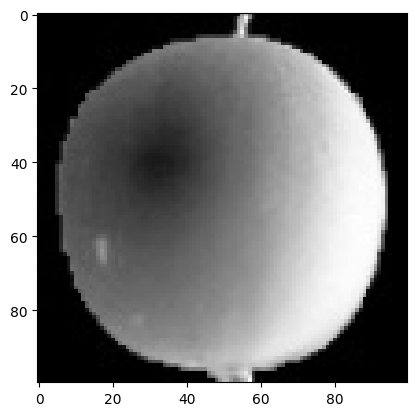
=> 위에서 첫번째 행을 출력했을 때 사진 상에서 맨 위에 꼭지 부분에 색이 달라서 숫자가 크게 나온 것을 알 수 있음
- 색 반전을 위해
gray_r을 사용, 원래랑 거꾸로 낮은 숫자가 높아지고 높은 숫자가 낮아짐
plt.imshow(fruits[0], cmap='gray_r')
plt.show()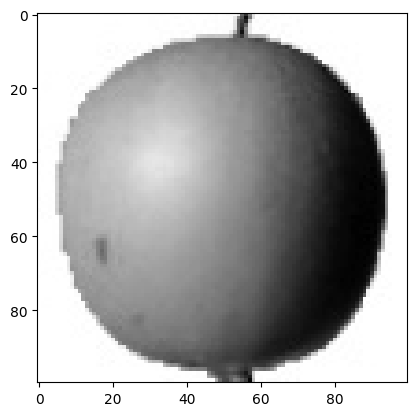
- 파인애플과 바나나도 그려보기
plt.subplots(a, b): a행 b열로 그래프 여러 개를 나란히 그려주는 함수axs:subplot으로 그린 여러 그래프를 갖고 있는 리스트 배열
fig, axs = plt.subplots(1, 2) # 1행 2열로 그래프 배치
axs[0].imshow(fruits[100], cmap='gray_r') # 100번째 사진
axs[1].imshow(fruits[200], cmap='gray_r') # 200번째 사진
plt.show()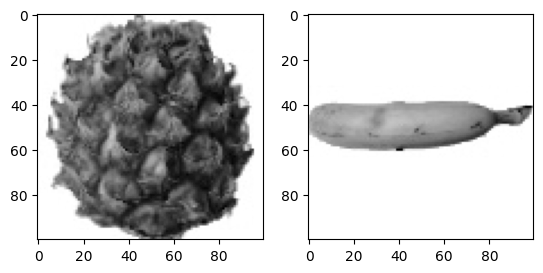
픽셀값 분석하기
1차원 배열로 변환
- 각 이미지 데이터(100x100)를 2차원->1차원으로 바꿈(복잡하니까)
-> 길이가 10,000인 1차원 배열이 됨
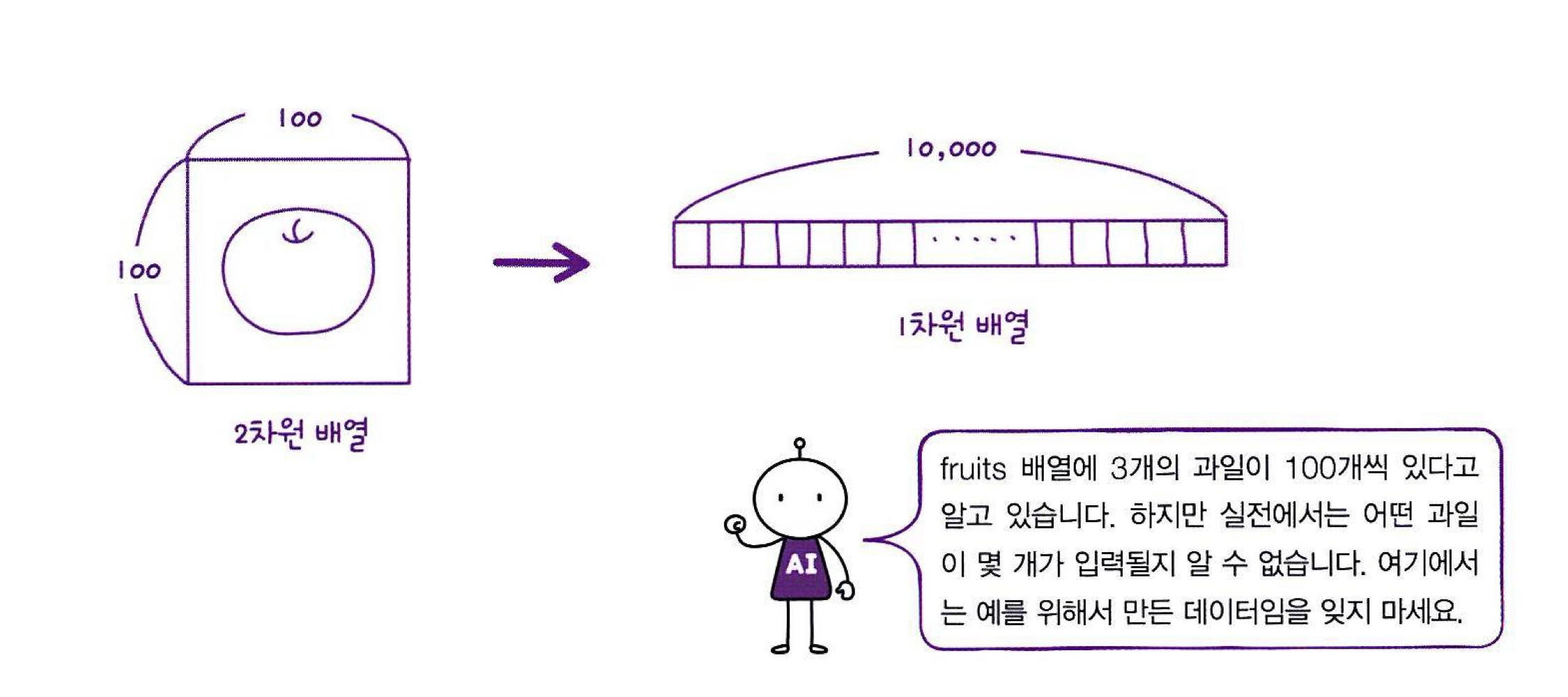
-.reshape를 사용해서 (100, 100, 100)이던 apple / pineapple / banana
-> 두번째 차원(100)과 세번째 차원(100)을 10,000(100, 100*100)으로 합침
- 첫번째 차원을 -1로 지정하면 자동으로 남은 차원 할당
apple = fruits[0:100].reshape(-1, 100*100) # 과일 100개씩 있음
pineapple = fruits[100:200].reshape(-1, 100*100) # 다 하나하나 길게 펼쳐줌
banana = fruits[200:300].reshape(-1, 100*100)
print(apple.shape)
->
(100, 10000) # 100개의 샘플, 샘플 당 10,000픽셀샘플의 픽셀 평균값
넘파이 배열 계산 시 주의
- 각 샘플마다 픽셀의 평균값 계산해야 함 ->
mean()메서드 사용, 평균을 계산할 축 지정해줘야하는데axis=0사용
ex)axis=0: 첫 번째 축인 행따라 계산,axis=1: 두번째 축인 열따라 계산
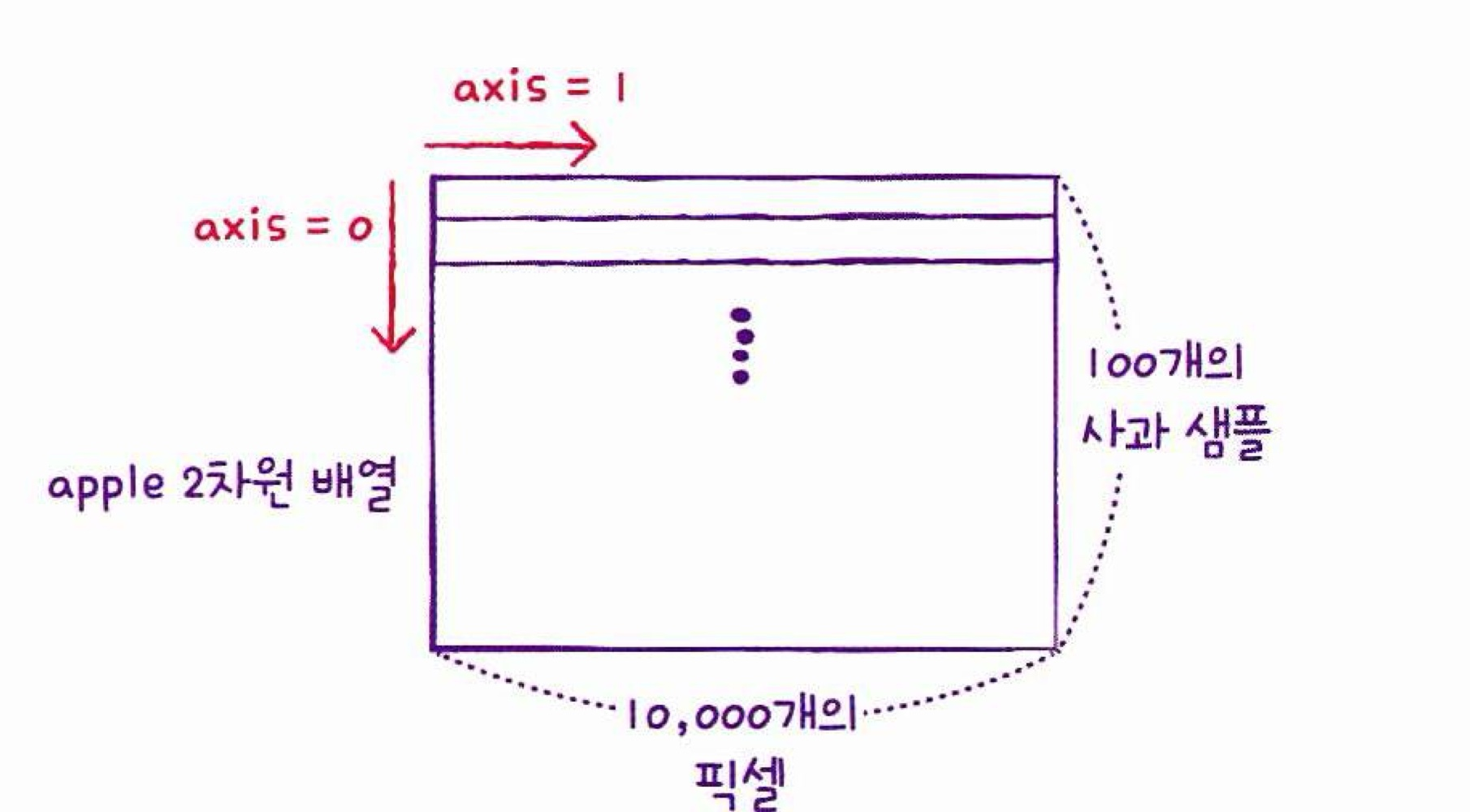
- 사과 100개에 대한 픽셀 평균값 계산
print(apple.mean(axis=1))
->
[ 88.3346 97.9249 87.3709 98.3703 92.8705 82.6439 94.4244 95.5999
90.681 81.6226 87.0578 95.0745 93.8416 87.017 97.5078 87.2019
88.9827 100.9158 92.7823 100.9184 104.9854 88.674 99.5643 97.2495
94.1179 92.1935 95.1671 93.3322 102.8967 94.6695 90.5285 89.0744
97.7641 97.2938 100.7564 90.5236 100.2542 85.8452 96.4615 97.1492
90.711 102.3193 87.1629 89.8751 86.7327 86.3991 95.2865 89.1709
96.8163 91.6604 96.1065 99.6829 94.9718 87.4812 89.2596 89.5268
93.799 97.3983 87.151 97.825 103.22 94.4239 83.6657 83.5159
102.8453 87.0379 91.2742 100.4848 93.8388 90.8568 97.4616 97.5022
82.446 87.1789 96.9206 90.3135 90.565 97.6538 98.0919 93.6252
87.3867 84.7073 89.1135 86.7646 88.7301 86.643 96.7323 97.2604
81.9424 87.1687 97.2066 83.4712 95.9781 91.8096 98.4086 100.7823
101.556 100.7027 91.6098 88.8976]- 세 과일 모두 평균 계산하고 히스토그램으로 그려서 비교
alpha: 그래프 투명도 설정plt.legend: 범례 첨부 설정
plt.hist(np.mean(apple, axis=1), alpha=0.8)
plt.hist(np.mean(pineapple, axis=1), alpha=0.8)
plt.hist(np.mean(banana, axis=1), alpha=0.8)
plt.legend(['apple', 'pineapple', 'banana'])
plt.show()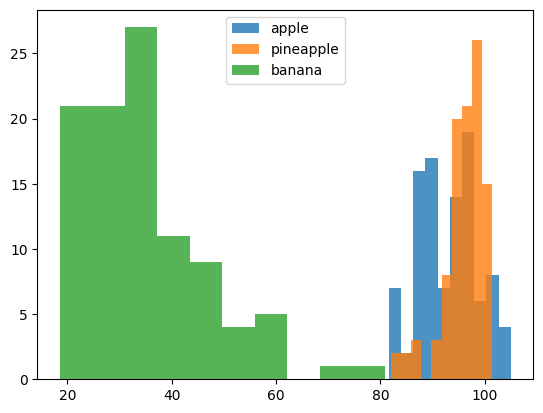
=> 바나나는 값들이 40 이하에 집중되어있어 이 값을 활용해 구분 가능
but, 사과랑 파인애플은 어찌해야될까
픽셀의 샘플 평균값
axis=0으로 픽셀마다 샘플의 평균값 계산
fig, axs = plt.subplots(1, 3, figsize=(20, 5))
axs[0].bar(range(10000), np.mean(apple, axis=0))
axs[1].bar(range(10000), np.mean(pineapple, axis=0))
axs[2].bar(range(10000), np.mean(banana, axis=0))
plt.show()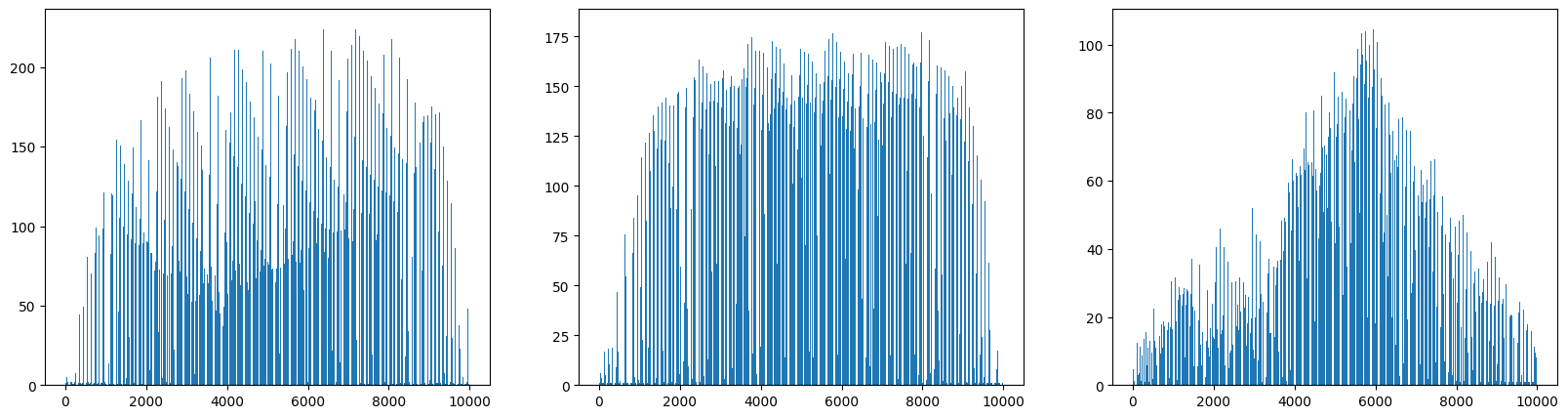
=> 과일마다 값이 높은 구간이 다 다름
평균 이미지 활용하기
평균 이미지와 가까운 사진 고르기
- 샘플 평균값
np.mean(apple, axis=0)-> 2차원 배열로 이미지처럼 출력(100x100)크기
apple_mean = np.mean(apple, axis=0).reshape(100, 100)
pineapple_mean = np.mean(pineapple, axis=0).reshape(100, 100)
banana_mean = np.mean(banana, axis=0).reshape(100, 100)
fig, axs = plt.subplots(1, 3, figsize=(20, 5))
axs[0].imshow(apple_mean, cmap='gray_r')
axs[1].imshow(pineapple_mean, cmap='gray_r')
axs[2].imshow(banana_mean, cmap='gray_r')
plt.show()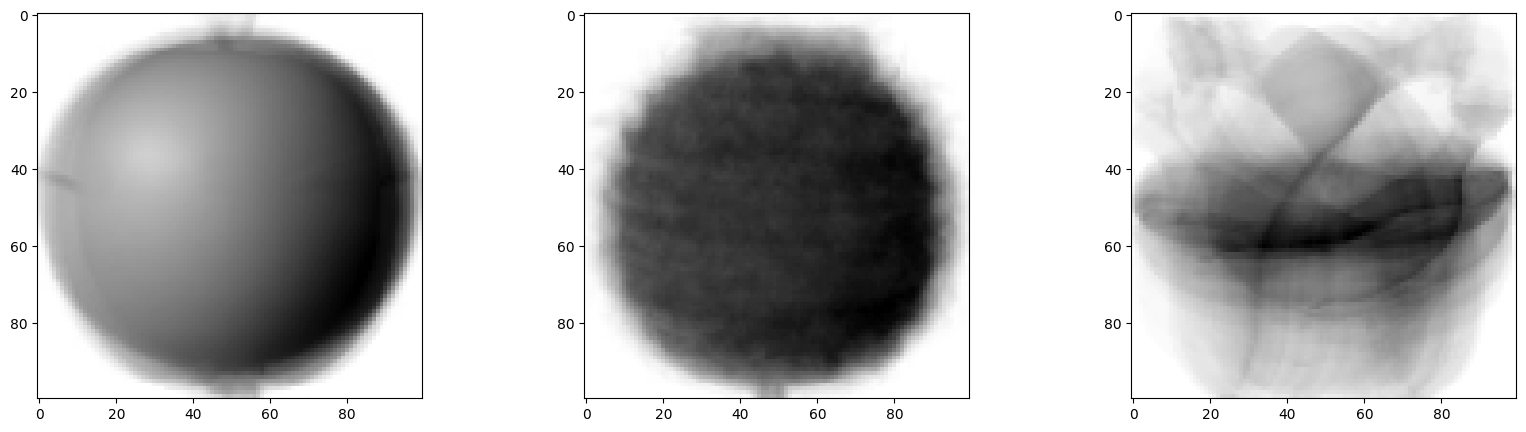
=> 픽셀마다 샘플들이 차지하는 값을 평균 낸 이미지
- 모든 사진을 합쳐놓은 '대표 이미지'처럼 보임
고객이 올린 사진을 '대표 이미지'와 비교하면 어떤 과일인지 구분 할 수 있지 않을까?, 대표 이미지와 차이가 가장 적은 것으로 채택하면 됨
- 그렇다면 우리가 가진 300개의 사진 중 사과의 대표 이미지와 가장 가까운 사진을 고르자
-apple_mean: 사과의 대표 이미지(= 사과 사진 100장의 평균 이미지)
.abs(): 절대값으로 계산하는 함수(absolute)
abs_diff = np.abs(fruits - apple_mean)
abs_mean = np.mean(abs_diff, axis=(1,2))
print(abs_mean.shape)
->
(300,)-
abs_mean: 차이값 요약한 값 -> 가장 작은 샘플이 가장 사과에 가까운 것 -
np.arsort(): 작은 값부터 순서대로 인덱스 반환하는 함수 -
처음 100개에 대해서 작은 순으로 나열해봄
-
인덱스 중 (i*10+j)번째 것을 [i, j]위치에 그림으로 그려봄
-> 사과 대표 이미지에 가장 가까운 것부터 100개의 그림이 그려짐
apple_index = np.argsort(abs_mean)[:100]
fig, axs = plt.subplots(10, 10, figsize=(10,10))
for i in range(10):
for j in range(10):
axs[i, j].imshow(fruits[apple_index[i*10 + j]], cmap='gray_r')
axs[i, j].axis('off')
plt.show()
↪ abs_mean에서 처음 100개만 했으니, 모두 사과로 나오면 OK!
군집(clustering)
- 사진들이 갖고 있는 픽셀값을 사용해 과일 사진을 모으는 작업을 해봄
→ 이렇게 비슷한 샘플끼리 그룹으로 모으는 작업을 군집이라고 하며, 군집 알고리즘을 통해 묶은 그룹들을 클러스터(cluster)라고 부름. (대표적인 비지도학습) - 근데 사실 이번 시간에는, 사과/파인애플/바나나의 평균값을 계산해서 찾은 거니까
사실상 타깃이 있는 셈 (≠비지도 학습)
→ 실제 비지도 학습에서는 이렇게 샘플의 평균값들을 미리 구할 수 없음
06-2 k-평균
[키워드]
k-평균
: k-평균 알고리즘은 처음에 랜덤하게 클러스터 중심을 정하고 클러스터를 만듦
-> 그 다음 클러스터의 중심을 이동하고 다시 클러스터를 만드는 식으로 반복해서 최적의 클러스터 구성을 만듦
클러스터 중심
: k-평균 알고리즘이 만든 클러스터에 속한 샘플의 특성 평균값으로 센트로이드(centroid)라고도 함
- 가장 가까운 클러스터 중심을 샘플의 또 다른 특성으로 사용하거나 새로운 샘풀에 대한 예측으로 활용가능
엘보우 방법
: 최적의 클러스터 개수를 정하는 방법 중 하나
- 이너셔: 샘플 사리 거리의 제곱의 합
- 클러스터 개수에 따라 이너셔 감소가 꺾이는 지점이 적절한 클러스터 개수 k가 될 수 있음
- 그래프의 모양을 따서 엘보우라고 함
k-평균 알고리즘이란?
[작동 방식]
1. 무작위로 k개릐 클러스터 중심을 정함
- '클러스터 중심'이란 앞선 apple_mean같은 평균값(대표 이미지를 구성하는 픽셀 10,000개)를 다르게 표현한 것
2. 각 샘플에서 가장 가까운 클러스터 중심을 찾아 해당 클러스터의 샘플로 지정
- 가장 가까운 '몇 개'를 할지 -> n_clusters로 설정
3. 클러스터에 속한 샘플의 평균값으로 클러스터 중심을 변경
- 처음과 다른 k개의 평균값이 구해짐(클러스터 중심 이동)
4. 클러스터 중심에 변화가 없을 때까지 2번으로 돌아가 반복
KMeans 클래스
모델 훈련하기
- 300개의 과일 데이터 준비
!wget https://bit.ly/fruits_300_data -O fruits_300.npy
import numpy as np
fruits = np.load('fruits_300.npy')- 샘플개수, 너비, 높이 크기의 3차원 배열을 (샘플개수, 너비*높이)크기의 2차원 배열로 바꿔줌
fruits_2d = fruits.reshape(-1, 100*100)KMeans(): 사이킷런에서 k-평균 알고리즘이 구현되어 있는 클래스n_clusters: 클러스터 개수를 지정하는 매개변수
from sklearn.cluster import KMeans
km = KMeans(n_clusters=3, random_state=42) # 클러스터 3개
km.fit(fruits_2d) # fit으로 모델 훈련, 타깃x -> 입력데이터만 전달
# 타깃을 모르는 상태로 모델이 직접 학습하는 것 -> '비지도 학습'- 군집(clustering)의 결과는
.lebels_에 저장되어 있음
print(km.labels_)
->
[2 2 2 2 2 0 2 2 2 2 2 2 2 2 2 2 2 2 0 2 2 2 2 2 2 2 0 2 2 2 2 2 2 2 2 2 2
2 2 2 2 2 0 2 0 2 2 2 2 2 2 2 0 2 2 2 2 2 2 2 2 2 0 0 2 2 2 2 2 2 2 2 0 2
2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 0 2 2 2 2 2 2 2 2 0 0 0 0 0 0 0 0 0 0 0
0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
1 1 1 1 1 1 1 1 1 0 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
1 1 1 1 1 1 1 1 1 1 1 1 1 1 0 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
1 1 1 1]np.unique: 각 레이블에 몇개씩 묶여있는지 확인return_counts: 고유값의 개수를 보여주는 옵션 매개변수
print(np.unique(km.labels_, return_counts=True))
->
(array([0, 1, 2], dtype=int32), array([112, 98, 90]))이미지 출력
-
클러스터에 들어있는 이미지를 그려주는 함수 만들기
-
draw_fruits: arr에 3차원 배열을 입력 받아서 그림으로 출력해줄 함수 -
squeeze = False: axs를 항상 2차원 배열로 다루기 위해서 설정
import matplotlib.pyplot as plt
def draw_fruits(arr, ratio=1): # 전달값 arr, ratio는 figsize때문에 설정
n = len(arr) # n은 샘플 개수
# 한 줄에 10개씩 이미지를 그림. 샘플 개수를 10으로 나누어 전체 행 개수를 계산
rows = int(np.ceil(n/10))
# 행이 1개 이면 열 개수는 샘플 개수 그렇지 않으면 10개
cols = n if rows < 2 else 10
fig, axs = plt.subplots(rows, cols,
figsize=(cols*ratio, rows*ratio), squeeze=False)
for i in range(rows):
for j in range(cols):
if i*10 + j < n: # n 개까지만 그립니다.
axs[i, j].imshow(arr[i*10 + j], cmap='gray_r')
# [i, j] 위치에 전달받은 arr의 i*10+j번째 샘플을 그려라
axs[i, j].axis('off') # 테두리 끄기
plt.show()draw_fruits(fruits[km.labels_==0])
draw_fruits(fruits[km.labels_==1])
클러스터 중심 활용
- 위에서 '클러스터 중심(=샘플 평균값)' ->
.cluster_centers_에 저장되어 있음 - 3차원 배열로 다시 바꿔서 그림으로 나타내보면 '평균 이미지(대표 이미지)'가 나옴
draw_fruits(km.cluster_centers_.reshape(-1, 100, 100), ratio=3)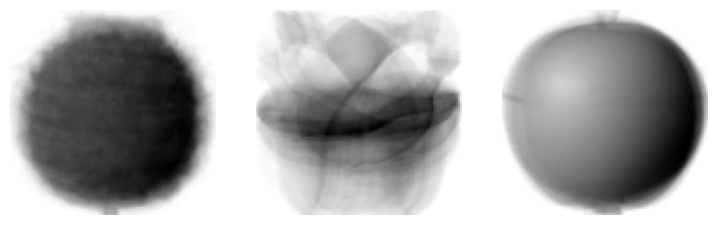
-.transform: KMeans의 메소드 중 하나, 특정 샘플을 넣으면 그 샘플과 클러스터 중심들까지의 거리 반환
- 10000개(픽셀)의 특성을 3개(거리)의 특성으로 차원을 줄여주는 변환기로 사용 가능
print(km.transform(fruits_2d[100:101]))
->
[[3400.24197319 8837.37750892 5279.33763699]].predict(): 위 거리를 이용해 예측 클래스를 출력하는 메소드
# 위에서 구한 거리가 가장 짧은 것이 그 샘플이 속하는 클러스터
print(km.predict(fruits_2d[100:101]))
->
[0].n_iter_: 클러스터 중심을 옮기는 '반복횟수'가 저장된 속성
# 실제로 그려보면 예측한 클러스터 0(파인애플)이 맞음
draw_fruits(fruits[100:101])
# 중심을 옮기면서 찾는 과정 반복 횟수 = 4
print(km.n_iter_)
->
4
➕참고로, .transform()과 .predict()는 다른 군집 알고리즘엔 잘 없음. KMeans의 특징적인 메소드임...!
최적의 K 찾기
k-평균 알고리즘의 단점
- 종류가 몇개인기 알기 때문에 n_cluster=3으로 fit함
-> 몇 개인지 모르는 상황에서는 클러스터 개수를 몇 개로 할지 판단 어려움 - 적절한 k값 찾기가 중요함
엘보우(elbow) 방법
1) 앞에서 .transform()으로 구했던 '거리'의 제곱 합을 이너셔(inertia)라 함
2) 이 이너셔 값이 작다는 것은, 클러스터 중심에 샘플들이 조밀하게 잘 모여있다고 해석할 수 있음. (= 작을수록 좋다고 볼 수 있음)
3) 보통 클러스터 개수(k)를 늘리면, 더 곳곳에 센트로이드가 자리하니까 이너셔가 줄어듦.
- 이 원리를 활용해, 클러스터 개수를 바꿔가면서 이너셔 값의 변화를 그래프로 살펴보면, 보통 감소 속도가 꺾이는 지점이 있기 마련 ➡️ 그 지점을 최적의 k로 채택
.inertia_: KMeans에서 자동으로 계산한 이너셔 값
# inertial 값은 inertia_에 계산되어 있음
inertia = []
for k in range(2, 7):
km = KMeans(n_clusters=k, n_init='auto', random_state=42)
km.fit(fruits_2d)
inertia.append(km.inertia_)
plt.plot(range(2, 7), inertia)
plt.xlabel('k')
plt.ylabel('inertia')
plt.show()
# 꺽이는 지점에서 n_cluster보다 늘려도 inertia 더 안 낮아지는 경우가 많음,
# 그래서 그 정도를 저걸한 k라고 봄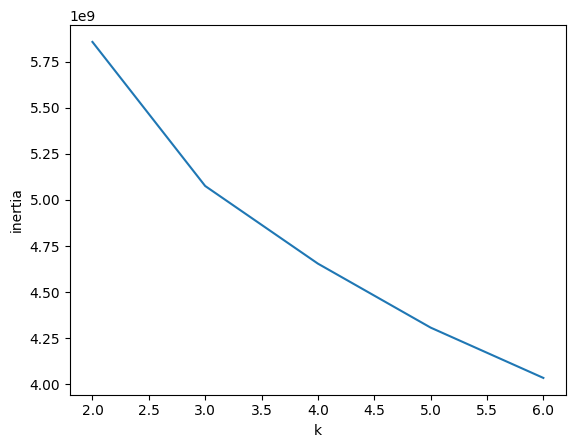
=> 이 방법도 명확하진 않아서 다른 지도학습이나 문제에 적용하면서 후작업을 진행하고 그 결과를 바탕으로 피드백하면서 모델을 개선하는 경우가 많음
06-3 주성분 분석
[키워드]
차원 축소
: 원본 데이터의 특성을 적은 수의 새로운 특성으로 변환하는 비지도 학습의 한 종류
- 저장 공간을 줄이고 시각화하기 쉬움
- 다른 알고리즘의 성능을 높일 수 있음
주성분 분석
: 차원 축소 알고리즘의 하나로 데이터에서 가장 분산이 큰 방향을 찾는 방법
- 원본 데이터를 주성분에 투영하여 새로운 특성을 만들 수 있음
- 일반적으로 주성분은 원본 데이터에 있는 특성 개수보다 작음
설명된 분산
: 주성분 분석에서 주성분이 얼마나 원본 데이터의 분산을 달 나타내는지 기록한 것
- 사이킷런의 PCA 클래스는 주성분 개수나 설명된 분산의 비율을 지정하여 주성분 분석을 수행가능
차원과 차원 축소
차원
: 데이터가 가진 속성 => 특성, 머신러닝에선 이 '특성'을 '차원'이라고 함
n차원 배열과 1차원 배열에서 용어가 다름
- n차원 배열: 차원 = 축의 개수
- 1차원 배열(벡터): 차원 = 원소으 ㅣ개수
=> '차원 축소'에서 축소하는 차원은 벡터로서의 차원(1차원 배열)
차원 축소
- 비지도학습의 한 종류로, 데이터를 가장 잘 나타내는 일부 특성을 선택하여 데이터 크기를 줄이고 다른 알고리즘의 성능을 향상시킬 수 있는 방법
- 특성 개수가 많아지면 과대적합되는데, 이를 막아줌
- 특성 개수가 줄어드니까 당연히 용량도 줄여줌.
- 시각화하기도 쉬워짐.
- 반대로, 줄어든 차원에서 원본 차원으로 복원할 수도 있음
- 대표적으로 주성분 분석(Principal Component Analysis)이 있음
주성분 분석(PCA)
첫번째 주성분
-
직관적으로 길게 늘어진 대각선 방향이 분산이 가장 크다고 불 수 있음
(= 가장 많이 퍼져있는 방향 = 데이터를 가장 잘 표현하는 방향)
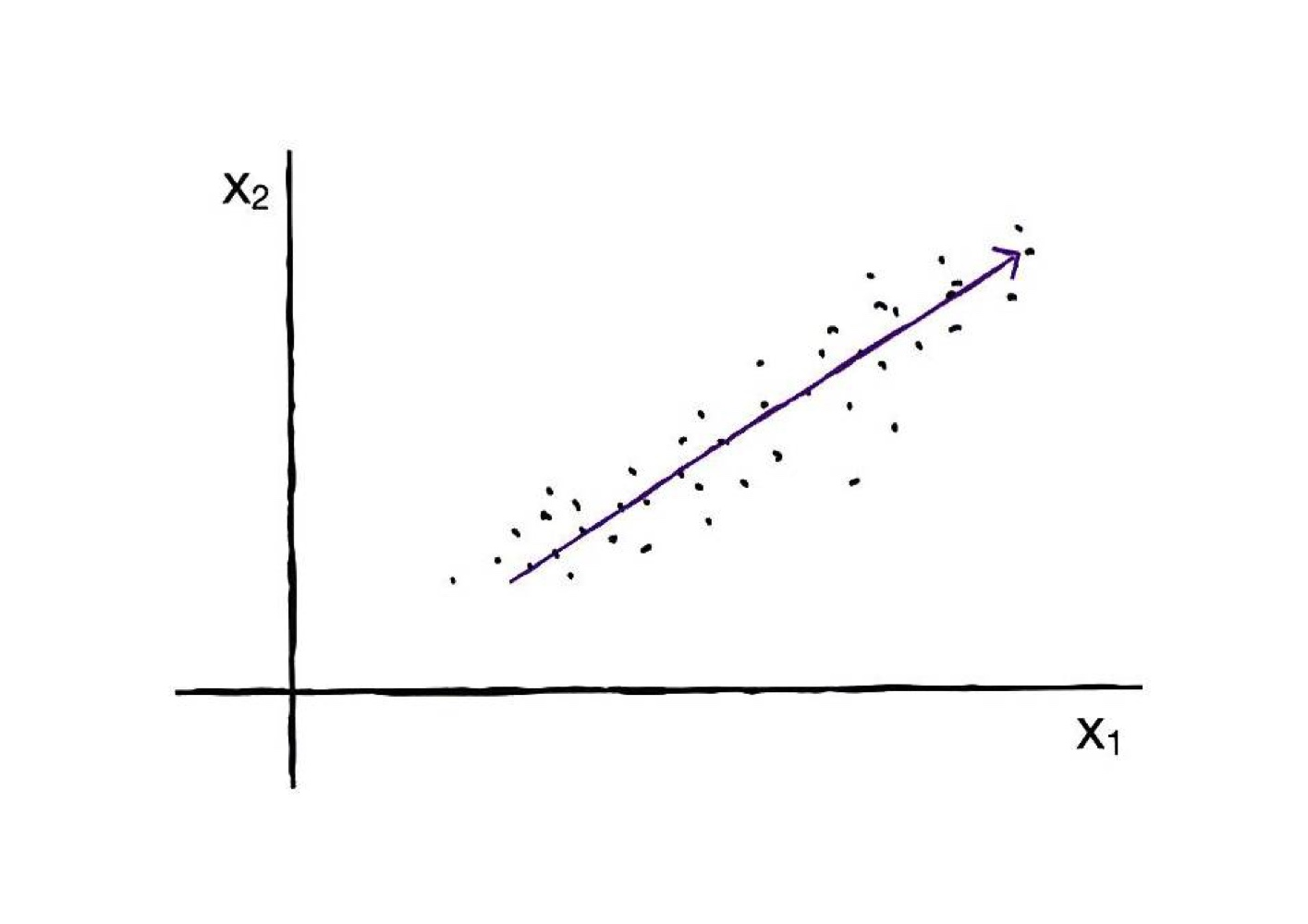
-
위 직선을 원점으로 옮기면 벡터로 표현하기 쉬워지고, 이 벡터 (2,1)이 주성분이 됨
-
특성은 X1, X2 로 2개였는데 ➡️ (2,1) 이라는 1개의 데이터로 표현가능
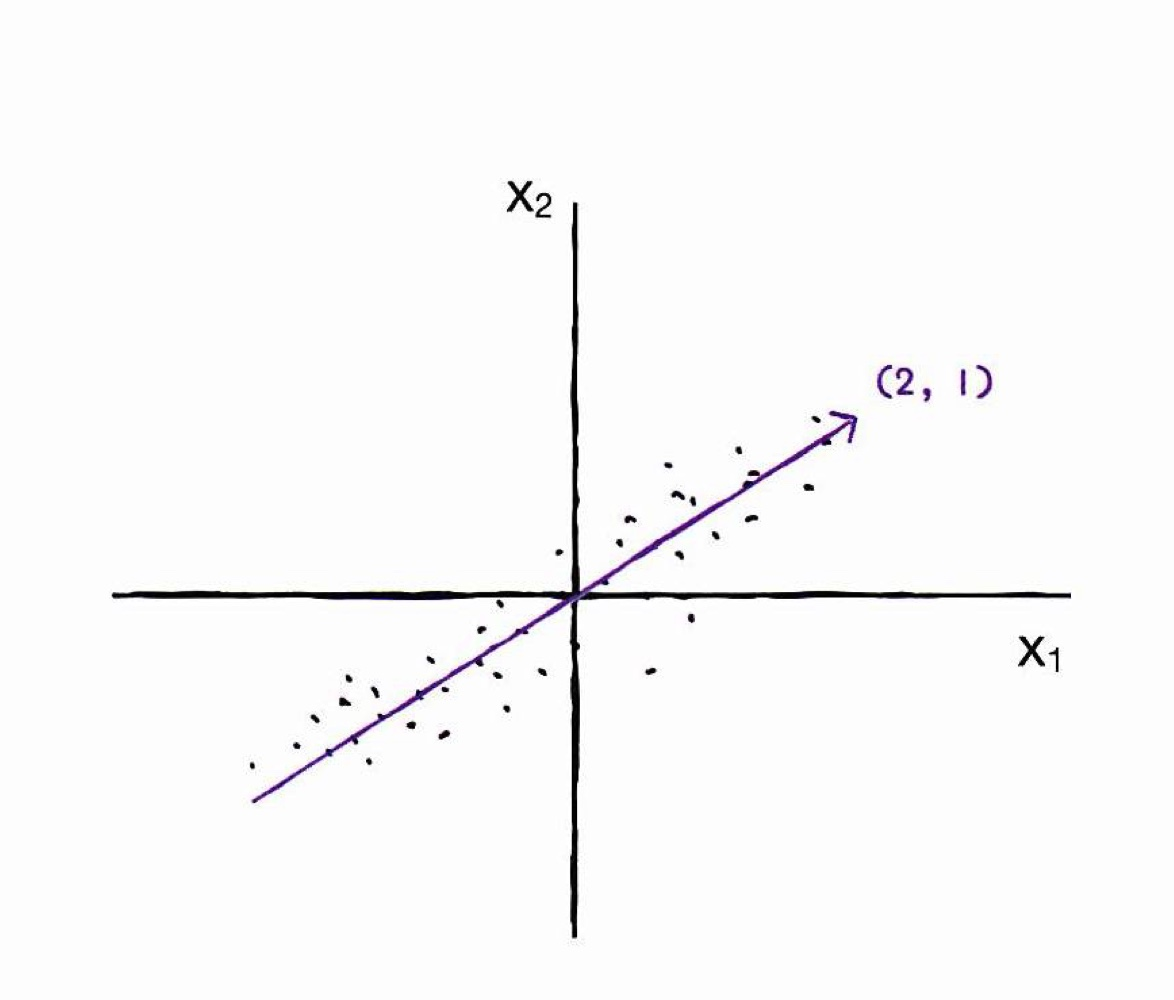
-
주성분(벡터)을 활용하면, 원본데이터의 차원을 줄일 수 있음
ex) s(4,2)라는 2차원 샘플을 "주성분에 투영하면" 1차원 데이터 p(4.5)로 만듦
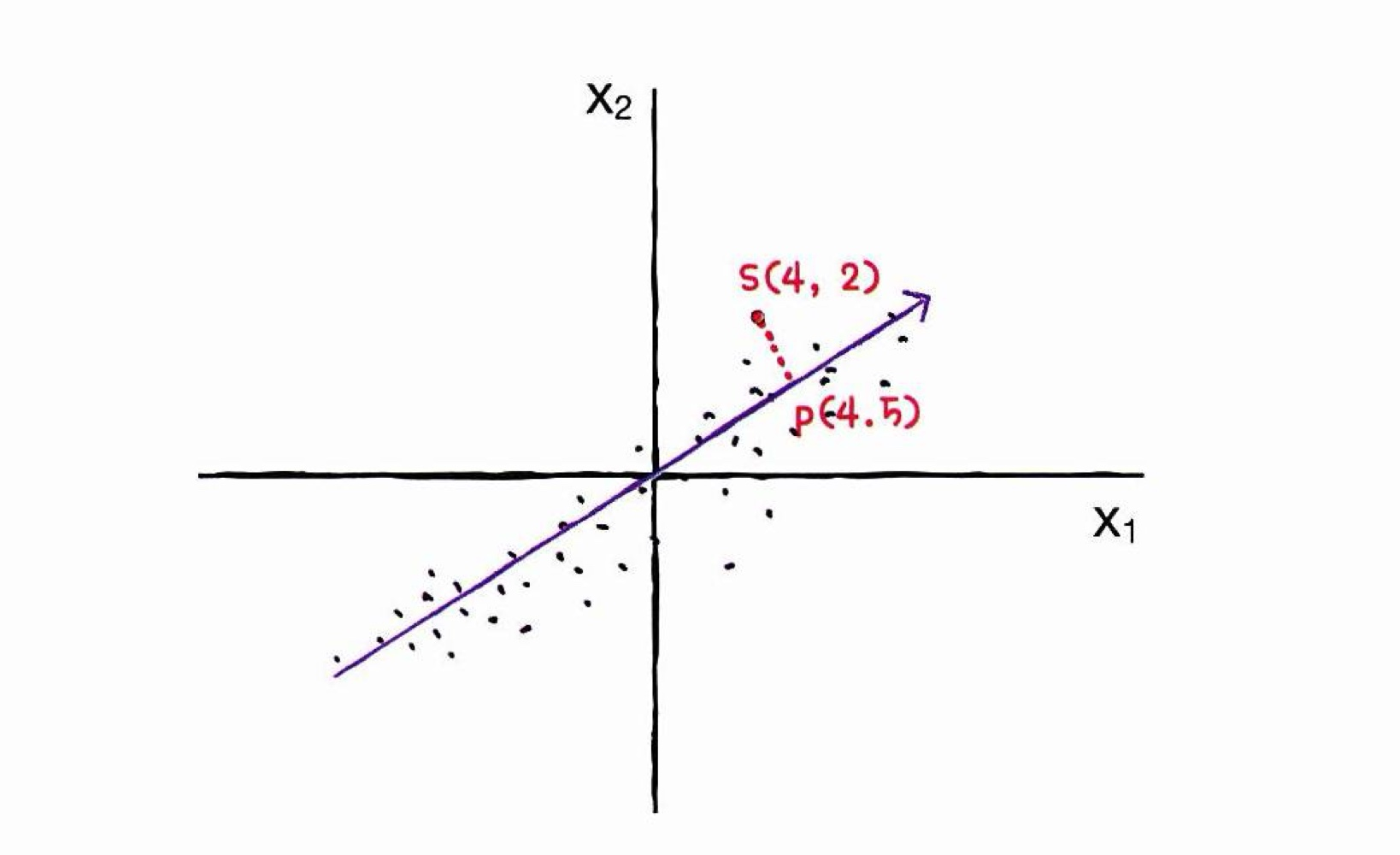
❗ 주성분을 원본차원과 같고 주성분으로 바꾼 데이터는 차원이 줄어들음
두번째 주성분
- 주성분은 원본의 특성 개수만큼 찾을 수 있음
(ex. 현재 그래프에서 X1, X2로 2차원이기 때문에 주성분 최대 2개까지 찾을 수 있음) - 첫 번째 주성분에 수직이면서, 분산이 가장 큰 다음 방향을 찾으면 됨
(첫번째 주성분이 표현하지 못하는 분산의 방향을 찾는 것이 효율적이기 때문)
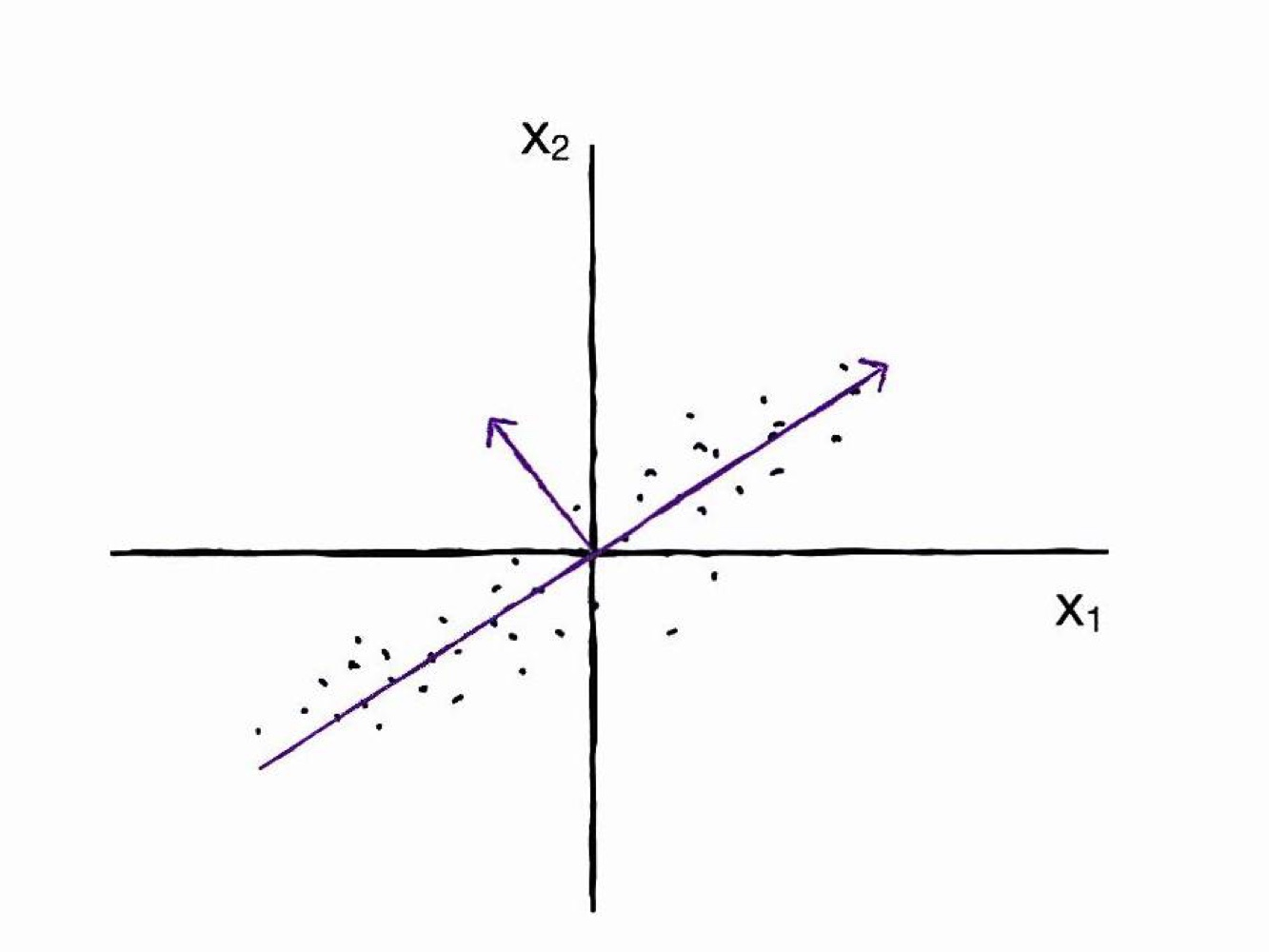
=> 원본 특성이 2개인데 주성분 2개를 다 찾으면 차원 축소가 안되는 셈이기 때문에 보통은 더 적은 개수의 주성분만 찾아서 활용
PCA 클래스
주성분 분석
- 이전 차시와 동일하게 데이터 준비
!wget https://bit.ly/fruits_300_data -O fruits_300.npy
import numpy as np
fruits = np.load('fruits_300.npy')
fruits_2d = fruits.reshape(-1, 100*100)- 비지도 학습이기 때문에 입력 데이터만 전달해서 모델 훈련
PCA(): 사이킷런에서 주성분 분석 알고리즘이 구현되어있는 클래스n_components: 찾을 주성분의 개수를 지정하는 매개변수
from sklearn.decomposition import PCA
pca = PCA(n_components=50)
pca.fit(fruits_2d).components_: 주성분이 저장되어 있음
print(pca.components_.shape)
# 찾은 주성분의 크기를 확인
# -> 50개의 주성분, 각 주성분들은 원소를 10000개씩 갖고 있음
->
(50, 10000)- 찾은 주성분 -> 이미지로 출력, 원본 데이터셋의 어떤 패턴을 읽은 것으로 보임
# 100x100으로 바꿔서
draw_fruits(pca.components_.reshape(-1, 100, 100))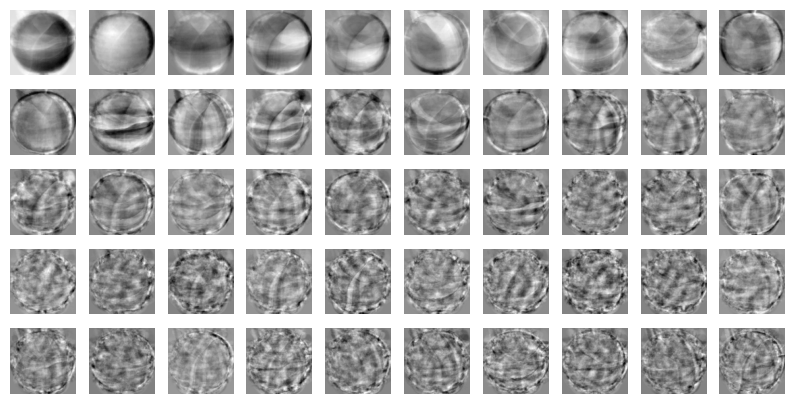
.transform(): 전달받은 데이터를 주성분으로 분해(차원 축소)하는 메소드
print(fruits_2d.shape) # 300개 샘플, 각각 10000개 특성
fruits_pca = pca.transform(fruits_2d) # 차원 축소
print(fruits_pca.shape) # 특성 개수 50개
->
(300, 10000)
(300, 50)원본 데이터 재구성
- (10000 -> 50), 반대로 원본 데이터로 복원(50 -> 10000) 가능
(압축 시 손실o -> 100% 복구 x) .inverse_transform(): 주성분을 바탕으로 원본 데이터를 재구성하는 메소드
fruits_inverse = pca.inverse_transform(fruits_pca)
print(fruits_inverse.shape)
->
(300, 10000)- 그림으로 출력, 거의 모든 과일이 복원됨(50 -> 10000, 일부 흐리거나 번진 부분 o)
fruits_reconstruct = fruits_inverse.reshape(-1, 100, 100)
for start in [0, 100, 200]:
draw_fruits(faruits_reconstruct[start:start+100])
print("\n")


=> 50개의 주성분이 원본의 분산을 매우 잘 나타내는 것을 의미
설명된 분산(explained variance)
: 주성분이 원본 데이터의 분산을 얼마나 잘 나타내는지 기록한 값
.explained_variance_ratio_: 각 주성분의 설명된 분산 비율이 저장됨
→ 이 값을 sum으로 다 더하면 주성분 50개가 설명하는 총 분산 비율을 얻을 수 있음
# 비율 50개를 다 더함 = 총 분산 비율
print(np.sum(pca.explained_variance_ratio_))
->
0.9215275787736402 (92%)- 설명된 분산 비율 그래프로 그림, 적절한 주성분 개수를 찾는데 도움됨
plt.plot(pca.explained_variance_ratio_)
plt.xlabel("pca")
plt.ylabel("explained variance ratio")
plt.show()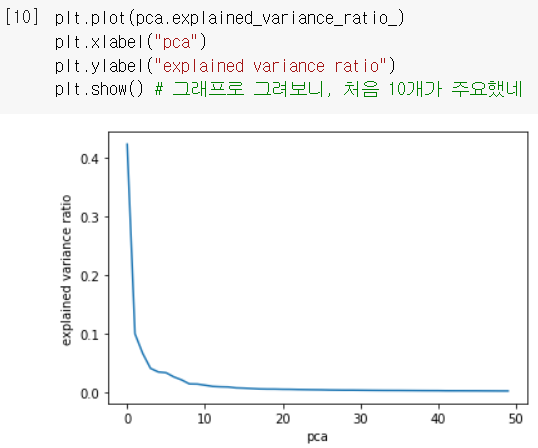
=> 처음 10개가 주성분을 가장 많이 설명, 주요했음을 알 수 있음
다른 알고리즘과 함께 사용하기
PCA는 주로 다른 알고리즘과 연계하여 많이 사용
분류 알고리즘 (로지스틱 회귀)
- 모델을 만들고 타겟값(사과 0/ 파인애플 1/ 바나나 2)을 설정
from sklearn.linear_model import LogisticRegression
lr = LogisticRegression()
target = np.array([0] * 100 + [1] * 100 + [2] * 100)
# 지도학습 모델이기 때문에 임의로 타겟값 만들어줌- 원본 데이터로 훈련해서 교차검증
from sklearn.model_selection import cross_validate
scores = cross_validate(lr, fruits_2d, target)
# 로지스틱 객체에 원본과 타깃 전달
print(np.mean(scores['test_score'])) # 점수 출력
print(np.mean(scores['fit_time'])) # 훈련하는데 걸린 시간 출력
->
0.9966666666666667
1.7471996784210204- PCA 차원 축소한 데이터
scores = cross_validate(lr, fruits_pca, target)
# 입력 데이터만 바꿈
print(np.mean(scores['test_score']))
print(np.mean(scores['fit_time']))
->
1.0
0.04023046493530273=> 특성 50개만 쓰면서 (성능은 유지) 훈련시간 감소
군집 알고리즘(k-평균)
- 모델을 만들고, (비지도학습이니까) 타깃값 없이 훈련
from sklearn.cluster import KMeans
km = KMeans(n_clusters=3, random_state=42) # KMeans 모델 만듦
km.fit(fruits_pca) # 2개의 특성 축소해놓은 데이터 fit
print(np.unique(km.labels_, return_counts=True))
# 구해진 클러스터, 각각 몇 개씩 묶였나 확인
->
(array([0, 1, 2], dtype=int32), array([110, 99, 91]))- 이미지로 출력해봄
for label in range(0, 3):
draw_fruits(fruits[km.labels_ == label])
print("\n")


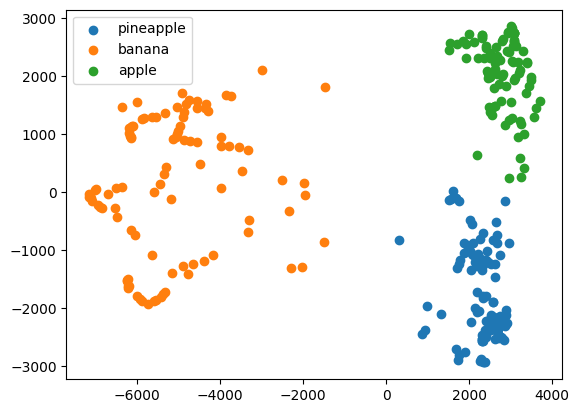
데이터 시각화
- 데이터를 차원축소 -> 시각화 가능
- 특성 2개 →
fruits_pca→ 3개의 과일 산점도
for label in range(0, 3):
data = fruits_pca[km.labels_ == label]
plt.scatter(data[:,0], data[:,1])
plt.legend(['pineapple', 'banana', 'apple'])
plt.show()