BioBERT : a pre-trained biomedical language representation model for biomedical text mining
논문
논문
Abstract
BERT는 범용학습 모델로 Biomedical 텍스트를 분석하면 성능이 떨어지는 경우가 있다. 이 논문에서는 Biomedical 텍스트를 분석하기 위해 BERT를 biomedical corpora로 Pretrain한 방법과 모델 BioBERT를 소개한다. BioBERT는 biomedical 과 관련된 task에서 BERT보다 높은 성능을 보인다.
Approach
BioBERT는 biomedical domain을 위하여 사전학습 된 language representation model이다.
1. pre-training
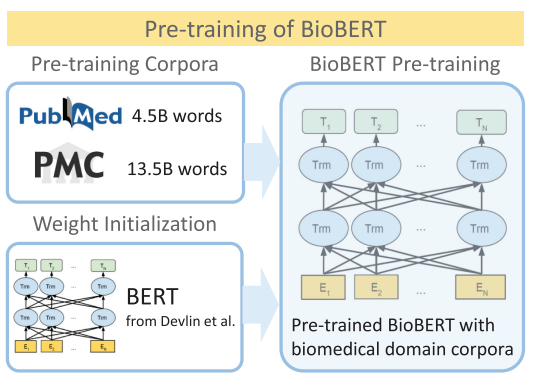
먼저, 범용적인 언어모델로 활용하기 위해 Wikipedia와 BooksCorpus로 학습된 BERT의 weights로 BioBERT의 weights를 초기화했다. 다음으로, BioBERT는 biomedical domain corpora로 학습을 진행했다.
- BioBERT는 8개의 NVIDIA V100 GPUs를 활용하여 23일동안 학습하였다.
- BioBERT가 BERT보다 biomedical task에 대해 높은 성능을 보였다.
- Architectural modifications을 최소화 하고도 성능을 향상시켰다.
2. find-tuning
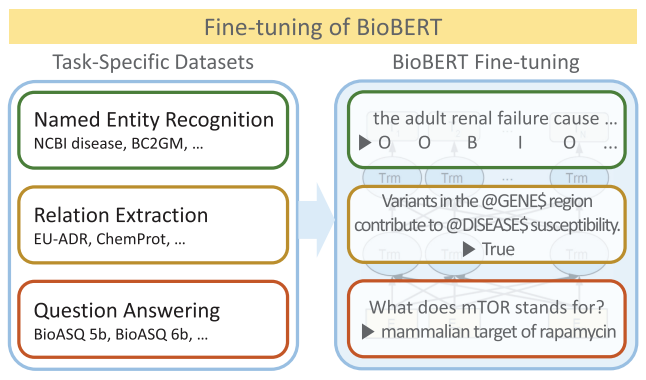
BioBERT의 모델 성능을 NER, RE, QA task로 평가했다. BioBERT 모델을 활용하면 biomedical domain data에 대한 performance가 기존 범용 모델인 BERT보다 뛰어난 것으로 나타났다.
Materials and methods
1. BERT
기본적으로 BioBERT의 모델 구조는 BERT와 동일하다. BERT에 대한 설명은 생략한다.
2. Pre-training BioBERT
Biomedical domain text는 domain에 특화된 명사를 많이 포함하고 있다. 이러한 특성 때문에 범용 언어 모델은 biomedical text mining texts에서 부족한 performance를 보이곤 한다. 이 문제를 해결하기 위해 BioBERT는 관련 데이터로 pre-training을 진행하였다.
BioBERT는 WordPiece Tokenizer을 활용했다. WordPiece Tokenizer을 활용하면 신조어가 등장하더라도 subword를 조합하여 이를 표현할 수 있다. (e.g. Immunoglobulin → I##mm##uno##g##lo##bul##in)
BioBERT에서는 Original BERT의 WordPiece Vocabulary을 사용했는데, 이는 Tokenizer의 특성으로 out-of-vocabulary 문제를 어느정도 해결할 수 있고, 범용 언어모델을 위해 학습된 BERT를 기반으로 BioBERT를 학습할 수 있기 때문이었다.
3. Fine-tuning
Named entity recognition, relation extraction, question answering task에 대해 fine-tuning을 진행했다.
Results
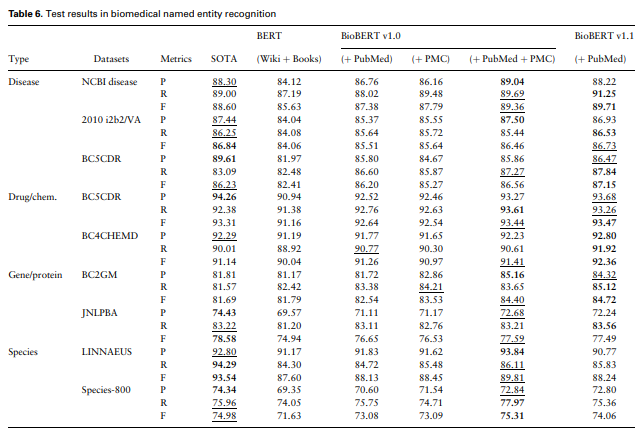
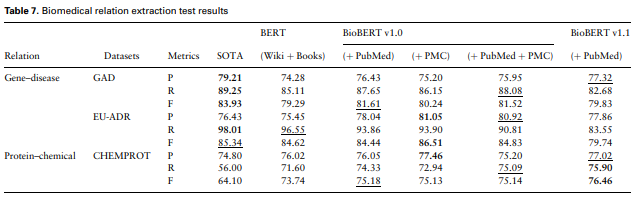
BioBERT는 BERT의 성능을 상회했다.
결론
BERT는 범용 언어모델이기 때문에 domain에 특화된 텍스트의 경우 성능이 떨어질 수 있다. 의료, 기술 관련 데이터를 다루게 되는 경우, BERT에 대해 pretrain을 추가로 진행하여 task를 수행하는 것이 좋아보인다.
