[Paper Review] Inferring Novel Tumor Suppressor Genes with a Protein-Protein Interaction Network and Network Diffusion Algorithms
Paper Review
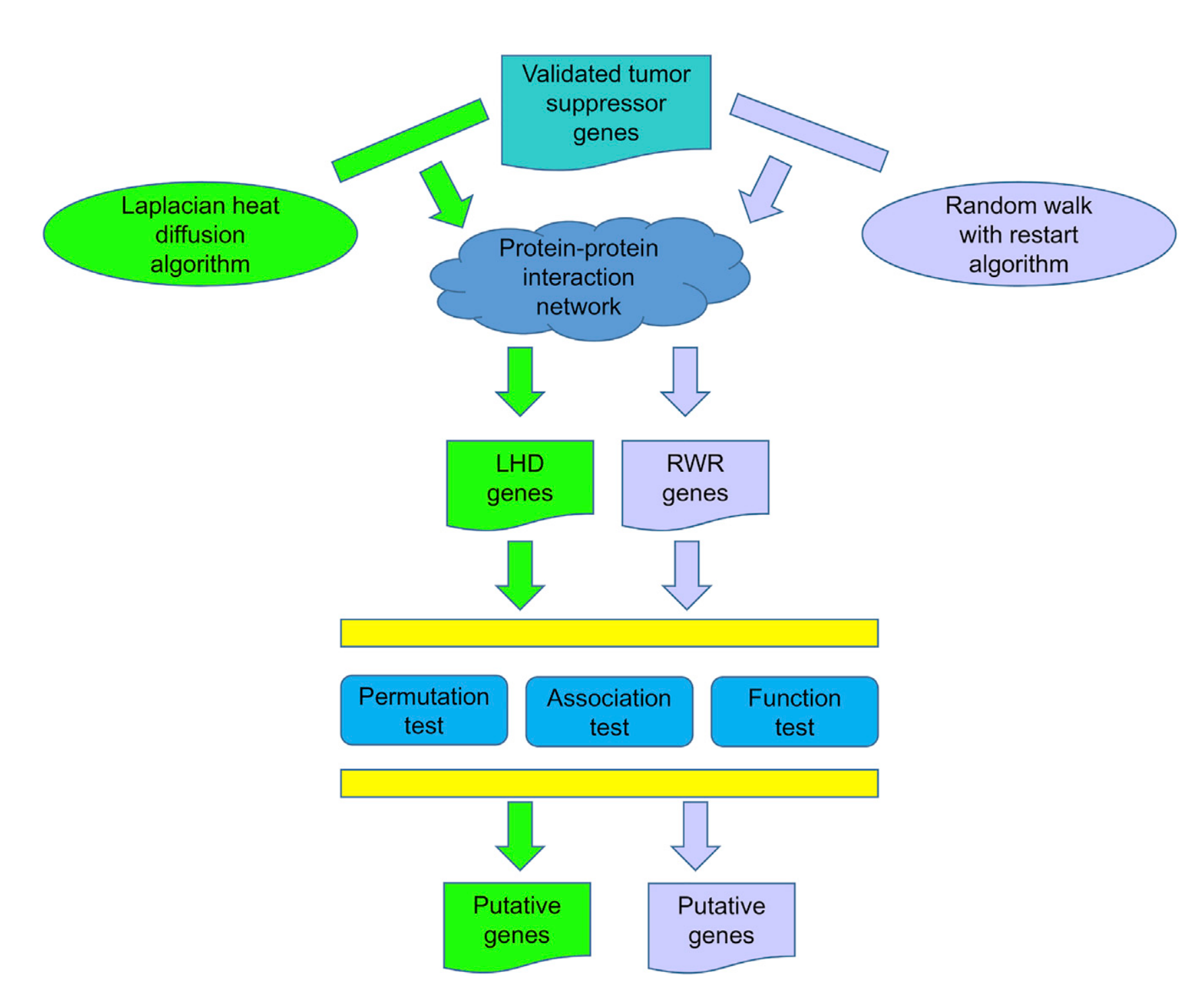
이 논문을 리뷰하는 이유는 전반적인 framework이 심플하며 앞으로 진행할 프로젝트에 가볍지만 의미 있는 수준으로 적용할 수 있을 것 같아서임. 이 리뷰에서는 모든 내용을 커버하는 것이 아닌 개인적으로 관심있는 부분만 할 것임.
Chen, Lei, et al. "Inferring novel tumor suppressor genes with a protein-protein interaction network and network diffusion algorithms." Molecular Therapy-Methods & Clinical Development 10 (2018): 57-67.
Introduction
특별한 내용이 없으므로 생략
Results
 Figure1. The procedures of the LHD-Based and RWR-Based Methods
Figure1. The procedures of the LHD-Based and RWR-Based Methods
Putative Genes Yielded by the LHD-based Method
LHD-method를 통해서 1차적으로 2,874개의 genes을 뽑아낸 다음 three tests (permutation test, association test, function test)를 통해 필터링하여 41개의 putative genes을 추려냈음
Putative Genes Yielded by the RWR-based Method
LHD-method와 유사한 방법으로 진행했으며, RWR-method를 통해 1차적으로 뽑아낸 5,889 genes에 three test를 적용하여 140개의 putative genes를 추려냈음

Comparison of Putative Genes Yielded by the Different Methods

서로 다른 methods를 이용해서 알아낸 putative genes은 많이 겹치지 않음. method별 putative genes set과 각 method간의 교집합은 figure 2와 같음.
Discussion
여기서는 사실 자기네들이 찾은 putative genes에 대한 evidence의 나열이라서 특별히 언급하지 않고 넘어가겠음
Materials and Methods
Validated TSGs
TSGene database(https://bioinfo.uth.edu/TSGene/)에 있는 716개의 human TSGs 중 PPI network에 등장하지 않는 85개를 제외한 631개의 human TSGs를 사용하였음
PPI Network
STRING(https://string-db.org/, version 10.0)을 사용하였음. STRING database는 19,247개의 Ensembl ID간의 4,274,001개의 interaction으로 이루어져 있으며 각 interaction은 150~999의 값을 가짐. 여기서 높은 score는 동시에 발생할 확률이 높다는 것을 의미함.
Searching Candidate Genes Using the LHD Algorithm
LHD가 무엇이고 어떻게 썼는지는 관심사가 아니라서 넘어감.
Searching Candiate Genes Using the RWR Algorithm
RWR이 무엇이고 어떻게 썼는지는 관심사가 아니라서 넘어감.
Screening Tests
LHD나 RWR은 network의 structure에 영향을 많이 받기 때문에 LHD와 RWR로 찾은 putative genes는 생물학적으로 의미 없는 false positive일 수도 있음. 이를 보완하기 위해 3가지 screening test는 제안함. false-positve를 컨트롤하기 위한 permutation test와 중요한 genes을 추출하기 위한 association test와 function test를 통해 screening test를 진행함.
Permutation test
- 631개의 gene을 랜덤 샘플링을 m번 수행하여 sub set을 만들고 각 sub set을 seed node로 하여 LHD 혹은 RWR을 적용
- m=1,000 으로 둔거 같은데.. 631이나 1000이라는 숫자에 대한 근거는 따로 없음
- 이 후 각 gene마다 원래 LHD 또는 RWR를 통해 얻어진 값과 m개의 sub set에서 얻어진 값과 비교해본다. 이 때 sub set에서 얻어진 값이 더 큰 경우가 많다면 해당 gene은 false-postive라 판단할 수 있고, 해당 경우의 비율이 0.05보다 낮은 gene만 사용함
Association test
- Permutation test를 통과한 gene을 대상으로 진행
- validated TSGs의 function들과 얼마나 유사한지를 판단함.
- max(interaction scores btw candidate gene and validated TSGs)
- 위 값에 cut off를 적용해서 스크리닝
Function test
- 두 gene의 GO terms과 KEGG pathway를 비교하여 score를 만드는데.. enrichment theory를 근거로 하고 있음(그게 뭐지?)
- 뭐.. 위 내용을 반영해서 여차저차 하면 각 gene별로 vector를 만들어 낼 수 있는데, 두 gene vectors의 cosine similarity를 두 gene의 functional score 정의함.
- association test와 비슷하게 putative gene과 validated TGSs간의 functional score의 max 값을 이용하여 스크리닝 함
이 논문에서 개인적으로 참고할만한 내용
- STRING database와 TSGene database에 LHD나 RWR보다 더 나아간 모델을 적용해서 PoC 해볼 수 있을 것 같음
- screening test는 간단하지만 효과적인 방법인 것 같아서 이용할 가치가 있다고 판단됨
- 그래도.. 여자저차해서 찾은 cadidates에 대해 하나하나 문헌검증 해야하는건 좀 귀찮은 일인듯ㅠㅠ