11. 불균형 데이터
11.1 불균형 데이터
불균형 데이터
- 분류 문제에서 타겟 데이터의 범주가 한 쪽으로 치우친 데이터
- 일반적으로 정상 데이터에 비해 이상 데이터가 매우 적은 경우
- ex1. 양품 데이터에 비해 불량 데이터가 적은 경우- ex2. 중상 사고 데이터에 비해 사망사고 데이터가 적은 경우
불균형 데이터의 문제점
- 불균형 데이터는 모델의 성능을 왜곡시킨다
- 정확도가 높아도, 이상 데이터는 검출해내지 못한다.- 정확도 외에 다른 성능 지표를 활용해야 한다.
- 이상 데이터에 과적합될 가능성이 높다.
- 이상 데이터가 적기 때문에 이상 데이터에 과적합 될 가능성 높음
11.2 불균형 데이터의 해결방안
1. minority class 데이터를 추가 수집
- 현실적으로 수집이 어려움
- 수집에 시간과 비용이 많이 필요함
2. 모델의 성능 평가 지표를 변경
- 정확도 대신에 다른 성능 평가지표를 활용해야함
- Precision, Recall, F1-score, ROC-AUC, PR-AUC 등
3. 샘플링 기법으로 클래스별 데이터 균형을 맞춤
3.1 Undersampling
- majority class의 인스턴스를 일부만 사용하여 minority class의 인스턴스와 양적 균형을 맞춤
- 데이터 소실이 매우 크고, 중요한 정상 데이터를 잃을 수 있음
3.2 Oversampling
- minority class의 인스턴스를 복제 또는 유사 인스턴스를 생성하여 인스턴스수를 증가시키는 방법
- 정보가 손실되지 않는다는 장점이 있으나 과적합(Over-fitting)을 초래할 수 있음
예) - 인스턴스 복제: random over-sampling
- 유사 인스턴스 생성: SMOTE(Synthetic Minority Over-sampling Technique), ADASYN(Adaptive Synthetic Sampling Approach), Borderline-SMOTE
3.3 Undersampling + Oversampling 혼합
- majority class의 인스턴스는 일부만 사용하고 minority class의 인스턴스는 복제 또는 유사 인스턴스를 생성하여 균형을 맞춤
- 예) SMOTE + Tomek Links
4. 불균형 데이터에 강건한 모델 적용
- 앙상블(Ensemble) 모델 적용
- 앙상블 모델은 서로 다른 여러 가지 모형들의 예측/분류 결과를 종합하여 최종적인 의사결정에 활용하는 기법
- 여러 개의 모델을 학습할 때, 기존의 데이터를 균등하게 여러 데이터 셋으로 분류하여 학습을 수행
- 과적합 감소효과 및 성능향상 가능
- 예) Decision Tree, Random Forest, XGBoost, LightGBM, CatBoost, …
11.3 불균형 데이터 처리 방안
imbalanced learn 패키지
imbalanced-learn은 클래스 간 불균형을 보여주는 데이터 세트에서 일반적으로 사용되는 여러 리샘플링 기술을 제공하는 Python 패키지
다양한 샘플링 기법 비교
분류 문제 생성함수 정의
import warnings
# hide warnings
warnings.filterwarnings("ignore")
from sklearn.datasets import make_classification
# 분류 문제 데이터 생성하는 함수 정의, 독립변수 2개, 범주 x개
def create_dataset(n_samples, weights):
return make_classification(
n_samples=n_samples,
n_features=2, n_informative=2, n_redundant=0,
n_classes=len(weights), n_clusters_per_class=1, weights=weights,
random_state=0)결과 그래프 출력 함수 정의
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
# 그래프 스타일 설정
sns.set_context("paper", font_scale=1)
# 분류기에 따른 결정영역을 표시하는 함수 정의
def plot_decision_results(X, y, clf, ax, title=None):
# 그리드 간격 설정
grid_step = 0.02
x_min, x_max = X[:, 0].min() - 1, X[:, 0].max() + 1 # X의 범위
y_min, y_max = X[:, 1].min() - 1, X[:, 1].max() + 1 # y의 범위
# meshgrid 함수를 사용하여 그리드 포인트 생성
xx, yy = np.meshgrid(
np.arange(x_min, x_max, grid_step), np.arange(y_min, y_max, grid_step)
)
# 예측 모델에 그리드 포인트 데이터를 입력하여 결정 영역을 계산
# meshgrid 함수의 결과인 2차원 배열을 1차원 배열로 펼쳐서(.ravel()) 수평으로 쌓음(np.c_[])
Z = clf.predict(np.c_[xx.ravel(), yy.ravel()])
# 예측 결과를 그리드 포인트의 데이터 형태로 재배열
Z = Z.reshape(xx.shape)
# 그리드 포인트의 등고선을 그림
ax.contourf(xx, yy, Z, alpha=0.4)
# 클래스의 산점도를 그림
ax.scatter(X[:, 0], X[:, 1], alpha=0.8, c=y, edgecolor="k")
if title is not None:
ax.set_title(title + f'\n({len(y[y==0])}, {len(y[y==1])}, {len(y[y==2])})', fontsize=12)11.3.1 Undersampling
소수 클래스의 수를 고려하여 데이터 전체 표분의 크기를 축소시켜 균형을 맞추는 방법
1) Random UnderSampling
무작위로 majority class의 인스턴스를 제거하여 균형을 맞춤
샘플링시마다 결과가 다름
2) SMOTE(Synthetic Minority Over-Sampling Technique)
- majority class에서 중요하지 않은 인스턴스를 제거하는 것
- K-Means 알고리즘 활용
- 군집화(Clustering)하여 군집의 중심점(Centroid)을 찾고, 중심점과 가장 가까운 인스턴스만 보존
3) Edited Nearest Neighbours
- majority class 인스턴스 중 가장 가까운 k개의 인스턴스가 모두 또는 다수가 majority class가 아니면 삭제
- minority class 주변의 majority class 인스턴스는 삭제됨
- 주요 목적은 minority class 인스턴스의 분류 정확도를 높이는 것
4) Condensed Nearest Neighbour
- 1-NN(1-Nearest Neighbour) 알고리즘을 사용하여 1-NN 모형으로 분류되지 않는 인스턴스만 보존
- majority class에 밀집된 데이터가 없을 때까지 인스턴스를 제거하여 대표적인 인스턴스만 남도록 하는 방법
5) Tomek Links
서로 다른 클래스에 속하는 한 쌍의 인스턴스 중에서 가장 가까운 인스턴스(경계선에 있는 인스턴스)를 찾고, 그 중에서 majority class에 속하는 인스턴스를 제거
6) Near Miss
majority class에 속하는 인스턴스 중에서 minority class에 가까운 인스턴스를 보존
import numpy as np
from sklearn.linear_model import LogisticRegression
from imblearn.under_sampling import RandomUnderSampler, TomekLinks, EditedNearestNeighbours
from imblearn.under_sampling import CondensedNearestNeighbour, NearMiss, ClusterCentroids
# 시드 설정
np.random.seed(0)
# 데이터 생성
X, y = create_dataset(n_samples=200, weights=(0.05, 0.25, 0.7))
# 샘플러 설정
samplers = [RandomUnderSampler(),
TomekLinks(),
EditedNearestNeighbours(),
CondensedNearestNeighbour(n_neighbors=3),
NearMiss(version=1, n_neighbors=3),
NearMiss(version=2, n_neighbors=3),
NearMiss(version=3, n_neighbors=3),
ClusterCentroids()
]
# 서브 그래프 설정
fig, axs = plt.subplots(nrows=3, ncols=3, figsize=(10, 10))
# 분류기 생성
clf = LogisticRegression()
clf.fit(X, y)
plot_decision_results(X, y, clf, axs[0,0], title="Without resampling")
for i, sampler in enumerate(samplers):
X_resampled, y_resampled = sampler.fit_resample(X, y)
clf.fit(X_resampled, y_resampled)
plot_decision_results(X_resampled, y_resampled, clf, axs[(i+1)//3,(i+1)%3], f"Using {sampler.__class__.__name__}")
fig.suptitle(f"Decision function of {clf.__class__.__name__}")
fig.tight_layout()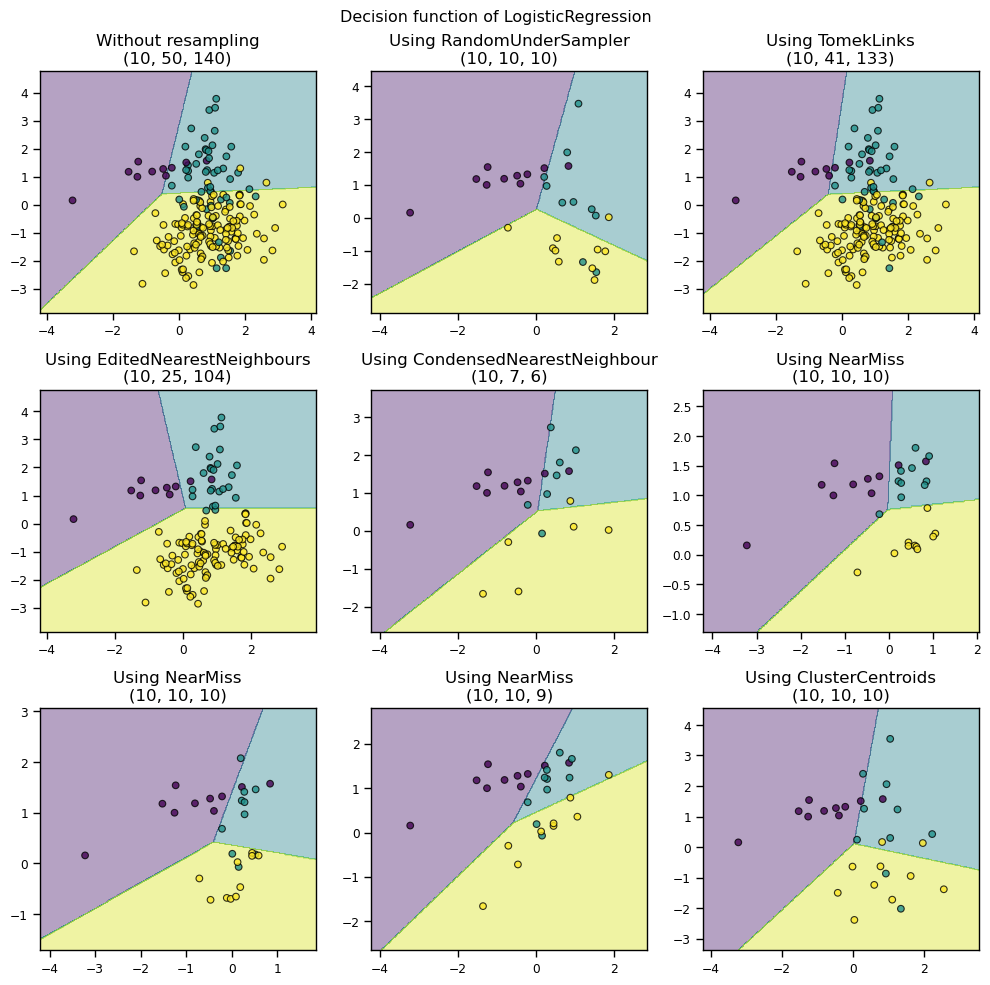
11.3.2 Oversampling
소수 클래스의 수를 다수 클래스의 수에 맞춰 증가시키는 방법
1) Random OverSampling
무작위로 minority class의 인스턴스를 복제하여 균형을 맞춤
동일하게 복제되기 때문에 과적합 발생에 주의해야함
2) SMOTE(Synthetic Minority Over-Sampling Technique)
각 minority class 인스턴스의 K-최근접 이웃을 찾고 그 중 하나를 무작위로 선택한 다음 선형 보간을 계산하여 이웃에 새로운 minority class 인스턴스를 생성
모델의 과적합을 방지하는데에 유용함
3) ADASYN (Adaptive Synthetic Sampling Approach)
모든 minority class 인스턴스에 대해 K-최근접 이웃을 측정한 다음 minority class 인스턴스와 majority class 인스턴스의 클래스 비율을 계산하여 새 인스턴스를 생성
4) Borderline-SMOTE
경계선(borderline)에 있는 인스턴스만 SMOTE를 적용
5) SVM-SMOTE
경계선(borderline)에 있는 인스턴스만 SMOTE를 적용
6) SMOTE + ENN
SMOTE를 사용하여 minority class의 데이터를 생성하고, 그 중에서 majority class에 속하는 데이터를 Edited Nearest Neighbours를 이용하여 제거
import numpy as np
from sklearn.linear_model import LogisticRegression
from imblearn.over_sampling import RandomOverSampler, SMOTE, ADASYN, BorderlineSMOTE, SVMSMOTE, KMeansSMOTE
from imblearn.combine import SMOTETomek, SMOTEENN
# 시드 설정
np.random.seed(0)
# 데이터 생성
X, y = create_dataset(n_samples=200, weights=(0.05, 0.25, 0.7))
# 샘플러 설정
samplers = [RandomOverSampler(),
SMOTE(),
ADASYN(),
BorderlineSMOTE(),
SVMSMOTE(),
KMeansSMOTE(),
SMOTETomek(),
SMOTEENN()
]
# 서브 그래프 설정
fig, axs = plt.subplots(nrows=3, ncols=3, figsize=(10, 10))
# 분류기 생성
clf = LogisticRegression()
clf.fit(X, y)
plot_decision_results(X, y, clf, axs[0,0], title="Without resampling")
for i, sampler in enumerate(samplers):
X_resampled, y_resampled = sampler.fit_resample(X, y)
clf.fit(X_resampled, y_resampled)
plot_decision_results(X_resampled, y_resampled, clf, axs[(i+1)//3,(i+1)%3], f"Using {sampler.__class__.__name__}")
fig.suptitle(f"Decision function of {clf.__class__.__name__}")
fig.tight_layout(pad=1.5)
plt.show()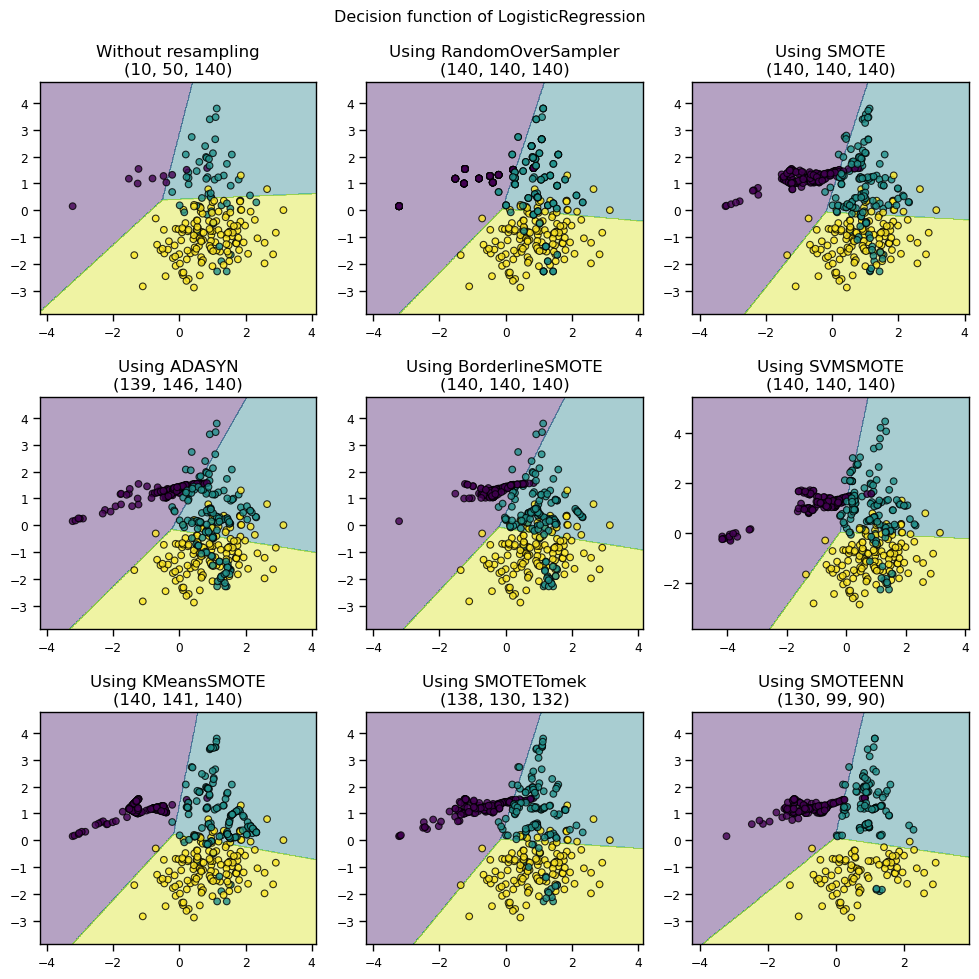
11.3.3 Undersampling + Oversampling
1) SMOTE + Tomek Links
- SMOTE를 사용하여 minority class의 데이터를 생성하고, 그 중에서 majority class에 속하는 데이터를 Tomek Links를 이용하여 제거
2) SMOTE + ENN
- SMOTE를 사용하여 minority class의 데이터를 생성하고, 그 중에서 majority class에 속하는 데이터를 Edited Nearest Neighbours를 이용하여 제거
import numpy as np
from sklearn.linear_model import LogisticRegression
from imblearn.over_sampling import RandomOverSampler, SMOTE, ADASYN, BorderlineSMOTE, SVMSMOTE, KMeansSMOTE
from imblearn.combine import SMOTETomek, SMOTEENN
# 시드 설정
np.random.seed(0)
# 데이터 생성
X, y = create_dataset(n_samples=200, weights=(0.05, 0.25, 0.7))
# 샘플러 설정
samplers = [RandomOverSampler(),
SMOTE(),
ADASYN(),
BorderlineSMOTE(),
SVMSMOTE(),
KMeansSMOTE(),
SMOTETomek(),
SMOTEENN()
]
# 서브 그래프 설정
fig, axs = plt.subplots(nrows=3, ncols=3, figsize=(10, 10))
# 분류기 생성
clf = LogisticRegression()
clf.fit(X, y)
plot_decision_results(X, y, clf, axs[0,0], title="Without resampling")
for i, sampler in enumerate(samplers):
X_resampled, y_resampled = sampler.fit_resample(X, y)
clf.fit(X_resampled, y_resampled)
plot_decision_results(X_resampled, y_resampled, clf, axs[(i+1)//3,(i+1)%3], f"Using {sampler.__class__.__name__}")
fig.suptitle(f"Decision function of {clf.__class__.__name__}")
fig.tight_layout(pad=1.5)
plt.show()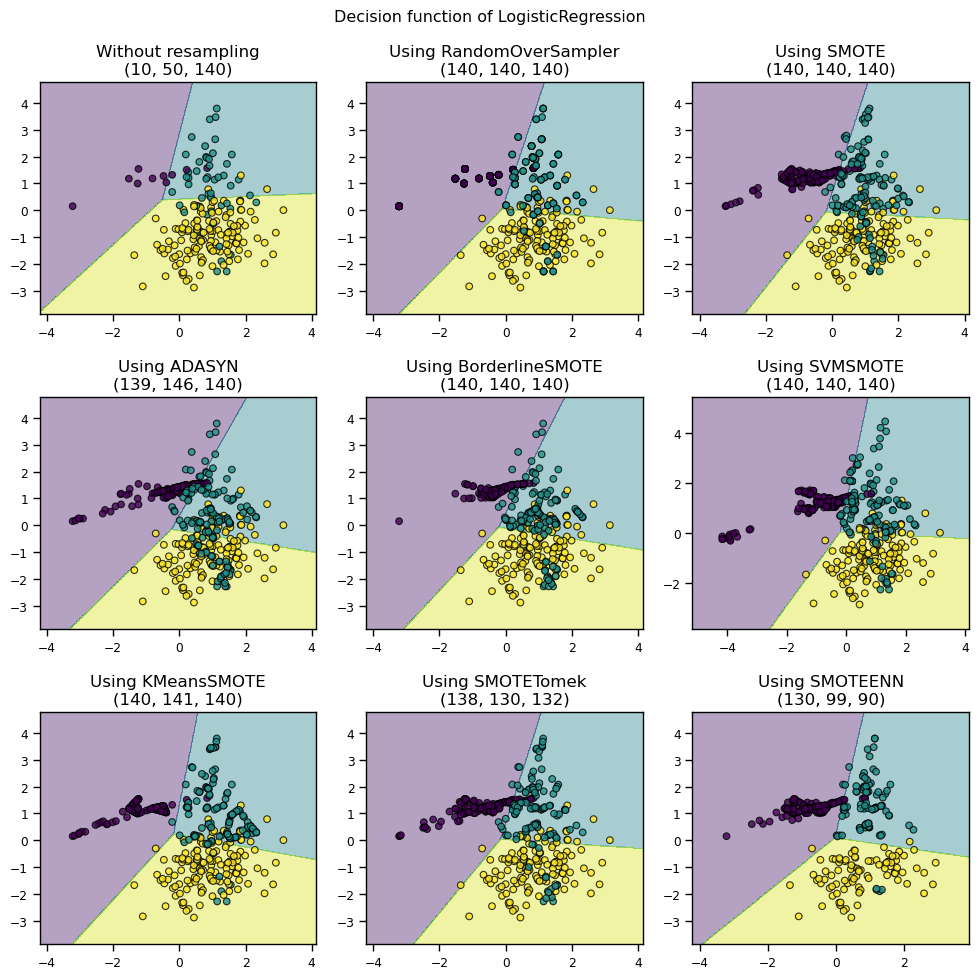
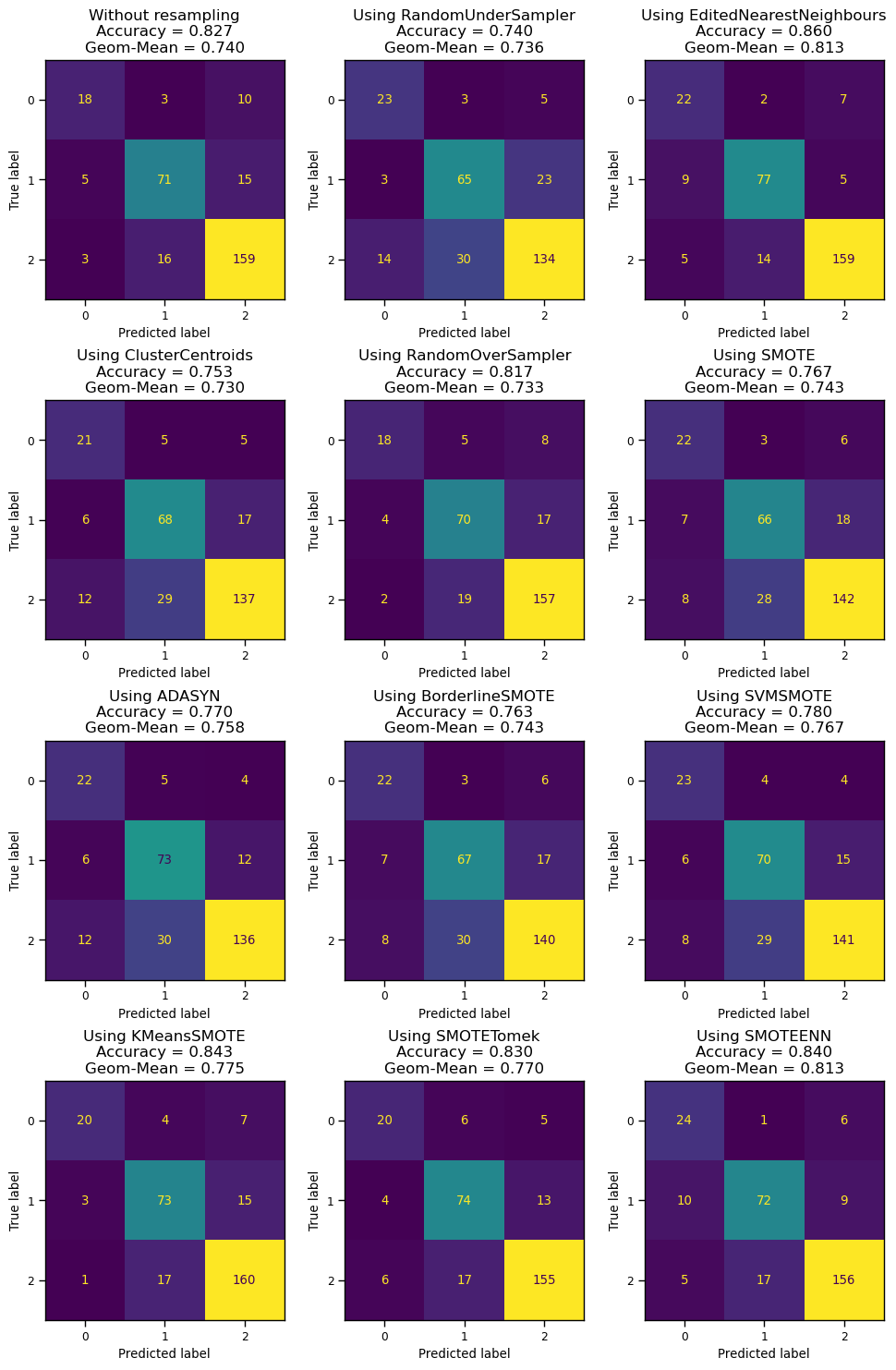
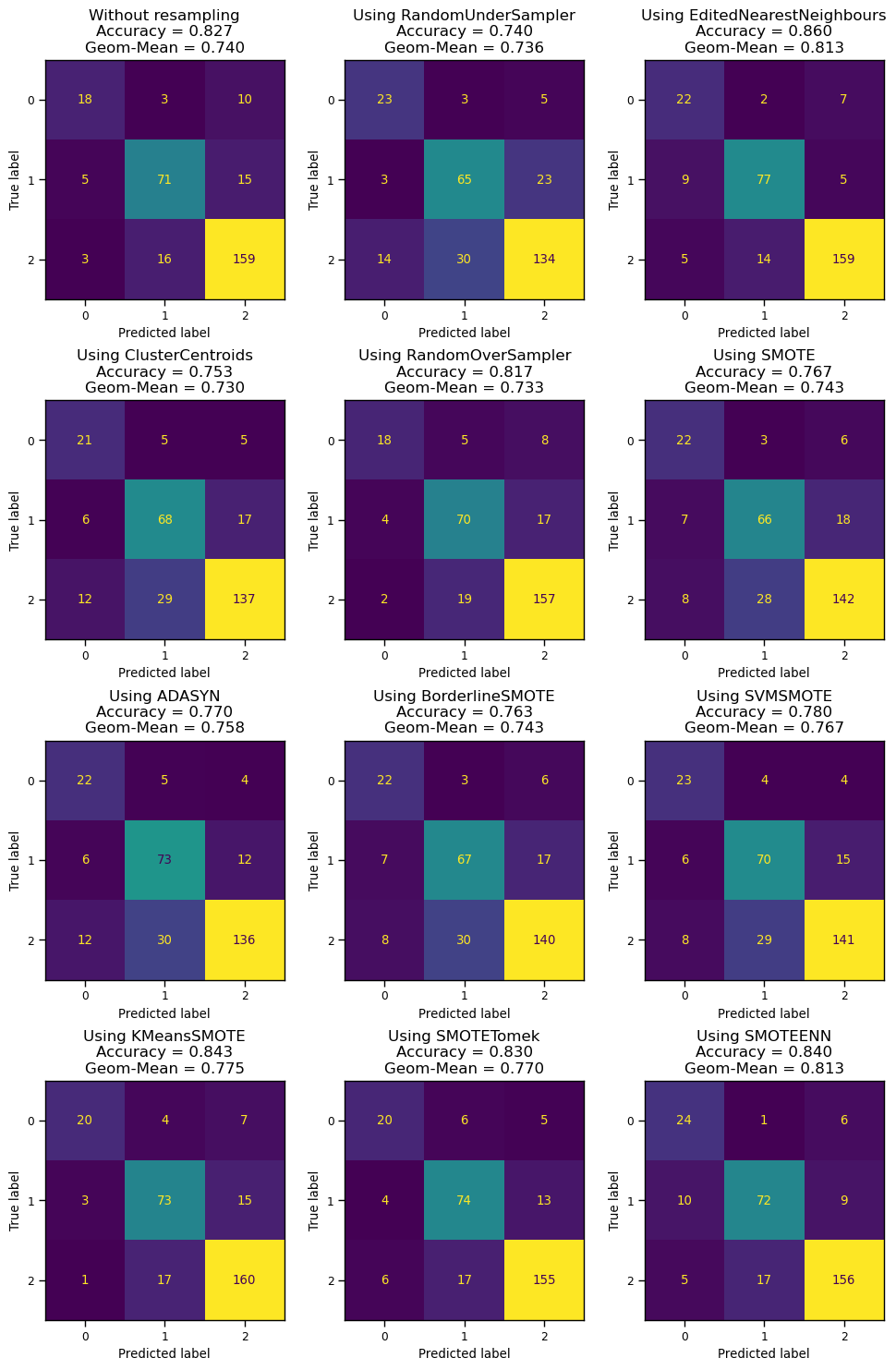
11.3.4 샘플링 기법 성능 비교
import numpy as np
from sklearn.metrics import ConfusionMatrixDisplay
from sklearn.model_selection import train_test_split
from sklearn.tree import DecisionTreeClassifier
from imblearn.under_sampling import RandomUnderSampler, TomekLinks, EditedNearestNeighbours, NearMiss, ClusterCentroids
from imblearn.over_sampling import RandomOverSampler, SMOTE, ADASYN, BorderlineSMOTE, SVMSMOTE, KMeansSMOTE
from imblearn.combine import SMOTETomek, SMOTEENN
from imblearn.metrics import geometric_mean_score
# 시드 설정
np.random.seed(0)
# 데이터 생성
X, y = create_dataset(n_samples=1000, weights=(0.1, 0.3, 0.6))
# 데이터셋 분리
X_train, X_test, y_train, y_test = train_test_split(X, y, stratify=y, test_size=0.3, random_state=0)
# 샘플러 설정
samplers = [RandomUnderSampler(),
EditedNearestNeighbours(),
ClusterCentroids(),
RandomOverSampler(),
SMOTE(),
ADASYN(),
BorderlineSMOTE(),
SVMSMOTE(),
KMeansSMOTE(),
SMOTETomek(),
SMOTEENN()
]
# 분류기 생성
clf = DecisionTreeClassifier()
# 서브 그래프 설정
fig, axs = plt.subplots(nrows=4, ncols=3, figsize=(10, 15))
clf.fit(X_train, y_train)
disp = ConfusionMatrixDisplay.from_estimator(clf, X_test, y_test, ax= axs[0,0], colorbar=False)
accuracy = clf.score(X_test, y_test)
geom_mean = geometric_mean_score(y_test, clf.predict(X_test))
disp.ax_.set_title(f"Without resampling\nAccuracy = {accuracy:.3f}\nGeom-Mean = {geom_mean:.3f}", fontsize=12)
for i, sampler in enumerate(samplers):
X_resampled, y_resampled = sampler.fit_resample(X_train, y_train)
clf.fit(X_resampled, y_resampled)
accuracy = clf.score(X_test, y_test)
geom_mean = geometric_mean_score(y_test, clf.predict(X_test))
disp = ConfusionMatrixDisplay.from_estimator(clf, X_test, y_test, ax=axs[(i+1)//3,(i+1)%3], colorbar=False)
disp.ax_.set_title(f"Using {sampler.__class__.__name__}\nAccuracy = {accuracy:.3f}\nGeom-Mean = {geom_mean:.3f}", fontsize=12)
fig.tight_layout(pad=2.0)
plt.show()
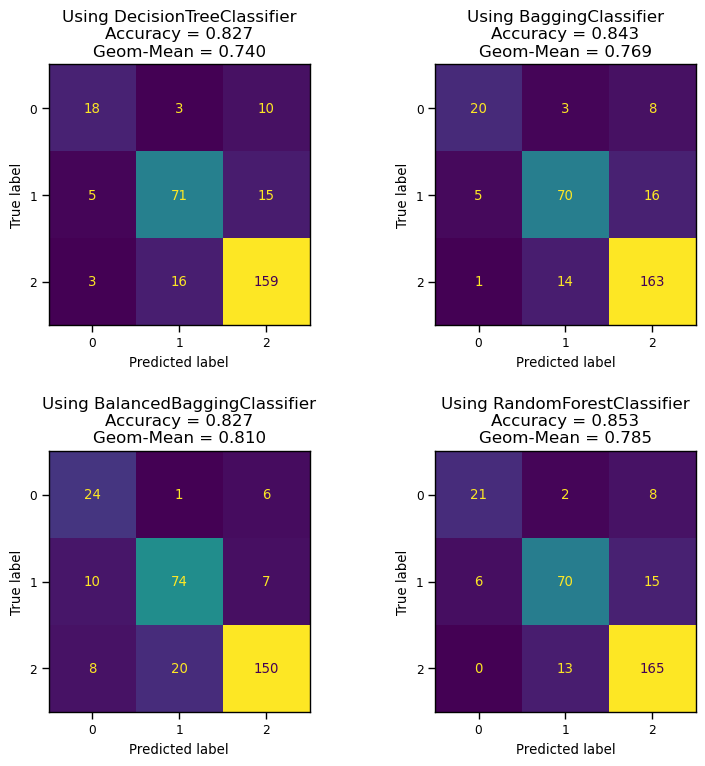
11.3.5 앙상블 방법 적용
import numpy as np
from sklearn.metrics import ConfusionMatrixDisplay
from sklearn.model_selection import train_test_split
from sklearn.tree import DecisionTreeClassifier
from sklearn.ensemble import BaggingClassifier, RandomForestClassifier
from imblearn.ensemble import BalancedBaggingClassifier, BalancedRandomForestClassifier
from imblearn.under_sampling import RandomUnderSampler, TomekLinks, EditedNearestNeighbours, NearMiss, ClusterCentroids
from imblearn.over_sampling import RandomOverSampler, SMOTE, ADASYN, BorderlineSMOTE, SVMSMOTE, KMeansSMOTE
from imblearn.combine import SMOTETomek, SMOTEENN
from imblearn.metrics import geometric_mean_score
# 시드 설정
np.random.seed(0)
# 데이터 생성
X, y = create_dataset(n_samples=1000, weights=(0.1, 0.3, 0.6))
# 데이터셋 분리
X_train, X_test, y_train, y_test = train_test_split(X, y, stratify=y, test_size=0.3, random_state=0)
# 분류기 생성
clfs = [DecisionTreeClassifier(),
BaggingClassifier(n_estimators=50),
BalancedBaggingClassifier(n_estimators=50),
RandomForestClassifier(n_estimators=50)
]
# 서브 그래프 설정
fig, axs = plt.subplots(nrows=2, ncols=2, figsize=(8, 8))
for i, clf in enumerate(clfs):
clf.fit(X_train, y_train)
accuracy = clf.score(X_test, y_test)
geom_mean = geometric_mean_score(y_test, clf.predict(X_test))
disp = ConfusionMatrixDisplay.from_estimator(clf, X_test, y_test, ax=axs[i//2,i%2], colorbar=False)
disp.ax_.set_title(f"Using {clf.__class__.__name__}\nAccuracy = {accuracy:.3f}\nGeom-Mean = {geom_mean:.3f}", fontsize=12)
fig.tight_layout(pad=2.0)
plt.show()