결측치(Missing Value)
- 데이터가 수집되지 않거나 누락되어 정보(값)가 존재하지 않음을 의미
결측치가 포함된 데이터는 모델 학습이 불가능하므로 사전에 반드시 결측치 처리를 진행
NULLNANAN
결측치 발생 원인
- 대부분 수집 및 관리 과정에서 결측치 발생
- 미수집 : 미 입력된 데이터를 수집 및 저장
- 시스템 오류 : 오류에 의해 누락되어 수집 및 저장
- 신규 항목 : 새롭게 수집 및 저장하는 항목이 추가됨
결측치 처리 방안
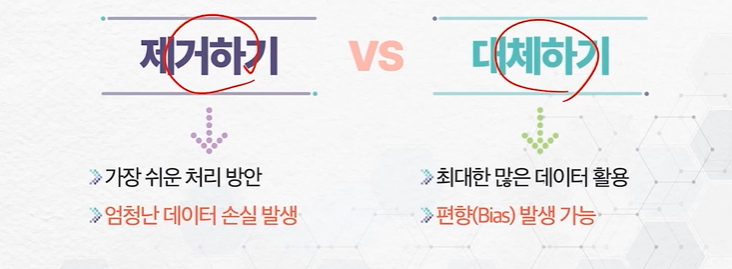
- 제거하기
- 제거 시 정보의 손실이 발생 할 수 있음
| Listwise deletion | Pairwise deletion |
|---|---|
| 결측치가 존재하는 행 삭제 | 모든 변수가 결측치로만 존재하는 행 삭제 |
| df.dropna() | df.dropna(how='all') |
- 대체하기
- 정보의 손실을 방지하나 변수 특성(평균, 상관관계 등)에 영향 발생
| 일정 값 대체 | 선형 값 대체 |
|---|---|
| 결측치를 각 변수의 평균값으로 대체 | 선형 함수 기반 앞뒤 관측치 활용 대체 |
| df['col1'].replace(np.nan, df['col'].mean()) | df.interpolate() |
결측 데이터 처리
데이터 전처리 과정 내 주의 사항
1. 데이터 전처리 과정 진행 시에는, 원본 데이터 Copy 필수
2. Python은 Copy 함수를 사용하지 않으면, 원본 데이터 값을 변경 시킴
3. 만일 전처리 내역이 변경되는 경우, 데이터 로딩부터 모든 과정을 다시 시작해야함
4. 따라서 원본 데이터와 전처리 과정을 진행할 데이터를 구분하여 작업 수행import numpy as np
import pandas as pd
#데이터 로딩 및 개요 확인
cancer = pd.read_csv("./data/wdbc.data", header=None )
cancer# 데이터 컬럼명 지정
cancer.columns = [" " ~~ ]
#id를 index화
cancer = cancer.set_index('id')
cancer
#데이터 복사
cancer_data = cancer.copy()
#데이터 내 결측치 생성
cancer_data = cancer_data[0:30]
#결측치 생성
cancer_data.iloc[2,:] = np.nan #3행 내 모든 데이터 결측치 생성
cancer_data.iloc[10,[3,4]] = np.nan # 11행 내 4,5열 데이터 결측치 생성
cancer_data.iloc[24,4] = np.nan #25행 내 5열 데이터 결측치 생성
결측치 제거하기
결측치 제거 방안
1. listwise deletion: 데이터 내 1개 변수 값에서 N/A(결측)이 존재하는 경우, 제거
2. pairwise delection: 모든 변수가 N/A(결측)이 존재하는 경우, 제거
- 결측 제거시 데이터 손실 발생
listwise
#데이터 개요
cancer_data.info()
#총 30개 중 6개 record에서 결측치존재
#listwise deletion 수행
cancer_copy = cancer_data.copy()
cancer_copy = cancer_copy.dropna()
#데이터 요약 : 총 30개중 하나의 결측치라도 보유한 6개 레코드 삭제
print(cancer_copy.info())
#데이터차원확인
print(np.shape(cancer_copy))pairwise
#pairwise deletion 수행
#30개 중 1개에서 모든 변수 결측치 존재 -> 삭제
cancer_copy = cancer_data.copy()
cancer_copy = cancer_copy.dropna(how = 'all')
#데이터요약 : 총 30개중 1개 삭제
print(canner_copy.info())
#데이터차원확인
print(np.shape(cancer_copy))결측치 대체하기
결측치 대체 방안
1. 일정 값 대체 : 결측치를 사전 지정 값으로 대체
2. 선형 값 대체 : 선형 함수 기반, 앞 뒤 record 값을 활용하여 값 대체
- 대체 시 많은 데이터 사용할 수 있지만 실 데이터와의 차이가 존재함일정 값 대체
#결측치 데이터 확인
cancer_copy = cancer_data.info()
#일정 값 대체 수행
#diagnosis 컬럼 내 결측치는 C라는 범주형 값 일괄 대체
cancer_copy['d~'] = cancer_copy['d~'].fillna('C')
cancer_copy.head(10)
#수치형 컬럼인 radius_mean 컬럼 내 결측치는 65라는 수치의 일정 값으로 대체
cancer_copy['radius_mean'] = cancer_copy['radius_mean'].fillna(65)
cancer_copy.head(10)#일정 값을 지정 값이 아닌 컬럼의 평균으로 대체
#texture_mean 컬럼 내 결측치를 texture_mean 평균값으로 대체
cancer_copy['texture_mean'] = cancer_copy['texture_mean'].replace(np.nan, cancer_copy['texture_mean'].mean())
print(cancer_copy['texture_mean'].mean())
cancer_copy.head(10)선형 값 대체 (선형보간법)
#데이터의 선형관계를 기반 대체
cancer_copy = cancer_data.copy()
#선형보간법
cancer_copy = cancer_copy.interpolate()
cancer_copy.head()
#선형 값 대체 확인
print((cancer_data.iloc[1,1] + cancer_data.iloc[3,1])/2)
