화이트 레드 와인 구별
화이트 와인과 레드 와인의 데이터를 학습시켜 와인 분류기를 만들어보자!
데이터 분석
와인 데이터에는 다음과 같이 다양한 컬럼이 존재하는데 화이트와 레드의 데이터가 따로따로 존재하므로 색상 컬럼을 추가하고 concat으로 합쳐줍니다. 이후 데이터를 확인해보면
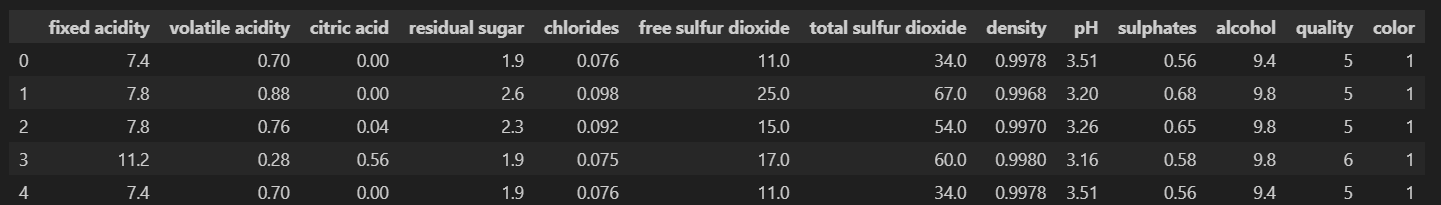
Fixed Acidity (고정 산도), Volatile Acidity (휘발성 산도), Citric Acid (구연산),
Residual Sugar (잔류 설탕), Chlorides (염화물), Free Sulfur Dioxide (자유 이산화황),
Total Sulfur Dioxide (총 이산화황), Density (밀도), pH, Sulphates (황산염),
Alcohol (알코올 도수), Quality (품질), Color (색상)
출처 전지전능하신 ChatGPT
다음과 같다.
우선 퀄리티를 기준으로 시각화 해보면 다음과 같이 5,6 등급의 데이터가 대부분임을 알수있다.
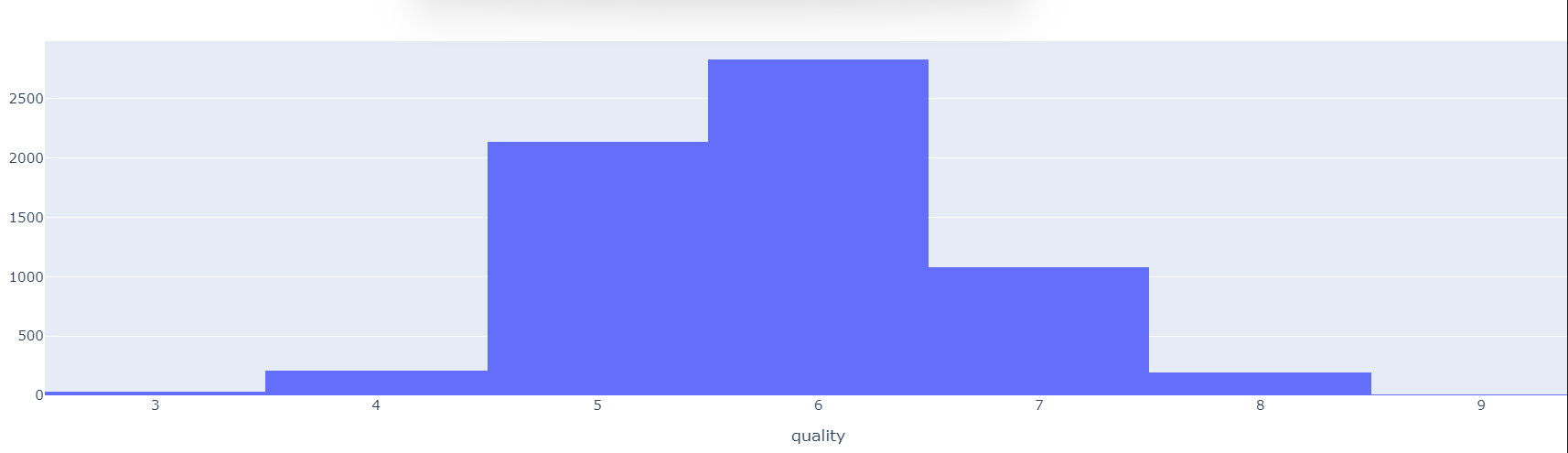
3 30
4 216
5 2138
6 2836
7 1079
8 193
9 5
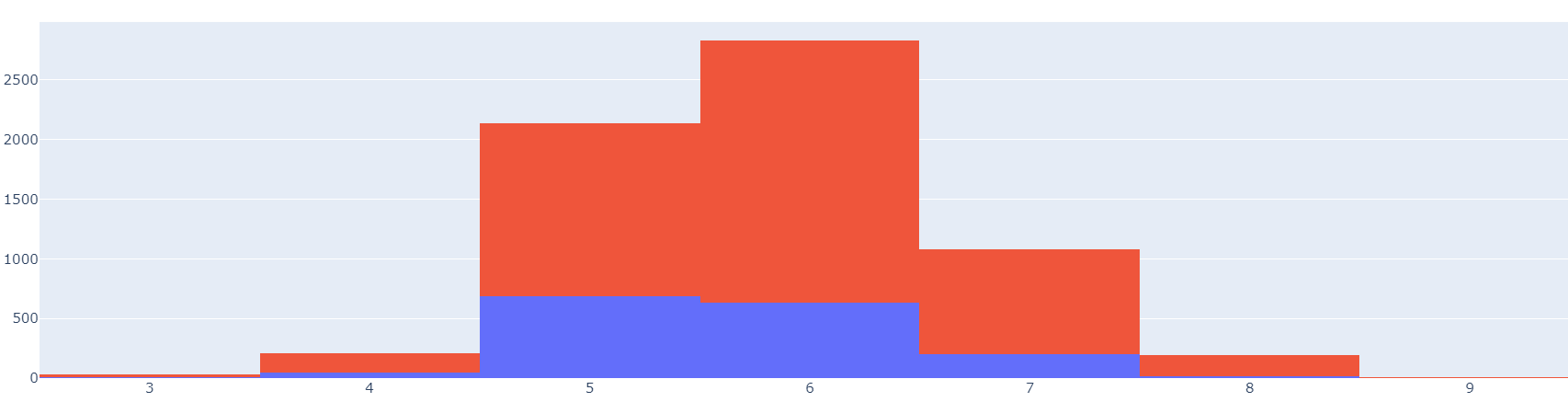
색상과 퀄리티를 기준으로 나타내면 다음과 같다.(빨강색 : 화이트 파랑색 : 레드)
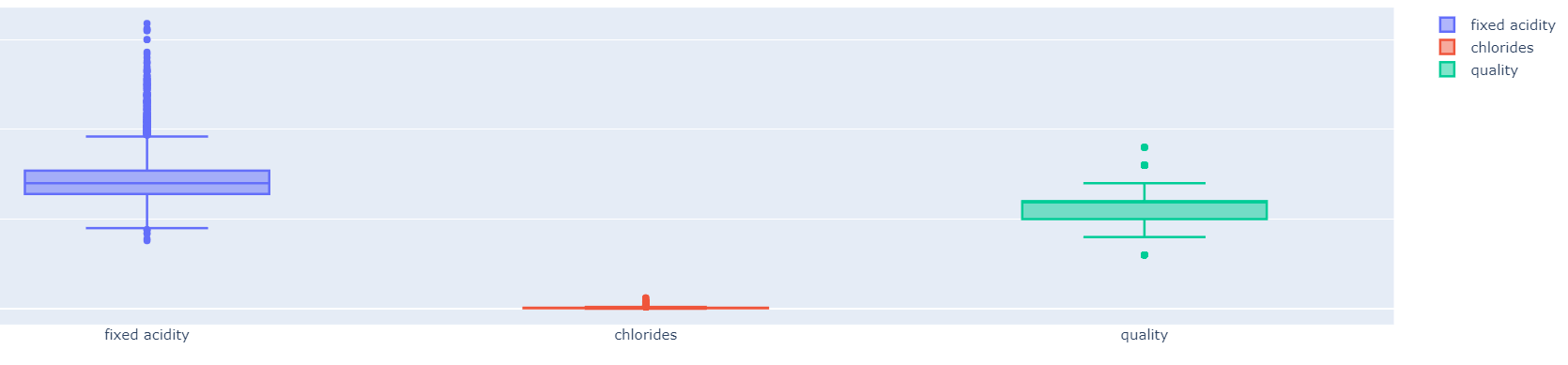
Boxplot으로 산도, Chlorides, 퀄리티를 기준으로 나타내면 다음과 같다.
fig = go.Figure()
fig.add_trace(go.Box(y=x_SS_pd['fixed acidity'], name='fixed acidity'))
fig.add_trace(go.Box(y=x_SS_pd['chlorides'], name='chlorides'))
fig.add_trace(go.Box(y=x_SS_pd['quality'], name='quality'))
fig.show()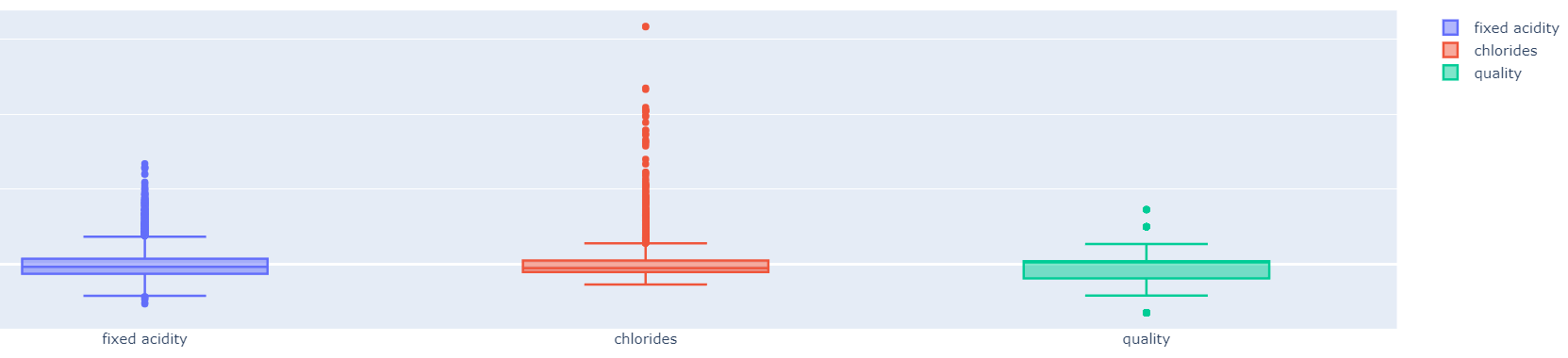
fig = go.Figure()
fig.add_trace(go.Box(y=x_MM_pd['fixed acidity'], name='fixed acidity'))
fig.add_trace(go.Box(y=x_MM_pd['chlorides'], name='chlorides'))
fig.add_trace(go.Box(y=x_MM_pd['quality'], name='quality'))
fig.show()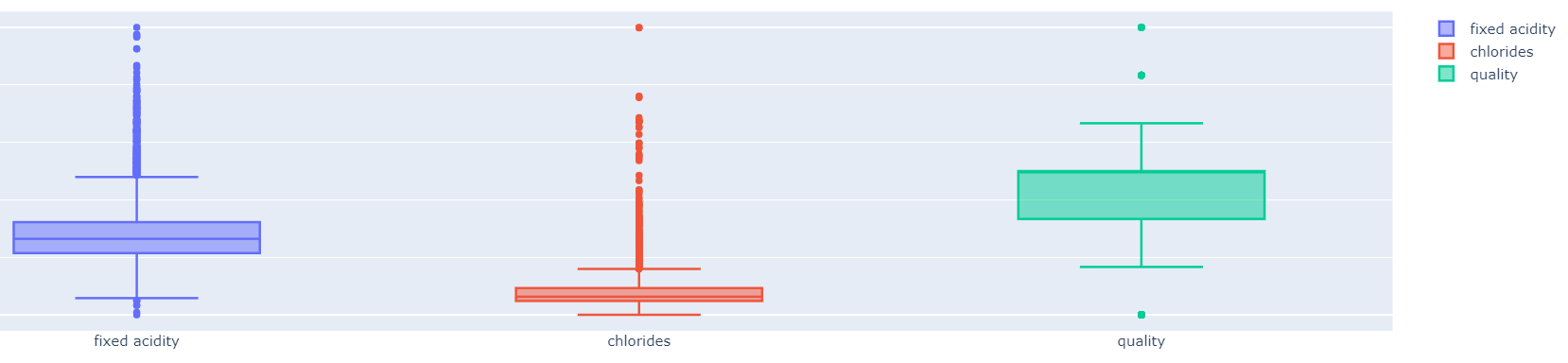
와인 감별 머신
import numpy as np
from sklearn.model_selection import train_test_split
from sklearn.tree import DecisionTreeClassifier
from sklearn.metrics import accuracy_score
x_train, x_test, y_train, y_test = train_test_split(x,y, test_size=0.2, random_state=13)
wine_tree = DecisionTreeClassifier(max_depth=2, random_state=13)
wine_tree.fit(x_train, y_train)
y_pred_tr = wine_tree.predict(x_train)
y_pred_test = wine_tree.predict(x_test)
accuracy_score(y_train, y_pred_tr)
accuracy_score(y_test, y_pred_test)Train Acc: 0.9553588608812776
Test Acc: 0.9569230769230769
지금까지 대로 사용했던 DecisionTreeClassifier 모델을 와인 감별기 모델을 만들어 보았습니다.
다음은 스케일링을 통해 데이터값을 변환 시키고 나서의 정확도를 측정 해보겠습니다.
from sklearn.preprocessing import MinMaxScaler, StandardScaler
MM = MinMaxScaler()
SS = StandardScaler()
SS.fit(x)
MM.fit(x)
x_SS = SS.transform(x)
x_MM = MM.transform(x)
x_SS_pd = pd.DataFrame(x_SS, columns=x.columns)
x_MM_pd = pd.DataFrame(x_MM, columns=x.columns)
x_train, x_test, y_train, y_test = train_test_split(x_MM_pd, y, test_size=0.2, random_state=13)
wine_tree = DecisionTreeClassifier(max_depth=2, random_state=13)
wine_tree.fit(x_train, y_train)
accuracy_score(y_train, y_pred_tr)
accuracy_score(y_test, y_pred_test)
print('Train Acc: ', accuracy_score(y_train, y_pred_tr))
print('Test Acc: ', accuracy_score(y_test, y_pred_test))Train Acc: 0.9553588608812776
Test Acc: 0.9569230769230769
그런데 결정트리 모델은 스케일링에 영향을 거의 받지 않아 점수가 똑같다는 사실을 알수있다.
영향이 없는 이유
결정 트리(Decision Tree) 모델은 특성의 스케일에 영향을 거의 받지 않습니다. 여기에는 몇 가지 이유가 있습니다:
스케일 불변성:
결정 트리는 각 노드에서 특성 값을 기준으로 데이터를 분할하는데, 이 분할은 스케일에 영향을 받지 않습니다. 즉, 특성의 스케일이 달라져도 트리의 구조는 변경되지 않습니다.
비선형 분할:
결정 트리는 각 노드에서 비선형 분할을 수행합니다. 이는 특성의 값이 어떻게 변하는지에 대한 정보를 사용하므로, 특성의 절대 크기나 범위가 중요하지 않습니다.
임계값 사용:
결정 트리에서는 특성을 기반으로 한 임계값(threshold)을 사용하여 데이터를 분할합니다. 이 임계값은 스케일에 따라 달라지지 않습니다. 따라서 특성의 스케일이 달라져도 결정 트리의 예측은 변하지 않습니다.
분할 규칙만 고려:
결정 트리는 각 분기에서 현재 노드의 특성 값만을 고려하여 데이터를 분할합니다. 이는 해당 특성의 스케일이나 다른 특성과의 관계를 고려하지 않는다는 것을 의미합니다.
스케일에 민감한 모델들, 예를 들면 선형 모델이나 거리 기반 모델(예: k-최근접 이웃) 등은 특성의 스케일에 영향을 크게 받을 수 있습니다. 하지만 결정 트리는 이러한 문제에서 자유롭습니다. 따라서 결정 트리는 스케일링이 필요 없거나 스케일링을 무시할 수 있는 경우가 많습니다.
출처 전지전능하신 CHATGPT
와인 퀄리티 감별기
이번에는 위에서 시각화한 퀄리티를 기준으로 퀄리티의 높낮이 감별기를 만들어보겠습니다.
wine['taste'] = [1 if grade > 5 else 0 for grade in wine['quality']]5이하는 0 6이상은 1로 새로운 컬럼을 만들어줍니다.
x = wine.drop(['taste'], axis=1)
y = wine['taste']
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2, random_state=13)
wine_tree = DecisionTreeClassifier(max_depth=2, random_state=13)
wine_tree.fit(x_train, y_train)
accuracy_score(y_train, y_pred_tr)
accuracy_score(y_test, y_pred_test)
print('Train Acc: ', accuracy_score(y_train, y_pred_tr))
print('Test Acc: ', accuracy_score(y_test, y_pred_test))Train Acc: 1.0
Test Acc: 1.0
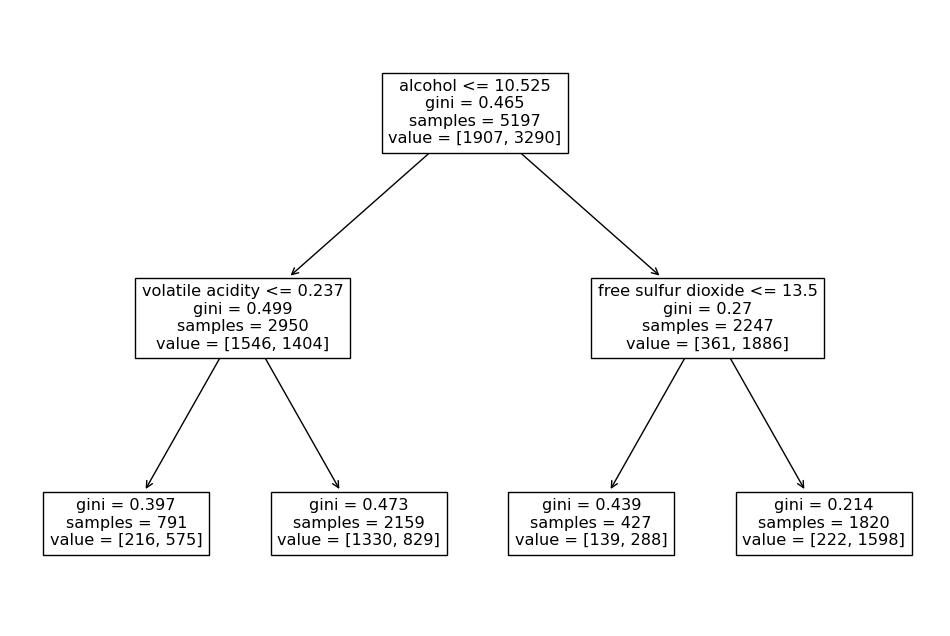
결정트리 모델의 정확도가 1.0이 나온다면, 이는 모델이 학습 데이터에 완벽하게 적합되어 과적합(overfitting)된 상태일 수 있는데 결정트리를 시각화하여 무엇이 잘못 확인 해보면
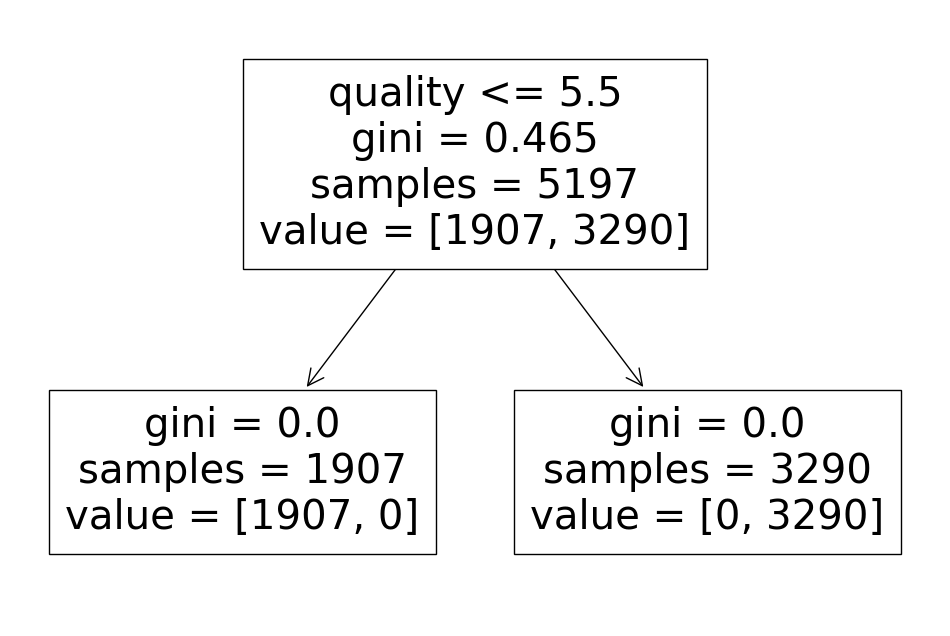
다음과 같이 퀄리티를 기준으로 taste 값을 예측했다는 것을 알 수 있습니다. 퀄리티를 기반으로 한 taste 컬럼을 생성하여 데이터를 예측했습니다. 이러한 접근 방식으로는 정확도가 1.0으로 나오기 쉽습니다. 왜냐하면 모델이 이미 주어진 퀄리티 정보를 통해 taste 값을 완벽하게 학습하였기 때문입니다.
따라서 퀄리티 컬럼까지 제거하고 다시 모델을 만들어 보겠습니다.
x = wine.drop(['taste','quality'], axis=1)
y = wine['taste']
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2, random_state=13)
wine_tree = DecisionTreeClassifier(max_depth=2, random_state=13)
wine_tree.fit(x_train, y_train)
y_pred_tr = wine_tree.predict(x_train)
y_pred_test = wine_tree.predict(x_test)
accuracy_score(y_train, y_pred_tr)
accuracy_score(y_test, y_pred_test)
print('Train Acc: ', accuracy_score(y_train, y_pred_tr))
print('Test Acc: ', accuracy_score(y_test, y_pred_test))Train Acc: 0.7294593034442948
Test Acc: 0.7161538461538461
PipeLine
전처리가 다 되어있다는 가정하에 모델은 만들었던 방법을 생각해보면
데이터를 스케일링 하고 훈련세트와 테스트세트로 분류하고 분류 모델을 적용하는 과정을 하는데 이 과정을 진행하다보면 헷갈리기도 하고 매번 하기가 귀찮아진다 이러한 문제를 해결해주는 기능이 pipeline이다.
import pandas as pd
red_wine = pd.read_csv('winequality-red.csv', sep=';')
white_wine = pd.read_csv('winequality-white.csv', sep=';')
red_wine['color'] = 1
white_wine['color'] = 0
wine = pd.concat([red_wine, white_wine])
x = wine.drop(['color'], axis=1)
y = wine['color']
앞서 지금까지하던데로 x,y로 feature와 labels값을 선언하는것까지는 똑같이하고
from sklearn.pipeline import Pipeline
from sklearn.tree import DecisionTreeClassifier
from sklearn.preprocessing import StandardScaler
estimators = [
('scaler', StandardScaler()),
('clf', DecisionTreeClassifier())
]
pipe = Pipeline(estimators)estimators에 스케일링 할 방법과 사용할 모델을 넣어두면 끝이다.
pipe.set_params(clf__max_depth=2)
pipe.set_params(clf__random_state=13)여기서 하이퍼파라메터 값을 선언해주고 싶으면 다음과 같이 해주면된다.
from sklearn.model_selection import train_test_split
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2, random_state=13, stratify=y)
pipe.fit(x_train, y_train)이제 모델에 x, y 값을 학습시키려면 위에서 만든 pipe 변수에 fit해주면
from sklearn.metrics import accuracy_score
y_pred_tr = pipe.predict(x_train)
y_pred_test = pipe.predict(x_test)
print('Train acc :', accuracy_score(y_train, y_pred_tr))
print('Test acc :', accuracy_score(y_test, y_pred_test))Train acc : 0.9657494708485664
Test acc : 0.9576923076923077
앞에서 했던것과 마찬가지의 정확도를 확인 할 수 있다.
KFold
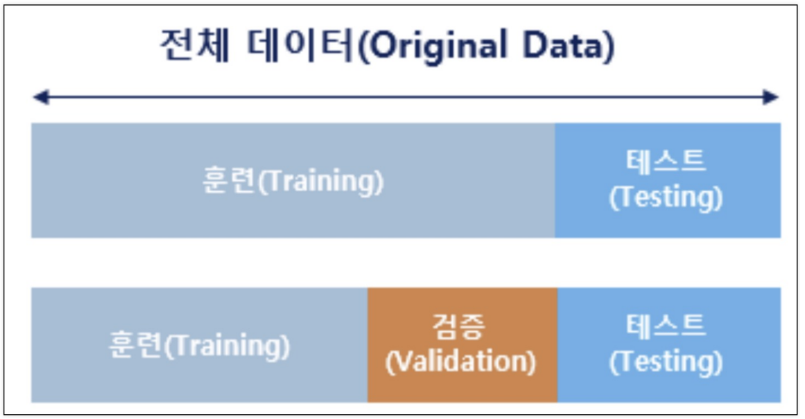
데이터세트를 분리할때 검증이라는 부분이 있었는데 이는 주어진 데이터를 훈련 세트와 테스트 세트로 나누어 모델을 평가합니다. 그러나 이렇게 나눌 경우에는 특정한 훈련 또는 테스트 세트의 구성에 따라 모델의 성능평가가 편향될 수 있는데 이런 상황에서 사용하는 기능이 KFold이다.
KFold를 사용하는 이유는 모델의 성능을 신뢰성 있게 평가하기 위해서입니다. KFold는 데이터를 여러 부분으로 나누고 각각의 부분에 대해 모델을 여러 번 훈련하고 평가하여, 다양한 조합으로 평가하므로 모델의 일반화 성능을 더 신뢰성 있게 측정할 수 있습니다. 이를 통해 모델의 안정성과 신뢰성을 높일 수 있습니다.
from sklearn.metrics import accuracy_score
from sklearn.tree import DecisionTreeClassifier
from sklearn.model_selection import train_test_split
import pandas as pd
red_wine = pd.read_csv('winequality-red.csv', sep=';')
white_wine = pd.read_csv('winequality-white.csv', sep=';')
red_wine['color'] = 1
white_wine['color'] = 0
wine = pd.concat([red_wine, white_wine])
wine['taste'] = [1. if grade > 5 else 0 for grade in wine['quality']]
x = wine.drop(['taste','quality'], axis= 1)
y = wine['taste']pipe라인과 마찬가지로 x,y로 feature와 labels값을 선언하는것까지는 똑같이하고
from sklearn.model_selection import KFold
kfold = KFold(n_splits=5)
wine_tree_cv = DecisionTreeClassifier(max_depth=2, random_state=13)n_splits 파라메터에 5를 넣어 5개의 조각으로 데이터세트를 분리하고
for train_idx, test_idx in kfold.split(x):
print(len(train_idx),len(test_idx))데이터 분리 검증
5197 1300
5197 1300
5198 1299
5198 1299
5198 1299
분리의 예시!! 전체데이터 12345
2345 1
1345 2
1245 3
1235 4
1234 5
6497개의 데이터가 4:1비율로 나눠진 것을 확인 할 수 있다.
cv_accuracy = []
for train_idx, test_idx in kfold.split(x):
x_train, x_test = x.iloc[train_idx], x.iloc[test_idx]
y_train, y_test = y.iloc[train_idx], y.iloc[test_idx]
wine_tree_cv.fit(x_train, y_train)
pred = wine_tree_cv.predict(x_test)
cv_accuracy.append(accuracy_score(y_test, pred))
cv_accuracy전체데이터를 4:1로 분리하고 훈련세트와 타깃데이터로 분류하고 이것을 학습한 데이터의 정확도를 cv_accuracy에 담아보았다.
[0.6007692307692307,
0.6884615384615385,
0.7090069284064665,
0.7628945342571208,
0.7867590454195535]
이 데이터의 평균을 내면 다음과 같다.
np.mean(cv_accuracy)0.709578255462782
StratifiedKFold
kfold와 같이 데이터를 분류하는 기능이긴하지만 무슨 차이인지는 몰라 전지전능하신분에게 물어보았습니다
StratifiedKFold를 사용하는 이유는 데이터의 클래스 분포를 고려하여 균등한 클래스 분포를 갖는 부분집합으로 데이터를 나누기 때문입니다. 일반 KFold와는 달리 StratifiedKFold는 각 폴드 내에서 클래스의 비율을 유지하여 훈련 및 평가를 진행합니다. 이렇게 하면 모델이 각 클래스에 대해 균형있게 학습하고 평가되므로, 특히 클래스 불균형이 심한 경우에 모델의 성능을 신뢰성 있게 평가할 수 있습니다. 따라서 StratifiedKFold는 데이터셋의 클래스 불균형을 고려하여 모델 평가를 수행하는 데 도움이 됩니다.
from sklearn.model_selection import StratifiedKFold
skFold = StratifiedKFold(n_splits=5)
wine_tree_cv = DecisionTreeClassifier(max_depth=2, random_state=13)
cv_accuracy = []
for train_idx, test_idx in skFold.split(x,y):
x_train, x_test = x.iloc[train_idx], x.iloc[test_idx]
y_train, y_test = y.iloc[train_idx], y.iloc[test_idx]
wine_tree_cv.fit(x_train, y_train)
pred = wine_tree_cv.predict(x_test)
cv_accuracy.append(accuracy_score(y_test, pred))
cv_accuracy0.5523076923076923,
0.6884615384615385,
0.7143956889915319,
0.7321016166281755,
0.7567359507313318
마찬가지로 평균을 내면 다음과 같다.
np.mean(cv_accuracy)0.6888004974240539
cross_val_score
지금까지는 원리를 배우고자 세부적으로 코드를 뜯어보았는데 cross_val_score 기능을 몇줄 안되는 코드로 사용할수있다.
from sklearn.model_selection import cross_val_score
skfold = StratifiedKFold(n_splits=5)
wine_tree_cv = DecisionTreeClassifier(max_depth=2, random_state=13)
cross_val_score(wine_tree_cv, x, y, scoring=None, cv=skfold)array([0.50076923, 0.62615385, 0.69745958, 0.7582756 , 0.74903772])
from sklearn.model_selection import cross_val_score
skfold = StratifiedKFold(n_splits=5)
wine_tree_cv = DecisionTreeClassifier(max_depth=5, random_state=13)
cross_val_score(wine_tree_cv, x, y, scoring=None, cv=skfold)[0.56846154 0.68846154 0.71439569 0.73210162 0.75673595]
GridSearchCV
이제부터 다양한 모델들을 사용하게 될것이고 현재 배운 하이퍼파라메터도 max_depth뿐이지만 추후 파라메터가 늘어나 여러가지 경우의 파라메터를 시도하여 최적을 파라메터를 찾기위해 노가다를 해야할지도 모른다. 이런 상황을 해결해주는기능이 GridSearchCV이다. GridSearchCV는 사용자가 지정한 여러 하이퍼파라미터 조합을 대상으로 교차 검증을 수행하고, 가장 좋은 조합을 찾아줍니다. 이를 통해 모델의 성능을 최대로 끌어올리고, 일반화 능력을 향상시킬 수 있습니다. 또한, 모든 가능한 조합을 시스템적으로 탐색하기 때문에 사용자는 수동으로 하이퍼파라미터를 조정하는 시간과 노력을 절약할 수 있습니다. 결국 GridSearchCV는 모델 튜닝을 자동화하여 최적의 모델을 찾을 수 있도록 도와줍니다.
from sklearn.model_selection import GridSearchCV
params = {'max_depth': [2, 4, 7, 10]}
wine_tree = DecisionTreeClassifier(max_depth=2, random_state=13)
# cv는 k-fold와 같은 의미
gridsearch = GridSearchCV(estimator=wine_tree, param_grid=params, cv= 5)
gridsearch.fit(x, y)params에 시도해볼 파라메터 값을 넣고 GridSearchCV에 시도할 모델과 파라메터 값들을 입력해주면

다음과 같이 최적의 값이 무엇인지 나온다.
import pprint
pp = pprint.PrettyPrinter(indent=4)
pp.pprint(gridsearch.cv_results_)pprint는 출력값을 보기좋게 출력해주는 라이브러리
{ 'mean_fit_time': array([0.00622358, 0.01010599, 0.02012391, 0.03010569]),
'mean_score_time': array([0.00120115, 0.00155535, 0.00149288, 0.00179901]),
'mean_test_score': array([0.6888005 , 0.66356523, 0.65340854, 0.64401587]),
'param_max_depth': masked_array(data=[2, 4, 7, 10],
mask=[False, False, False, False],
fill_value='?',
dtype=object),
'params': [ {'max_depth': 2},
{'max_depth': 4},
{'max_depth': 7},
{'max_depth': 10}],
'rank_test_score': array([1, 2, 3, 4]),
'split0_test_score': array([0.55230769, 0.51230769, 0.50846154, 0.51615385]),
'split1_test_score': array([0.68846154, 0.63153846, 0.60307692, 0.60076923]),
'split2_test_score': array([0.71439569, 0.72363356, 0.68360277, 0.66743649]),
'split3_test_score': array([0.73210162, 0.73210162, 0.73672055, 0.71054657]),
'split4_test_score': array([0.75673595, 0.7182448 , 0.73518091, 0.72517321]),
'std_fit_time': array([0.00077733, 0.00074787, 0.00101098, 0.00088018]),
'std_score_time': array([0.00039984, 0.00065497, 0.0004526 , 0.00037116]),
'std_test_score': array([0.07179934, 0.08390453, 0.08727223, 0.07717557])}
다음과 같이 파라메터에서 시도한 결과값을 확인할수있다.
gridsearch.best_score_최적의 정확도를 출력하는 코드
0.6888004974240539
GridSearchCV과 pipeline 기능 응용
pipeline과 GridSearchCV을 동시에 사용하는 방법은 다음과 같다.
from sklearn.pipeline import Pipeline
from sklearn.tree import DecisionTreeClassifier
from sklearn.preprocessing import StandardScaler
estimators = [
('scaler', StandardScaler()),
('clf', DecisionTreeClassifier())
]
pipe = Pipeline(estimators)
param_grid = [ {'clf__max_depth': [2, 4, 7, 10]} ]
GridSearch = GridSearchCV(estimator=pipe, param_grid=param_grid, cv=5)
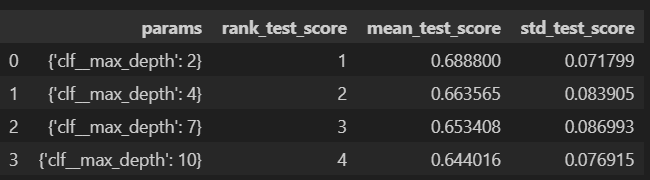
GridSearch.fit(x, y)고오급 기술
score_df = pd.DataFrame(GridSearch.cv_results_)
score_df
score_df[['params', 'rank_test_score', 'mean_test_score', 'std_test_score']]GridSearch 출력의 최적의 값을 데이터프레임화 하고 필요한 데이터를 확인하는 방법