주성분분석(PCA) 차원 축소 알고리즘
우리는 데이터를 분석하고자 할 때 고려하고자 하는 상황이 매우 많을 수 있습니다. 그런 상황에 어떤 요소를 줄여 내가 얻고자하는 것을 명확히 하게 도와줍니다.
- 수학적으로 말하면 큰 방향벡터가 있는데 그 데이터들의 분산을 보존하면서 직교와 기저를 찾아 고차원을 저차원으로 사영하는 것입니다.
- PCA에서는 기존 feature 중 중요한 것을 선택하는 방식이 아닌 기존의 feature를 선형결합 하는 방식을 사용하고 있습니다.
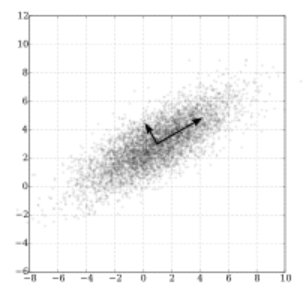
위 그림과 같이 타원형 분포로 되어있는 데이터들이 있을 때, 차원의 수는 줄이면서 데이터 분포의 분산을 최대한 유지하기 위해 가장 분산이 긴 축을 첫 기저로 잡고, 그 기저에 직교하는 축 중 가장 분산이 큰 값을 다음 기저로 잡게 됩니다. 이 과정이 반복되면 차원의 수를 최대한 줄일 수 있고 분포의 분산도 유지할 수 있습니다.
# 차원 축소 예제: 유방암 데이터셋
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
from sklearn.datasets import make_blobs
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import random
# 데이터 로드
cancer=load_breast_cancer()
# y=0(Malignant:악성 종양), y=1(Benign:양성 종양)
cancer_X, cancer_y= cancer.data, cancer['target']
train_X, test_X, train_y, test_y = train_test_split(cancer_X, cancer_y, test_size=0.1, random_state=10) # train 데이터셋과 test 데이터셋으로 나눔
print("전체 검사자 수: {}".format(len(cancer_X)))
print("Train dataset에 사용되는 검사자 수: {}".format(len(train_X)))
print("Test dataset에 사용되는 검사자 수: {}".format(len(test_X)))
cancer_df = pd.DataFrame(cancer_X, columns=cancer['feature_names'])
cancer_df.head()전체 검사자 수: 569
Train dataset에 사용되는 검사자 수: 512
Test dataset에 사용되는 검사자 수: 57
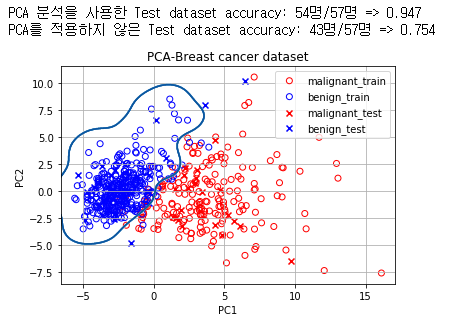
유방암 데이터를 이용해서 주성분분석을 진행할 것이고 train데이터에 적용한 주성분분석 즉 포함시키는 변수들을 test에 똑같이 적용을 시킬 것입니다. 서포트 벡터머신을 통해서 유방암 데이터에서 음성인지 양성인지 경계선을 구분지어주고 확인해 보겠습니다.
from sklearn.preprocessing import StandardScaler
from sklearn.decomposition import PCA
from sklearn import svm
from sklearn.metrics import accuracy_score
from collections import Counter
# color dictionary
color_dict = {0: 'red', 1: 'blue', 2:'red', 3:'blue'}
target_dict = {0: 'malignant_train', 1: 'benign_train', 2: 'malignant_test', 3:'benign_test'}
#Train data에 PCA 알고리즘 적용
train_X_ = StandardScaler().fit_transform(train_X) # 불러온 데이터에 대한 정규화 -> 각 column의 range of value가 전부 다르기 때문에 정규화를 진행해 주어야 합니다.
train_df = pd.DataFrame(train_X_, columns=cancer['feature_names'])
pca = PCA(n_components=2) # 주성분의 수를 2개, 즉 기저가 되는 방향벡터를 2개로 하는 PCA 알고리즘 수행
pc = pca.fit_transform(train_df)#Test data에 PCA 알고리즘 적용
test_X_ = StandardScaler().fit_transform(test_X) # normalization
test_df = pd.DataFrame(test_X_, columns=cancer['feature_names'])
pca_test = PCA(n_components=2)
pc_test = pca_test.fit_transform(test_df)# 훈련한 classifier의 decision boundary를 그리는 함수
def plot_decision_boundary(X, clf, ax):
h = .02 # step size in the mesh
# create a mesh to plot in
x_min, x_max = X[:, 0].min() - 1, X[:, 0].max() + 1
y_min, y_max = X[:, 1].min() - 1, X[:, 1].max() + 1
xx, yy = np.meshgrid(np.arange(x_min, x_max, h), np.arange(y_min, y_max, h))
Z = clf.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
ax.contour(xx, yy, Z, cmap='Blues')
# PCA를 적용한 train data의 classifier 훈련: classfier로 Support Vector Machine(SVM) 사용
clf = svm.SVC(kernel = 'rbf', gamma=0.5, C=0.8) # 여기서는 classifier로 SVM을 사용한다는 정도만 알아둡시다!
clf.fit(pc, train_y) # train data로 classifier 훈련
# PCA를 적용하지 않은 original data의 SVM 훈련
clf_orig = svm.SVC(kernel = 'rbf', gamma=0.5, C=0.8) # 여기서는 classifier로 SVM을 사용한다는 정도만 알아둡시다!
clf_orig.fit(train_df, train_y)SVC(C=0.8, gamma=0.5)
# 캔버스 도식
fig = plt.figure()
ax = fig.add_subplot(1, 1, 1)
# malignant와 benign의 SVM decision boundary 그리기
plot_decision_boundary(pc, clf, ax)
#Train data 도식
for cluster in range(2):
sub_cancer_points = pc[train_y == cluster]
ax.scatter(sub_cancer_points[:, 0], sub_cancer_points[:, 1], edgecolor=color_dict[cluster], c='none', label=target_dict[cluster])
#Test data 도식
for cluster in range(2):
sub_cancer_points = pc_test[test_y == cluster]
ax.scatter(sub_cancer_points[:, 0], sub_cancer_points[:, 1], marker= 'x', c=color_dict[cluster+2], label=target_dict[cluster+2])
ax.set_xlabel('PC1')
ax.set_ylabel('PC2')
ax.set_title('PCA-Breast cancer dataset')
ax.legend()
ax.grid()
# Scoring
pca_test_accuracy_dict = Counter(clf.predict(pc_test) == test_y)
orig_test_accuracy_dict = Counter(clf_orig.predict(test_df) == test_y)
print("PCA 분석을 사용한 Test dataset accuracy: {}명/{}명 => {:.3f}".format(pca_test_accuracy_dict[True], sum(pca_test_accuracy_dict.values()), clf.score(pc_test, test_y)))
print("PCA를 적용하지 않은 Test dataset accuracy: {}명/{}명 => {:.3f}".format(orig_test_accuracy_dict[True], sum(orig_test_accuracy_dict.values()), clf_orig.score(test_df, test_y)))