의사결정트리(Decision Tree), 랜덤포레스트 분류 실습
📍 Python 라이브러리
%matplotlib inline # notebook 환경에서 바로 볼 수 있도록 해줌
import matplotlib.pyplot as plt # 시각화
import numpy as np
from sklearn.model_selection import train_test_split
from sklearn.datasets import load_breast_cancer
from sklearn.tree import DecisionTreeClassifier
from sklearn import tree
from sklearn.ensemble import RandomForestClassifier
* 의사결정트리
DecisionTreeClassifier(sklearn DT 페이지 참고) :criterion: default='gini', 불순도 측정 종류 (gini,entropy,log_loss).plot_tree: 트리모델 시각화 plot_tree(doc)
* 랜덤포레스트
RandomForestClassifier(sklearn RF 페이지 참고) :criterion: default='gini', 불순도 측정 종류 (gini,entropy,log_loss)n_estimatorsint: default=100, forest안의 tree개수
* 공통
load_breast_cancer: sklearn 유방암 데이터 사용 (load_breast_cancer)train_test_split:train_size: default=0.25, train 데이터 셋의 비율test_size: default=0.25, test(validation) 데이터 셋의 비율shuffle: default=True, split해주기 전에 섞을건지 여부. 보통 default값으로 놔둠stratify*: default=None, stratify 값을 target으로 지정하면, 각각의 class 비율을 train / test(validation)에 유지하여, 한 쪽에 쏠려서 분배되는 것을 방지함.
ex) 목적변수 Y가 25%의 0과 75%의 1로 이루어진 Binary Set일 때, stratify=Y로 설정하면 나누어진 데이터셋들도 0과 1을 각각 25%, 75%로 유지한 채 분할됨.random_state: 세트를 섞을 때, 해당 int값을 보고 섞으며, 매번 데이터셋이 변경되는 것을 방지할 수 있다.
🌲 의사결정트리(DT) 실습
- 전체코드
def Decision_Tree_Classifier(dataset, max_depth, test_size):
""" train - test(validation) set 분리 """
X_train,X_test,y_train,y_test = train_test_split(dataset.data,dataset.target,stratify=dataset.target, test_size=test_size, random_state=60)
""" 일정 깊이에 도달하면 멈추는 트리 모델 생성 및 학습"""
dt_clf = DecisionTreeClassifier(max_depth=max_depth, random_state=0)
dt_clf.fit(X_train, y_train)
""" train, test 정확도 확인 """
train_accuracy = dt_clf.score(X_train,y_train)
test_accuracy = dt_clf.score(X_test,y_test)
print("train_accuracy:{:.3f}".format(train_accuracy))
print("test_accuracy:{:.3f}".format(test_accuracy))
""" 트리 시각화 """
plt.figure( figsize=(20,15) )
tree.plot_tree(dt_clf,
class_names=cancer.target_names,
feature_names=cancer.feature_names,
impurity=True, filled=True,
rounded=True)
plt.savefig('DT_python.png', #파일이름
facecolor='#eeeeee', #여백색
edgecolor='black', #테두리색
format='png', #파일형식
dpi=100) #해상도(default:100)
plt.show()
""" 트리 중요도 feature_importances """
features = cancer.feature_names
importances = dt_clf.feature_importances_
importance_dict = {name:value for name, value in zip(features, importances)}
print("Feature Importances: \n", importance_dict)
plt.figure(figsize=(15,5))
n_features = cancer.data.shape[1]
plt.barh(np.arange(n_features), importances, align="center")
plt.yticks(np.arange(n_features), features)
plt.xlabel("feature importance")
plt.ylabel("feature")
plt.ylim(-1, n_features)
plt.savefig('DT_feature_python.png', #파일이름
facecolor='#eeeeee', #여백색
edgecolor='black', #테두리색
format='png', #파일형식
dpi=100) #해상도(default:100)
plt.show()
""" 유방암 데이터구조 살펴보기 """
cancer = load_breast_cancer()
# print(cancer.data) # X
# print(cancer.feature_names)
# print(cancer.target) # label(Y)
# print(cancer.DESCR)
""" 의사결정트리 함수 실행 """
Decision_Tree_Classifier(cancer, 4, 0.25)
- 결과
- train_accuracy:0.986 test_accuracy:0.937
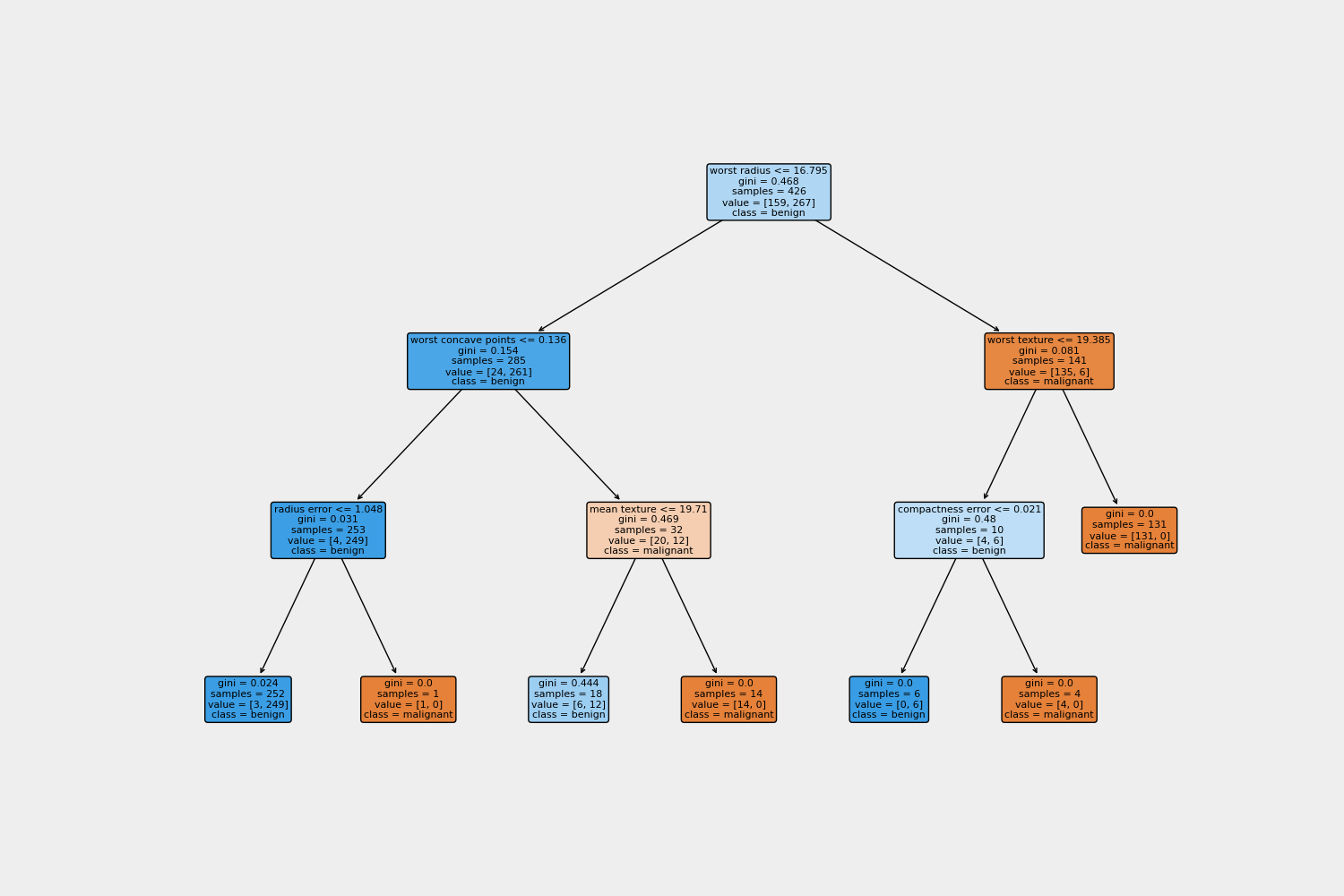
- 트리 시각화 (class - malignant:악성 , benign:양성)
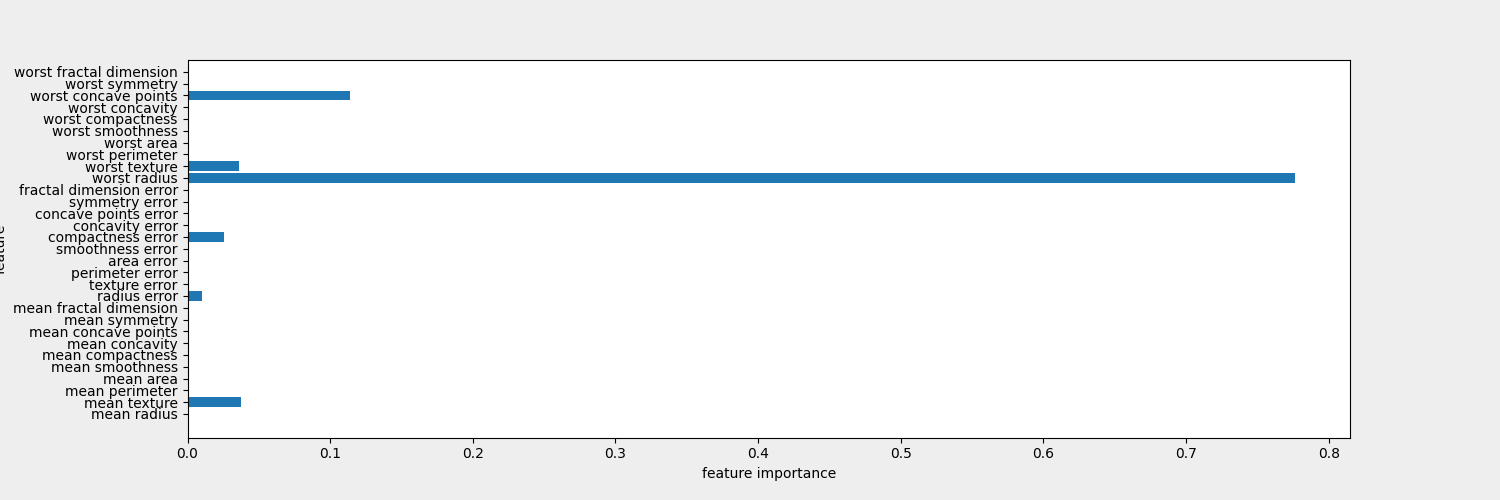
- 트리의 특성 중요도(feature_importance) (0: 전혀사용되지 않음 ~ 1:완벽하게 타겟 클래스 예측)
- 트리 분류 모델 형성에 각 특성이 얼마나 작용했는지 평가하는 지표
🌲🌲랜덤포레스트 실습
- 전체코드
def Random_Forest_Classifier(dataset, max_depth, test_size):
""" train - test(validation) set 분리 """
X_train,X_test,y_train,y_test = train_test_split(dataset.data,dataset.target,stratify=dataset.target, test_size=test_size, random_state=60)
""" 랜덤포레스트 모델 생성 및 학습"""
rf_clf = RandomForestClassifier(n_estimators=100, max_depth=max_depth, random_state = 0)
rf_clf.fit(X_train, y_train)
""" train, test 정확도 확인 """
train_accuracy = rf_clf.score(X_train,y_train)
test_accuracy = rf_clf.score(X_test,y_test)
print("train_accuracy:{:.3f}".format(train_accuracy))
print("test_accuracy:{:.3f}".format(test_accuracy))
""" 랜덤포레스트 중요도 feature_importances """
features = cancer.feature_names
importances = rf_clf.feature_importances_
importance_dict = {name:value for name, value in zip(features, importances)}
print("Feature Importances: \n", importance_dict)
plt.figure(figsize=(15,5))
n_features = cancer.data.shape[1]
plt.barh(np.arange(n_features), importances, align="center")
plt.yticks(np.arange(n_features), features)
plt.xlabel("feature importance")
plt.ylabel("feature")
plt.ylim(-1, n_features)
plt.savefig('RF_python.png', #파일이름
facecolor='#eeeeee', #여백색
edgecolor='black', #테두리색
format='png', #파일형식
dpi=100) #해상도(default:100)
plt.show()
""" 유방암 데이터구조 살펴보기 """
cancer = load_breast_cancer()
# print(cancer.data) # X
# print(cancer.feature_names)
# print(cancer.target) # label(Y)
# print(cancer.DESCR)
""" 랜덤포레스트 함수 실행 """
Random_Forest_Classifier(cancer, 4, 0.25)- 결과
- train_accuracy:0.988 test_accuracy:0.951
- 트리의 특성 중요도(feature_importance)
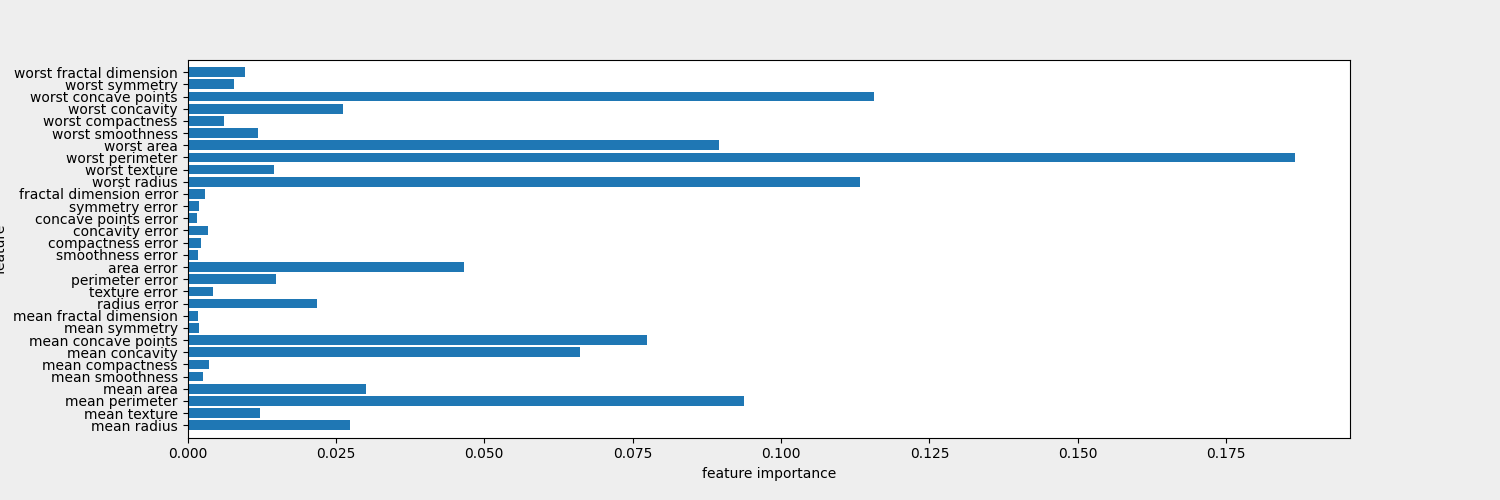
- 의사결정트리의 과적합 완화