[Paper Review] RetCCL: Clustering-guided contrastive learning for whole-slide image retrieval
Paper Review
RetCCL: Clustering-guided contrastive learning for whole-slide image retrieval
디지털 병리 AI 심포지움에서 reference 되었던 breast npj 2023 논문에서 차용한 SSL 방법이다. WSI 에서 SSL 방식을 도입할 때 발생할 수 있는 문제점을 인지하고 이를 해결하기 위해 fancy 한 방법을 사용했다. 결과 좋은 성능을 내었고 많은 논문에서 인용되고 있어 의미 있는 논문이라 생각한다.
Introduction
- WSI annotation 및 computation cost 높음.
- SSL 방식을 사용하면 이 문제를 alleviate 할 수 있음.
- 기존 SimCLR 등의 방식에서 patch 를 instance 로 여겨 학습하면, serious bias 생김.
- unbalanced tissue type distribution 와 large portion of similar tissues
- 이를 위해 다음 두가지 방법을 사용한 framework 를 제안함.
1) clustering-guided contrastive learning (CCL) for feature extraction
2) distinctive query patch selection, ranking for searched pathes, and aggregation algorithm for interpretable WSI searching
Related works
Self-supervised representation learning
- natural image application to the histopathology domain : domain shift
- focus on semantic features within the specific task : limited model generalization
- not tested on large and diverse histopathological image datasets
Histopathological image retrieval
- traditional hand-craft features
- DL method based high-level features
- suboptimal performance due to the domain shift btw natural and histopathological images
Methods
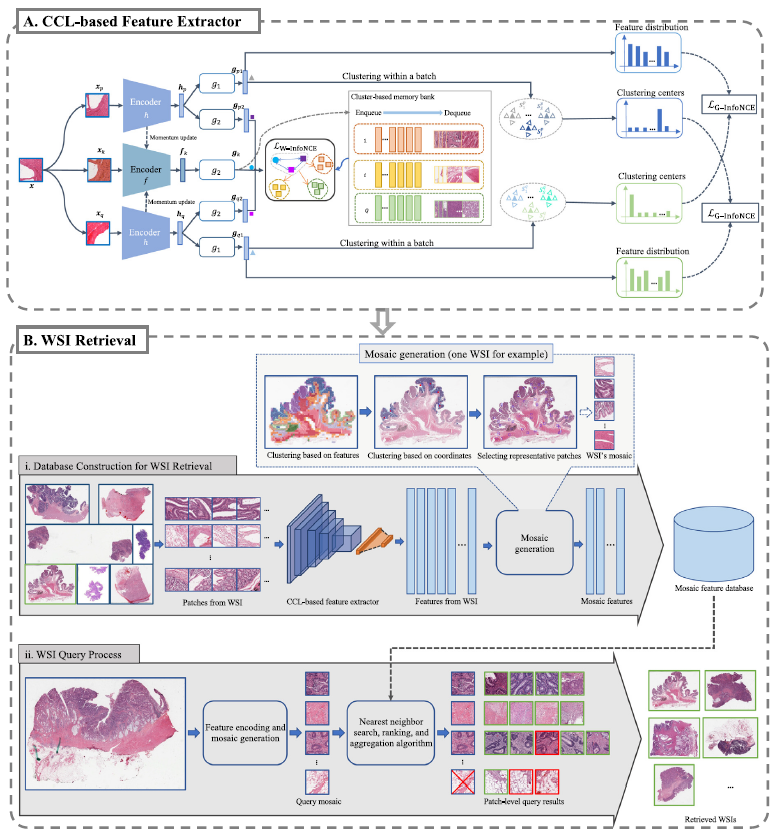
다음과 같은 구조로 되어있다.
1. weighted InfoNCE, group-level InfoNCE 활용한 feature extraction
2. 1) offline database construction for WSI retrieval
2. 2) online WSI query process
Contrastive learning based feature extractor
Preliminary of contrastive learning
x: image, x_q, x_k: augmented views 라 할 때, contrastive learning 은 q & k+ sample 을 가까이에, q & k- sample 을 멀리 두도록 학습한다. 가장 기본적인 loss 는 다음과 같이 쓸 수 있다.
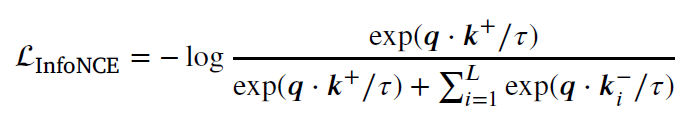
sigma L 이 포함된 묶음은 memory bank 내 negative samples 을 의미한다. 이를 통해 momentum encoder 이 moving average 방식으로 update 된다.
proposed clustering-guided contrastive learning
위와 같은 방식을 사용했을 때 다른 데이터셋에선 잘 나올지 몰라도 WSI 데이터에는 맞지 않을 수 있다. 위 식으로 봤을 때, negative sample memory bank에 positive sample 로 consider 되어야 할 highly correlated sample 이 포함되어 있을 가능성이 높다. 또, different patch 들이 같은 tissue 로부터 샘플링 되었고 strikingly similar appearance 를 가졌다면 문제는 더 심각해진다. 이는 histopathology 분야에서 SSL 방식을 도입할 때 줄곧 언급되어 왔던 한계점이기도 하다.
이 문제를 해결하기 위해, 두가지 loss (weighted InfoNCE & group-level InfoNCE) 로 구성된 clustering-based contrastive learning method 를 제안한다. W-InfoNCE loss 는 possible false-negative sample 의 영향을 줄이기 위함이고, G-InfoNCE loss 는 distinctive group center 끼리는 멀도록 하고 다른 instance 는 group 으로 모으는 역할을 한다.

먼저 three augmented images 를 얻는다. (xp, xk, xq) 그리고 두개의 encoder h (xp, xq) 와 f (xk) 를 통과시켜 feature (hp, fk, hq) 를 얻는다. 이후 two MLP heads g1, g2 를 이용해, hp 를 gp1, gp2 로 encoding 하고, hq 를 hq1, hq2 로 encoding 한다. fk 의 경우 g2 를 이용해 gk 를 얻는다. MLP head g2 를 통과시켜 나온 세 개의 feature gp2, gq2, gk 를 이용해 weighted InfoNCE loss 를 계산하게 되고, MLP head g1 을 통과시켜 나온 두 개의 feature gp1, gq1 은 cluster 하는 데에 사용되어 향후 group-level infoNCE loss 에 활용된다. 이후 final loss function 은 두 loss 의 조합으로 계산된다.
Online clustering-guided memory bank construction
weight 를 주어 false-negative-like sample 의 영향을 줄임으로써 기존 memory bank 와는 차별된 online clustering-guided memory bank 를 제안한다.
매 training epoch 마다, two positive pairs {gp2, gk}, {gq2, gk} 를 활용해, shared memory bank 로 contrastive learning 을 수행한다. 우선 negative sample 을 k-means clusering 을 활용해 Q classes 로 나누고, 이를 Q sub-memory queues 로 본다. 이 때, 각 centroid c (q개) 와 gk 간의 similarity 를 계산하고, 이 similarity 값 중 가장 큰 값을 갖는 Q 를 Q_max 라고 지정한다. 이후 negative sample 과의 similarity 를 계산할 때 다음 을 씌워서 영향력을 조절한다.
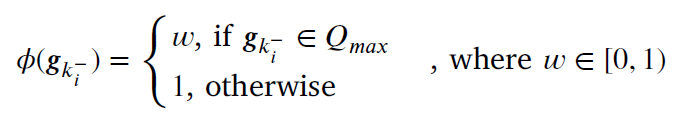
즉 가장 가까운 Q cluster 에 해당하는 negative sample, 즉 false-negative-like sample 의 영향력을 줄이는 것이다. 다음과 같은 식으로 weighted infoNCE loss 를 표현한다.
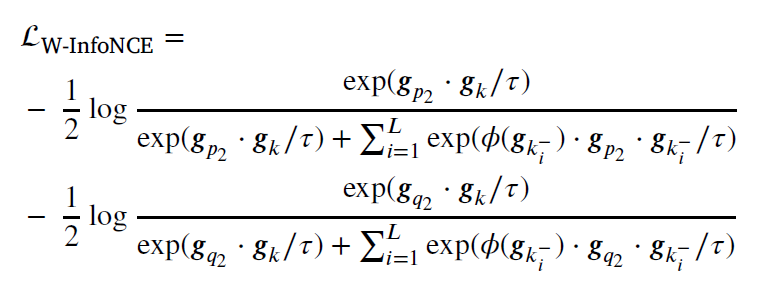
Group-level discrimination
이 식은 2021 CVPR 에 올라왔던 cross-level discrimination (CLD) loss function 으로부터 따왔다. 여기에 auxiliary branch 를 붙여 unbalanced positive/negative sample ratio 를 해소하려 하였다.

다시 위 그림에서, 이번엔 g1 MLP head 를 보자. hp 와 hq 는 다시 g1 을 거쳐 gp1, gp2 vector 를 생성한다. mini-batch 내에 있는 embedding 들은 각 두 branch 에서 S개의 cluster 로 clustering 되고, 각각의 cluster centroid 도 할당한다. 이때 요 cluster centroid 를 이용해서 다시 positive negative sample 을 branch 에서 정의하고 group-level InfoNCE loss 를 계산하게 된다. 구체적으로, gp1 (p branch이다) 이 있을 때, 이것의 positive sample 은 S개의 cluster 중 가장 가까운 1개의 cluster centroid (, q branch 임) 의 sample 이 되는 것이고, 나머지 S-1 cluster 는 negative sample 이 된다. 식은 다음과 같다.
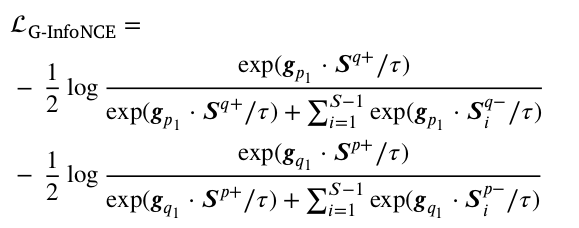
Group-level infoNCE loss 와 위에서 계산했던 weighted InfoNCE loss 의 combination 으로 final loss 를 꼐산하고 update 한다.
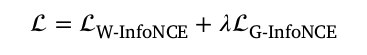
WSI retrieval method
Preliminary of WSI retrieval
WSI retrieval의 background 를 알려주고 있긴 한데 우선 나는 WSI retrieval 이 무엇인지도 잘 몰랐다. retrieval은 대략 "검색" 이라는 뜻을 가지고 있는데, 다른 reference 논문을 타고 들어가보니, 다음과 같은 작업을 가리키는 것 같았다.
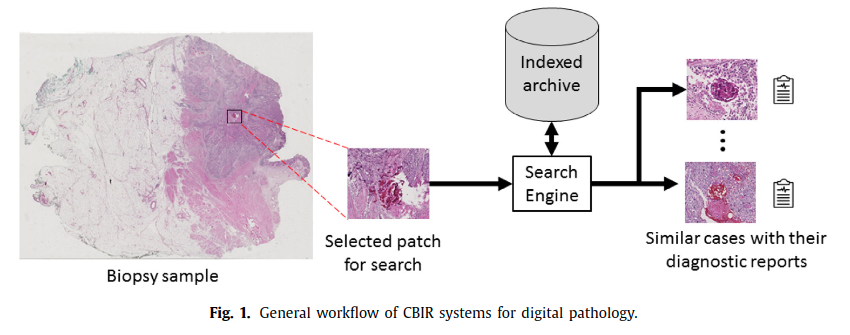
결국 feature extraction 을 잘 수행하고, similarity 계산을 잘 수행해서 WSI 내에서 빠르게 잘 찾아내는 것이 task 의 핵심이라고 생각하면 될 것 같다. 두가지 방식을 소개하고 있다.
- Yottixel method (Y)
- FISH
공통적으로 두 방식은 WSI 를 patch 로 나누고 clustering 을 한 후 query 에서 patch-by-patch matching 후 database 에서 가장 비슷한 patch 를 선정한다고 한다. 이 때 Y 는 pretrained DenseNet 으로 feature를 뽑고, 이를 binary code 로 compression 하는 반면, FISH 는 VQ-VAE pretrained on TCGA 로 generate texture feature 한다. 또한 Y 는 false match 를 줄이기 위해 Hamming distance 를 계산하고, FISH 는 Van Emde Boas tree with an uncertainty-based ranking algorithm 을 사용한다고 한다.
Proposed WSI retrieval method
overall procedure 는 다음과 같다. 크게 offline database construction, online WSI query process 두가지로 나뉜다.
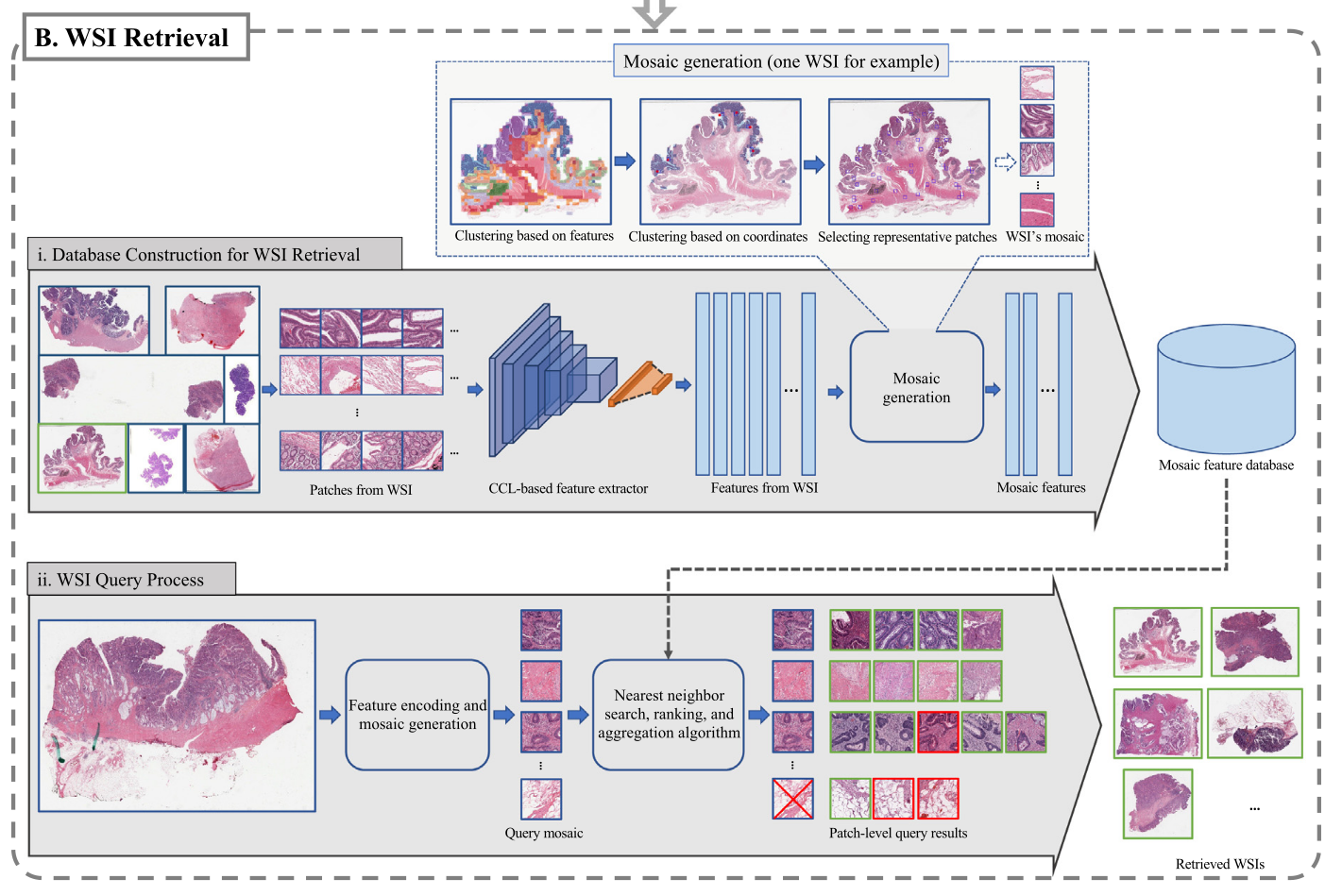
Database Construction for WSI Retrieval
offline database construction 에서 먼저 patchify 하고 CCL-based feature extractor 를 거쳐 나온 feature 로 mosaic generation 을 하는데, mosaic generation 은 dual clustering 으로 이루어져 있다. 먼저 CCL-feature-based K-means clustering 을 수행하고, 그 coordinate 를 받아 spatial-coordinate-based clustering algorithm 을 수행한다. 그 결과 distinctive patches (called a mosaic) 이 생성되고 full WSI 를 대표하게 되는 것이다.

역시 말보단 알고리즘이지. FeatureKMeans 함수로 K1 개의 cluster 로 나눈 뒤, 이를 SpatialKMeans 를 거쳐 다시 각 cluster 를 4-5개의 cluster 로 나눈다. 즉, 4-5개 * K1 개의 cluster centroid 가 생기고 이것이 distinctive patch 가 되는 것이다.
WSI Query Process
WSI database construction 이 완료되고, online 으로 patch-level retrieval 을 시작한다. nearest neighbor searching method 를 사용하고, 이 retrieved patch 와 meta-information 으로 ranking and aggregation algorithm 을 활용해 가장 비슷한 WSI 를 찾아준다.

이 부분도 알고리즘으로 보는게 이해가 잘된다. WSI image query 를 받으면, 각 patch k 개에 대해 feature vector 를 얻고, 이에 대한 retrieval bag 을 k개 얻는다. bag 1개 당 t 개의 retrieved patch 가 있는데 bag 마다 다르다고 한다. bag 1개의 retrieved patch 와 WSI 의 patch 사이의 cosine similarity d 를 구하고, 이 d 와 diagnosis 정보를 활용하여 probability pm 을 계산한다.
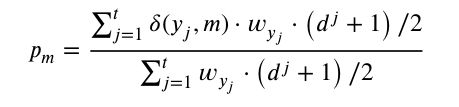
이 pm 은 결국 요약해서 말하면, bag 안에서 m번째 diagnosis type 의 확률이다. yj 는 database 로부터 나온 diagnosis information (bag 의 정보) 이다. 는 두 개의 input 이 같으면 1 을 뱉고 다르면 0을 뱉는 일종의 indicator function 이다. 는 occurrence frequency (normalized probability) 인데, 이것도 역시 jth diagnosis type 이 얼마나 database 에 있는지를 나타낸다. 는 [-1,1] 인 cosine similarity 의 range 를 [0,1] 로 transform 해준 것이다. 간단히 생각해서 자체를 j 번째 sample 에 대한 score 라고 치환해서 생각하면 된다. 그럼 식 자체는 전체 score 의 합 중에서, m 번째 diagnosis subtype 에 포함된 score 의 합이 되어 m 번째 diagnosis type 의 확률로 여길 수 있다.
이 pm 을 이용해서 entropy 를 각 bag 에 대해서 계산하고, ranking 즉 reorder 한다. 이후 procedure REMOVE BAGS WITH LOW QUALITY 에서, AveTop function 을 이용해서 criterion 를 구하는데, AveTop function 은 각 bag 마다 상위 5개의 cosine similarity 를 평균 낸 것이다. 이 criterion 을 넘지 못하는 bag 들은 remove 한다. 이 bag 내부에서 top 5개의 patch 를 고르는 것으로 마무리가 된다.
Experimental results and discusions
Datasets
- TCGA
- PAIP
- UniToPatho
- TissueNet
- DiagSet-A.2.
Experimental metric
다음 두가지 (많이 쓰인다고 하는) Acc@k (top-k accuracy) and mMV@k(majority vote at the top k search results) 를 썼다.
- Acc@k: retrieved similar image 가 query image와 correct label 이면 높게 측정
- mMV@k: more strict metric, majority 가 query image 와 correct label 이어야 높게 측정
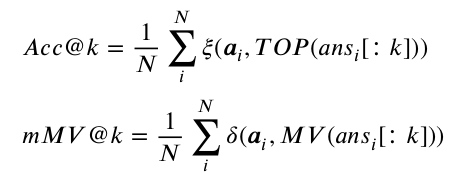
Results of patch-level retrieval
Effect of network components
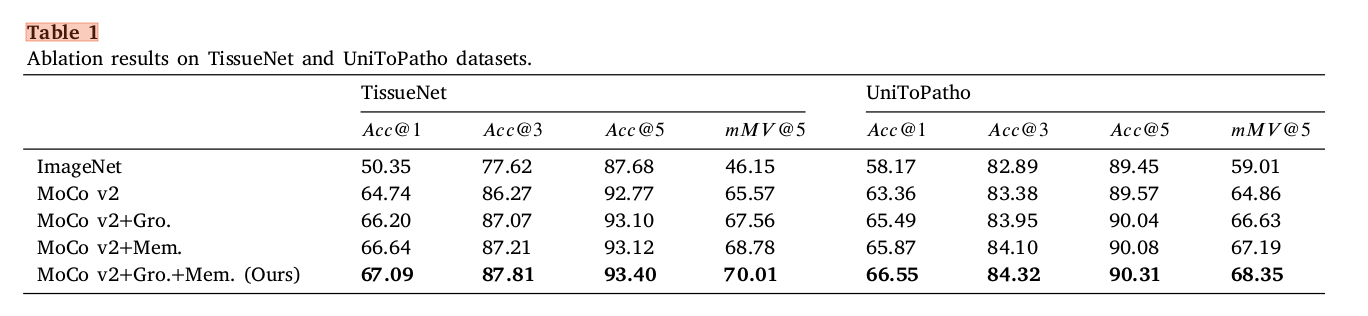
key innovation 인 clustering-based memory bank construction (Mem) 을 추가했을 때와 group-level InfoNCE (Gro) 를 추가했을 때 baseline 보다 성능이 향상되었고 같이 혼합해서 사용했을 때 가장 높았다.
이외에도 다양한 hyperparameter 및 setting 에 대한 abltation study 를 진행했고, 논문에 자세히 언급되어 있다.
Comparison between our CCL and other SSL-based feature extractors
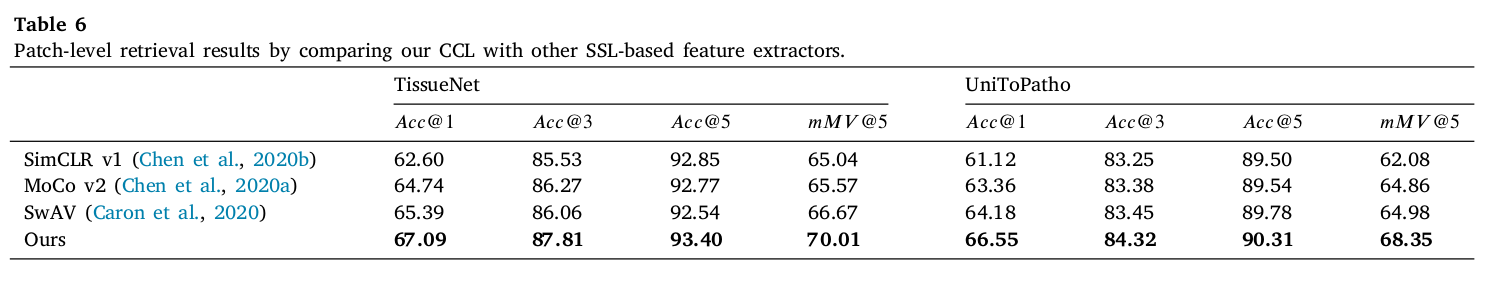
SimCLR v1, SwAV, Moco v2 과 CCL-based feature extractor 의 retrieval 성능을 비교했다. 성능면에서 측정한 모든 metric 에 대해 가장 좋은 성능을 보여줬고, 특히 가장 strict 한 metric 인 mMV 에서의 성능 향상이 컸다.
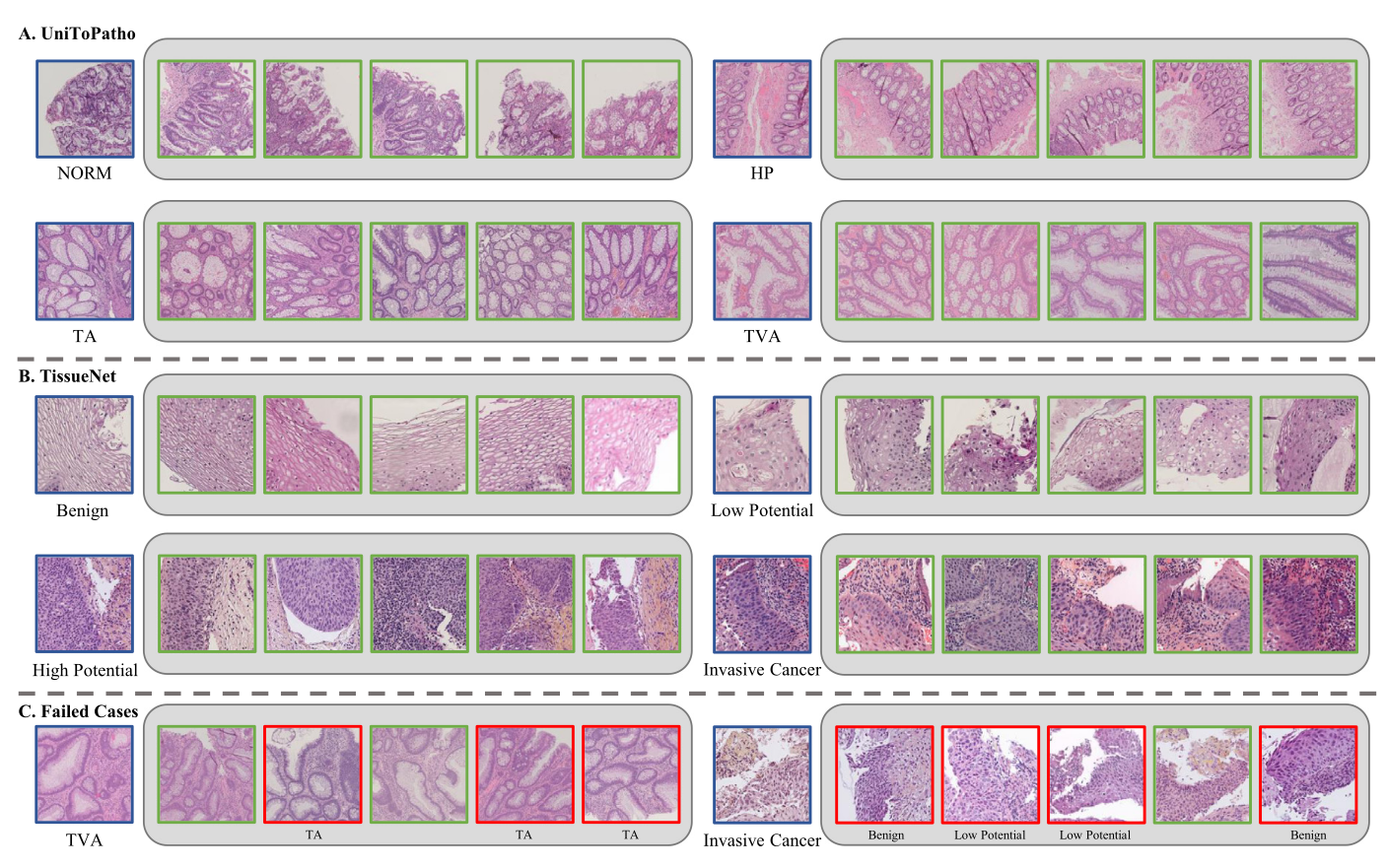
UniToPatho (normal, hyperplastic polyp, tubular adenoma, tubulo-villous adenoma) 에서 각 subtype 을 nice 하게 잘 찾은 것을 볼 수 있다. 또한 TissueNet의 결과를 봤을 때, texture 와 color 가 많이 다름에도 잘 찾은 것을 볼 수 있다. 그 아래 failed case 가 소개 되어 있는데, 저자는 실패했음에도 morphological feature 가 pathologists 간의 disconcordance 를 야기할 정도로 굉장히 비슷했다고 주장한다.
Results of WSI retrieval
1) searching for anatomic sites
2) searching for cancer subtypes based on the same human site
Results of anatomic site retrieval
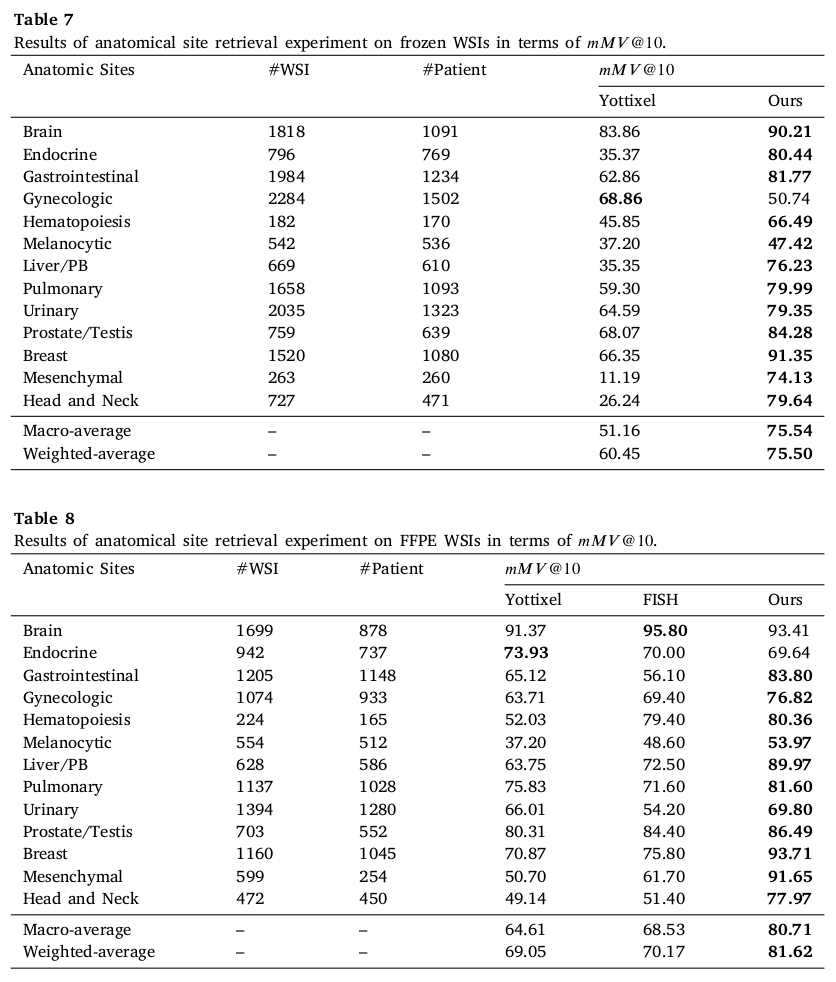
앞선 Yottixel 과 FISH 에서 사용했던 frozen and FFPE WSIs in TCGA 를 사용해서 성능 측정이 이루어졌다. 당연하게도 database 는 모든게 포함된 database 가 아니라 TCGA 3만장 (FFPE 11791, frozen 15237) 이 포함된 database 에서 진행했다고 한다. 결과 20% 넘는 성능향상까지 보여주었다.
Results of cancer subtype retrieval
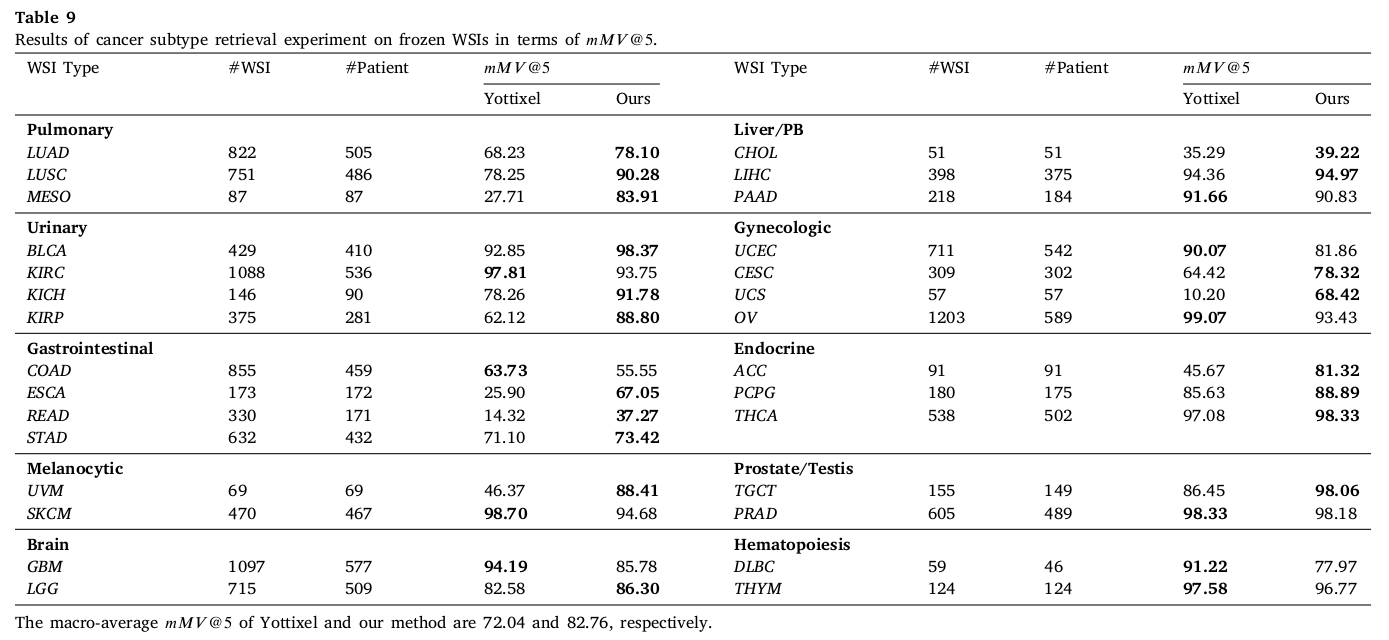
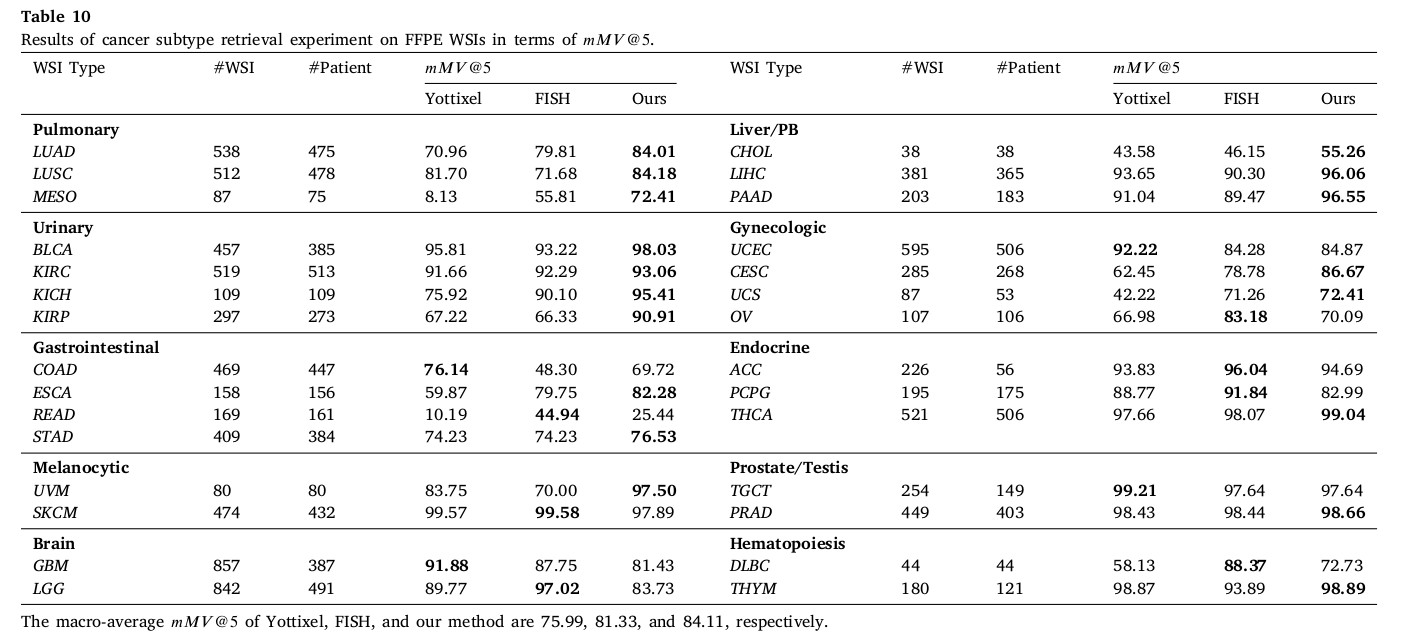
각 frozen 과 FFPE WSI 에 대해서 +10% than Yottixel and +3% than FIST in FFPE,
+40% improvement on specific subtype such as MESO 등 큰 성능 향상을 보였다.
Interpretability analysis
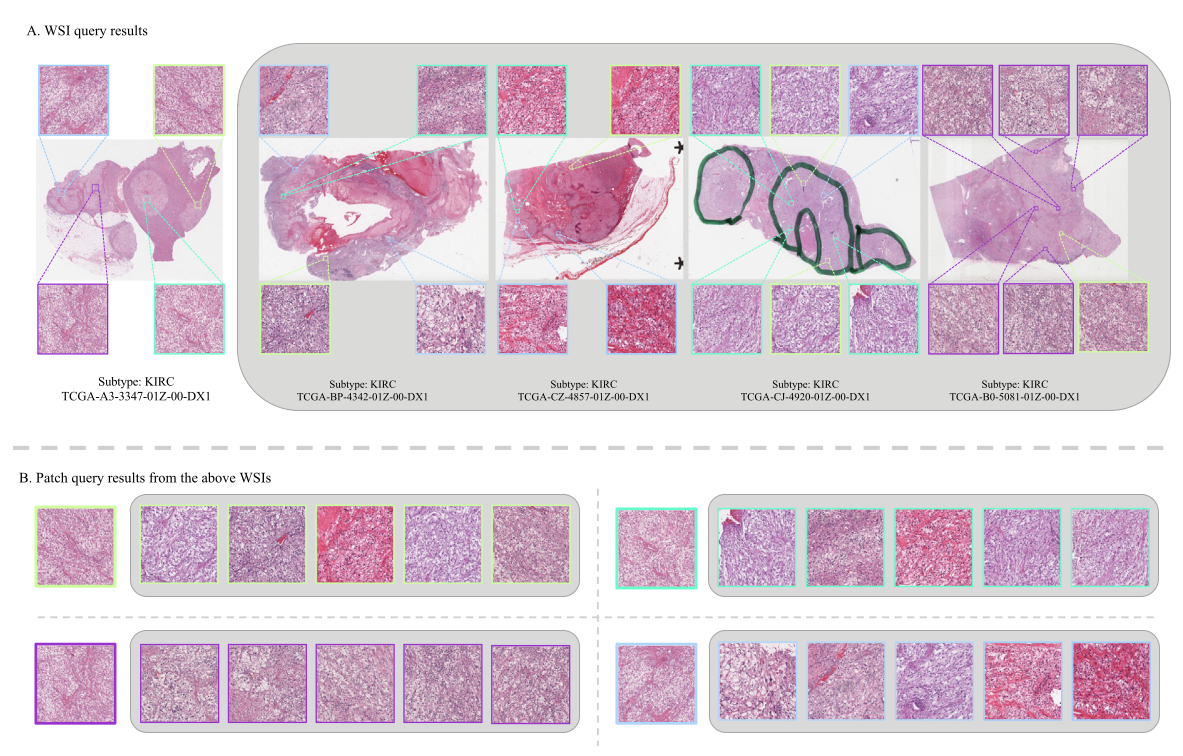
Results of downstream classification task
제안한 SSL pre-trained feature extractor 가 다른 downstream task 에서도 유용하게 사용될 수 있음을 보였다.
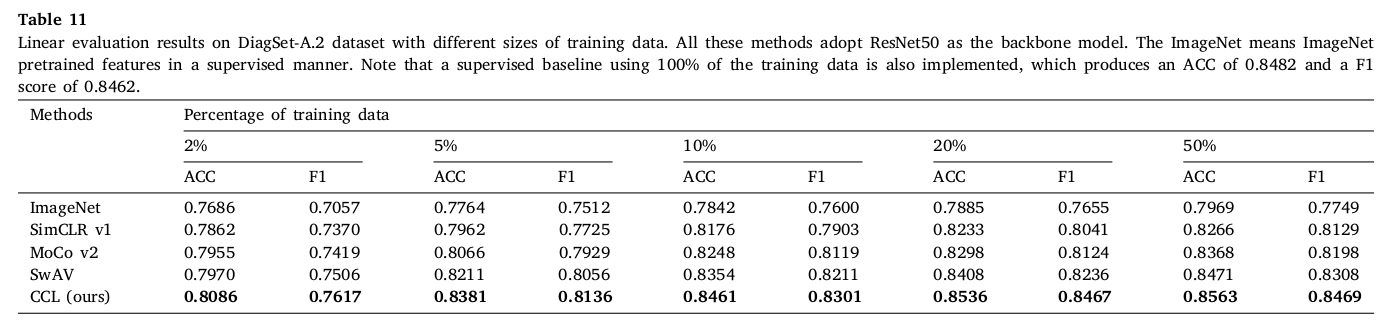
DiagSet-A.2 dataset 에서 four-class classification task 를 진행했고 그 결과를 나타내었다. highest 를 보였던 SwAV 보다도 성능이 높게 측정되었고, 20% 데이터를 training 에 사용했을 때 ImageNet 이 100% 를 사용했을 때 (supervised learning setting) 보다 성능이 높게 측정되어, SSL 방식의 효용성을 보였다.
Conclusion
WSI-level, patch-level 에서 사용가능한 histopathological image retrieval algorithm 을 제안했고, visually interpretable result 를 보여주었다. CCL-based backbone model 을 새로 고안했으며, database construction 후 ranking, curation, and aggregation 이 포함된 retrieval algorithm 을 사용했다. 이는 current WSI retrieval method에서 보여주는 성능을 큰 폭으로 뛰어넘었고 feature 는 다른 downstream task 에서 사용될 수 있을만큼의 potential 을 보여주었다.
와 진짜 실험하느라 힘들었겠다
