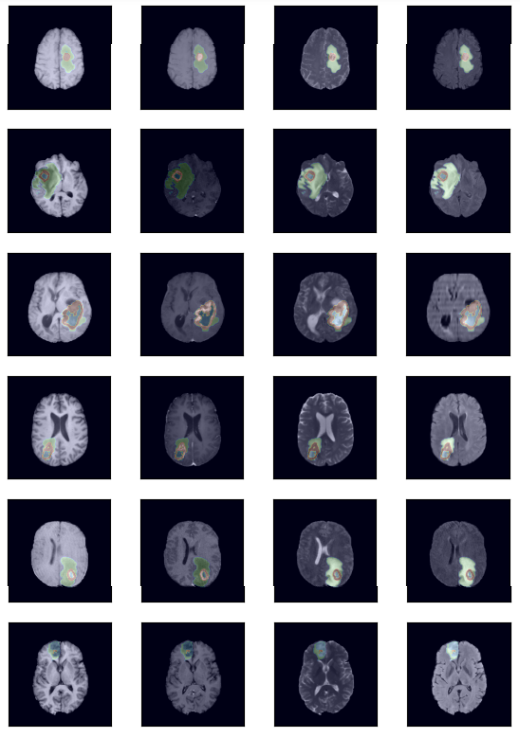
Swin UNETR은 MRI 사진에서 뇌 종양의 위치를 segmentation하는 인공지능 기술이다. 이는 Brain Tumor Segmentation 분야에서 SOTA를 달성했으며 Multi-Organ Segmentation에서도 SOTA를 달성했다.
이름만 봐도 UNETR에 Swin을 적용한 모델이라는 것을 알 수 있다. 기존의 Medical Vision AI에서는 Transformer와 U-NET을 활용해서 segmentation을 수행해왔다. 그러나 Transformer의 구조적 특성 상 ViT와 같은 모델을 사용할 때 세밀한 segmentation이 어렵다는 단점이 있었다. 이를 해결하기 위해서 Swin-Transformer 구조를 이용해 더 세밀한 segmentation을 수행할 수 있도록 했다.
배경 지식
Swin-Transformer
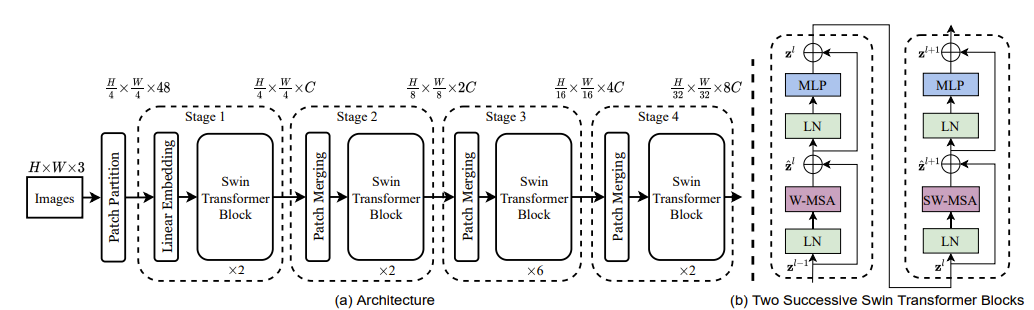
Swin-Transformer의 구조이다. input images를 patch로 만들고 이를 Swin transformer block에 넣는 것을 반복한다. 중간에 Patch merging 작업을 통해 Patch를 합치며 크기를 늘리고 나서 이를 block에 넣는다.
3D U-Net
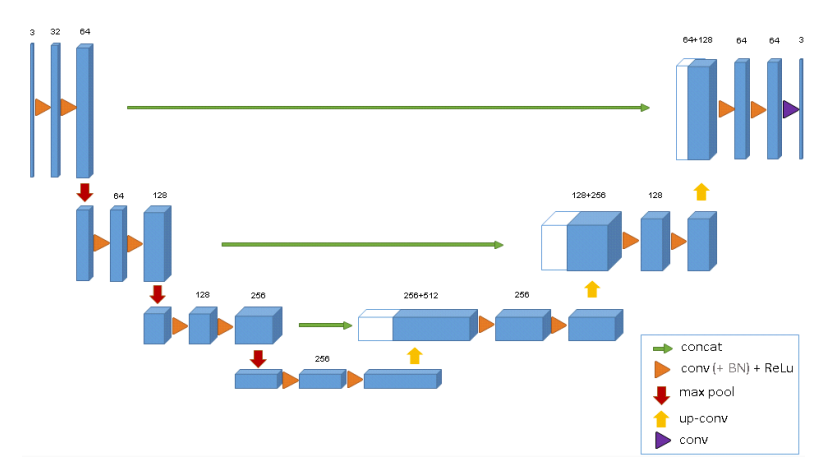
segmentation을 목적으로 하는 모델로, input과 output의 차원이 동일해야 한다. (약간의 resizing은 허용되지만 어쨋든...) 그래서 CNN을 통과하면서 줄어든 차원을 다시 늘려주는 과정이 필요한게 그것을 위와 같이 표현하면 그 모양이 U와 같다고 해서 U-net이라 한다.
U-Net의 단계는 다음과 같다.
1. Input images에서 feature extraction을 수행한다. (CNN - batch normalization, ReLU) - feature map 크기 유지 / channel 증가
2. Max Pooling으로 feature map 축소
3. 1, 2 반복 -> 반복 횟수 N번
4. 3번에서 반복 수행하면서 얻은 N개의 결과를 Up convolution 연산. Upsampling - ConvTranspose(Deconvolution) 사용
5. Upsampling 결과는 N-1번째 결과와 차원이 같다.
6. N-1번째 결과와 Upsampling 과정의 channel을 연결 (conv + Batch normalization + ReLU + Upsampling)
7. 차원 복구 후 channel 동일하게 만들어서 output을 낸다.
Method
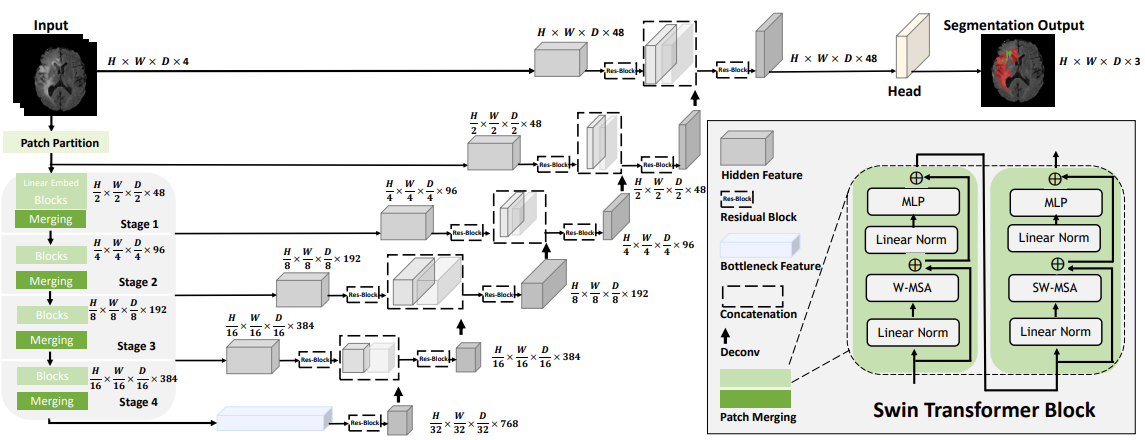
Swin UNETR의 구조는 위와 같다.
1. Patch partition: 3D token에 대한 sequence를 만듦.
2. Encoder
- Swin Transformer Block: W-MSA, SW-MSA 메커니즘 적용
-> 총 4개의 중간 output을 skip-connection에 이용
- Decoder
- 3D U-Net: 각각의 sequence를 다시 3D voxel 형태로 reshape -> deconvolution 통해서 upsampling
-> 4번의 skip-connection 후 voxel 사이즈 복원
- 1x1x1 conv layer와 sigmoid를 통해 최종 output으로 HxWxDx3 shape을 만듦.
MRI dataset은 channel을 4로 해야 한다.
위의 Result의 channel 3은 segmentation 대상이 3개여서 그렇다.
W-MSA
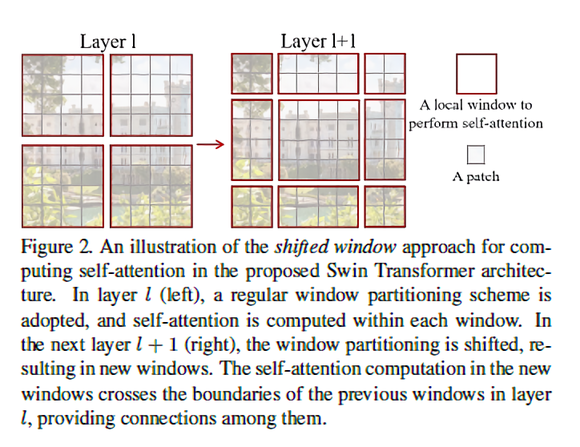
같은 window 내에 있는 patch끼리는 연관성이 높다고 판단하여 window 안에서만 self-attention을 수행함.
SW-MSA
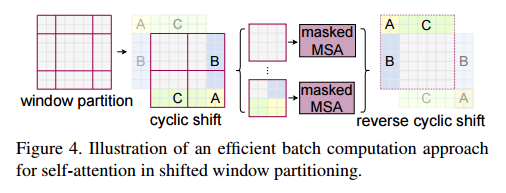
shift mechanism을 이용해서 window를 재배치한 후, 재배친한 곳의 patch들은 전부 masking한 후 self-attention을 수행함.
Results