1.학습한 내용
1)L1제약 , L2제약
L1 제약은 제약이 강하게 걸수록 강하게 걸린다.
너무 강하게 걸면 0이 될수도있다.
예를들어 Lasso 가 있다.
L2 제약은 우리가 사용하려는 feature들의 속성중에서 전체적인 속성에 평균적으로 다 제약을 거는것. 0이 되지 않는다.
예를들어 Lidge 가 있다.
2)선형회귀 Linear Regression
이전의 수업을 이어서 하는것으로, 우선 라이브러리를 불러온다.
from sklearn.linear_model import LogisticRegression
from sklearn.svm import LinearSVC
X,y = mglearn.datasets.make_forge()
fig,axes = plt.subplots(1,2,figsize=(10,3))
for model, ax in zip([LinearSVC(max_iter=5000),LogisticRegression()],axes):
clf = model.fit(X,y)
mglearn.plots.plot_2d_separator(clf,X,fill=False,eps=0.5,alpha=0.7,ax=ax)
mglearn.discrete_scatter(X[:,0],X[:,1],y,ax=ax)그리고 사용할 데이터셋을 불러온다.
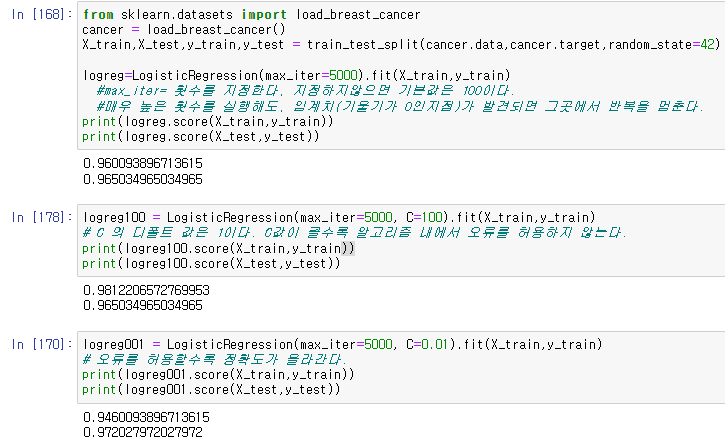
from sklearn.datasets import load_breast_cancer
cancer = load_breast_cancer()
X_train,X_test,y_train,y_test = train_test_split(cancer.data,cancer.target,random_state=42)
logreg=LogisticRegression(max_iter=5000).fit(X_train,y_train)
#max_iter= 횟수를 지정한다, 지정하지않으면 기본값은 100이다.
#매우 높은 횟수를 실행해도, 임계치(기울기가 0인지점)가 발견되면 그곳에서 반복을 멈춘다.
print(logreg.score(X_train,y_train))
print(logreg.score(X_test,y_test))이 때 기존의 데이터에서 C, 오류를 허용하는 정도의 값을 추가로 입력한다.
logreg100 = LogisticRegression(max_iter=5000, C=100).fit(X_train,y_train)
print(logreg100.score(X_train,y_train))
print(logreg100.score(X_test,y_test))C= 100 일때,
logreg001 = LogisticRegression(max_iter=5000, C=0.01).fit(X_train,y_train)
# 오류를 허용할수록 정확도가 올라간다.
print(logreg001.score(X_train,y_train))
print(logreg001.score(X_test,y_test))C = 0.01 일때,
그림과 같이 C를 높게 적용하게 되면 훈련데이터의 정확도가 올라가고, C를 낮게 적용하면 훈련데이터의 정확도가 낮아지며, 그에 따라서 테스트 데이터의 정확도가 바뀌는것을 확인할수 있었다.
2-1) 3개 이상의 군집을 가진 선형회귀
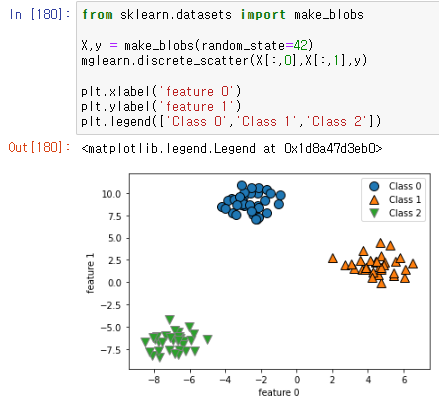
3개 이상의 군집이 있을때, 어떻게 선을 그어서 분류를 할것인지를 확인하기 위해서. 3개의 군집을 가진 데이터값을 가져온다.
from sklearn.datasets import make_blobs
X,y = make_blobs(random_state=42)
mglearn.discrete_scatter(X[:,0],X[:,1],y)
plt.xlabel('feature 0')
plt.ylabel('feature 1')
plt.legend(['Class 0','Class 1','Class 2'])
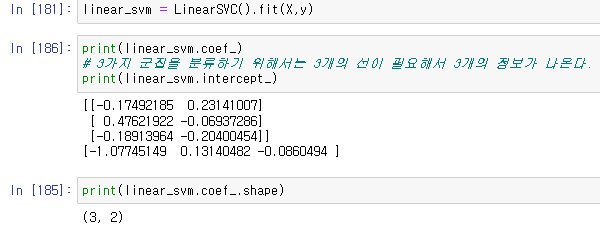
그 후 선을 입력하고 그 선의 정보들을 확인한다.
linear_svm = LinearSVC().fit(X,y)
print(linear_svm.coef_)
print(linear_svm.intercept_)
print(linear_svm.coef_.shape)
이때, 3개의 군집을 선형회귀 하게되면 3개의 선이 필요하여서 3개의 정보가 나오는것을 확인할수 있다.
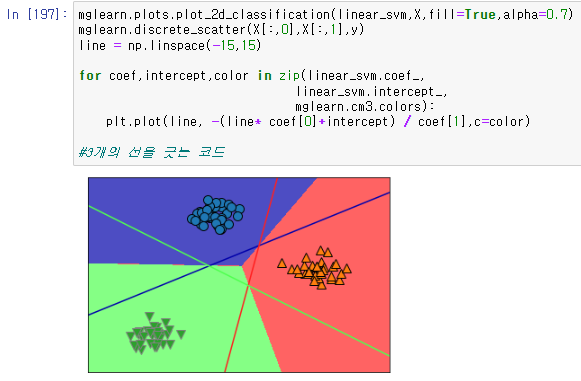
이것을 시각적으로 확인하기 위해서 선을 긋고 분류하는것을 볼수있는 코드를 입력한다.
mglearn.plots.plot_2d_classification(linear_svm,X,fill=True,alpha=0.7)
mglearn.discrete_scatter(X[:,0],X[:,1],y)
line = np.linspace(-15,15)
for coef,intercept,color in zip(linear_svm.coef_,
linear_svm.intercept_,
mglearn.cm3.colors):
plt.plot(line, -(line* coef[0]+intercept) / coef[1],c=color)
3개의 선을 그어서 분류를 한뒤
군집에 따라서 배경색을 칠하는것으로 더 눈에 띄게 분류하는것을 확인할수 있다.
3) Tree 트리 알고리즘
알고리즘이 거꾸로하면 Tree 의 형태와 닮았다고 하여 이름 지어진 알고리즘이다.
이를 우선 라이브러리에서 불러온다.
from sklearn.tree import DecisionTreeClassifier이때 DecisionTreeClassifier 는 의사결정나무 이다.
그리고 Tree 알고리즘을 사용할 데이터셋을 불러온다.
from sklearn.datasets import load_breast_cancer
cancer = load_breast_cancer()불러온 데이터를 분류한다.
X_train,X_test,y_train,y_test = train_test_split(cancer.data,
cancer.target,
random_state=42)
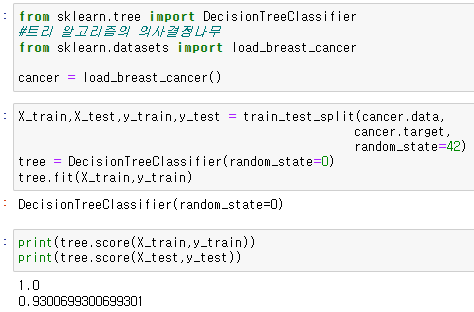
tree = DecisionTreeClassifier(random_state=0)
tree.fit(X_train,y_train)이 tree 알고리즘을 사용한 값의 정확도를 확인한다.
print(tree.score(X_train,y_train))
print(tree.score(X_test,y_test))
학습용 데이터의 정확도가 100퍼센트, 테스트 데이터의 정확도가 93% 가 나오는것을 확인할수있다.
이때의 값은 tree 가 나뉘는 깊이, 횟수를 지정하지 않아서 모두가 나뉠때까지 진행된것이기 때문에 학습용 데이터의 정확도가 100퍼센트가 나오는것으로, 이때 조금이라도 다른 데이터가 들어가면 오류가 일어날 가능성이 높기 때문에,
그 깊이depth 를 설정을 한다.
tree =DecisionTreeClassifier(max_depth=4, random_state=0)
tree.fit(X_train, y_train)
print(tree.score(X_train,y_train))
print(tree.score(X_test,y_test))
이때 학습용 데이터의 정확도는 내려갔지만, 테스트 데이터의 정확도는 오히려 올라간것을 확인할수있다.
이제 트리를 그리는 것을 해보기 위해서
트리를 그리는 라이브러리를 불러온다.
from sklearn.tree import plot_tree이를 통해 그림을 그린다.
plot_tree(tree,class_names=['A','B'], filled=True, fontsize=5, feature_names=cancer.feature_names)
이를 통해서 트리를 그리는것이 가능해졌다.
OS
os 모듈은 Operating System의 약자로서 운영체제에서 제공되는 여러 기능을 파이썬에서 수행할 수 있게 해준다.
import os그리고 알고리즘을 사용할 데이터를 불러온다.
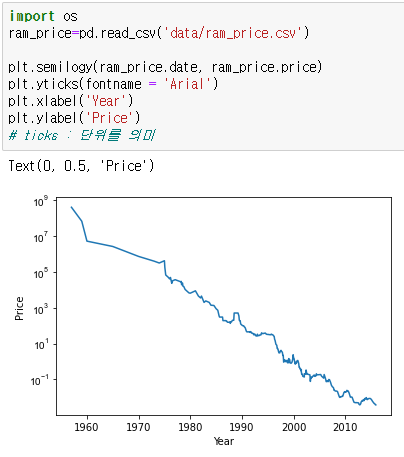
ram_price=pd.read_csv('data/ram_price.csv')이것은 연도별 ram의 가격 데이터이다.
그리고 이 데이터로 그래프를 그린다.
plt.semilogy(ram_price.date, ram_price.price)
plt.yticks(fontname = 'Arial')
plt.xlabel('Year')
plt.ylabel('Price')
연도가 지나갈수록 ram의 가격이 내려가는것을 확인할수있다.
tree 알고리즘을 불러온 뒤 데이터분류를 한다.
from sklearn.tree import DecisionTreeClassifier
data_train =ram_price[ram_price.date < 2000]
data_test = ram_price[ram_price.date >= 2000]이때 데이터의 배열이 series 배열이므로 numpy 배열로 바꾼다.
X_train = data_train.date.to_numpy()[:,np.newaxis]
y_train = np.log(data_train.price)필요한 라이브러리를 불러오고,
분류된 데이터를 바탕으로 알고리즘을 적용한다.
from sklearn.linear_model import LinearRegression
from sklearn.tree import DecisionTreeRegressor
tree = DecisionTreeRegressor().fit(X_train,y_train)
linear_reg = LinearRegression().fit(X_train,y_train)
X_all = ram_price.date.to_numpy()[:,np.newaxis]이를 통해서 예측값을 만든다.
pred_tree = tree.predict(X_all)
pred_lr = linear_reg.predict(X_all)또한 데이터를 만드는 과정에서 price 의 값은 log를 취하였기 때문에, 이를 원래대로 하기위해서 exp를 취해준다.
price_tree = np.exp(pred_tree)
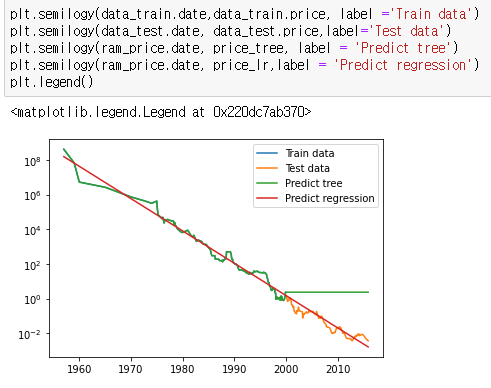
price_lr = np.exp(pred_lr)이렇게 분류한 데이터를 시각화시키기 위해서 그래프를 그려본다.
plt.semilogy(data_train.date,data_train.price, label ='Train data')
plt.semilogy(data_test.date, data_test.price,label='Test data')
plt.semilogy(ram_price.date, price_tree, label = 'Predict tree')
plt.semilogy(ram_price.date, price_lr,label = 'Predict regression')
plt.legend()4) Forest 숲
tree 나무들이 모여서 forest 숲이 된다.
forest 라이브러리와, 필요한 데이터셋을 불러온다.
from sklearn.ensemble import RandomForestClassifier
from sklearn.datasets import make_moons
X, y =make_moons(n_samples=100, noise=0.25, random_state=3)이때 만들어내는 데이터셋이 너무 과적합한 데이터가 나올수있으므로 임의의 오차로 noise를 설정한다.
만들어낸 데이터셋의 데이터를 분류하고 그를 통해서 forest 알고리즘을 사용한다.
X_train , X_test, y_train , y_test = train_test_split(X,y, random_state=42)
forest = RandomForestClassifier(n_estimators=5, random_state=2)
forest.fit(X_train,y_train)이때 n_estimators 는 몇개의 tree 를 사용할것인지를 지정하는것으로, default 값은 100이다.
이제 나온 값을 시각화 하고 비교하기 위해서 subplot 을 통해서 여러개의 그림을 그린다.
fig, axes = plt.subplots(2,3,figsize=(20,10))
for i,(ax,tree) in enumerate(zip(axes.ravel(),forest.estimators_)):
ax.set_title('tree {}'.format(i))
mglearn.plots.plot_tree_partition(X,y,tree,ax=ax)
mglearn.plots.plot_2d_separator(forest, X, fill=True, ax=axes[-1,-1],
alpha=0.4)
mglearn.discrete_scatter(X[:,0],X[:,1],y)이때 사용한 enumerate는 열거형 으로 만들어준다. 통째로 자료형으로 만들어준다. 의 기능을 가진다.
이때 5번째까지의 그림은 트리의 갯수를 5개로 지정했을때의 각각의 트리의 형태를 반복문을 통해서 그린것이고, 6번째의 값은 앞의 트리들의 값들을 총합해서 그려진 값이다.
위의 경우는 트리를 5개로 하여 시각화 하기 위한것이었고,
이번에는 트리를 5개가 아닌 100개를 사용하는 forest 로 데이터분류를 진행한다.
X_train , X_test , y_train, y_test = train_test_split(
cancer.data,cancer.target,random_state=0)
forest = RandomForestClassifier(n_estimators=100, random_state=0)
forest.fit(X_train,y_train)이를 그래프로 보기위해서 함수를 정의한다.
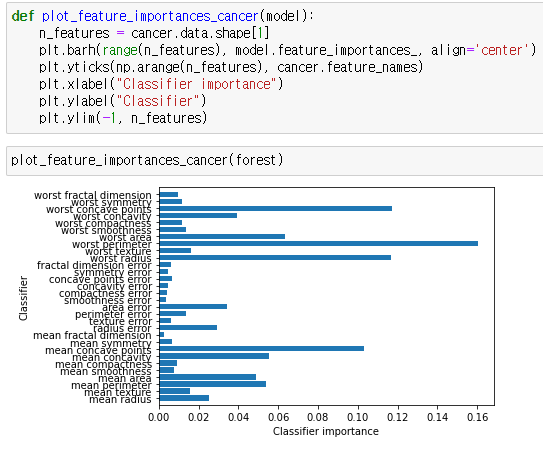
def plot_feature_importances_cancer(model):
n_features = cancer.data.shape[1]
plt.barh(range(n_features), model.feature_importances_, align='center')
plt.yticks(np.arange(n_features), cancer.feature_names)
plt.xlabel("Classifier importance")
plt.ylabel("Classifier")
plt.ylim(-1, n_features)이 plot_feature_importances_cancer() 함수를 사용한다.
plot_feature_importances_cancer(forest)그래디언트부스팅 라이브러리를 불러온 뒤, gradientboosting 알고리즘을 사용하기 위한 데이터를 가져온다.
from sklearn.ensemble import GradientBoostingClassifier
X_train , X_test , y_train, y_test = train_test_split(cancer.data,
cancer.target,
random_state=0) gradientboosting 알고리즘을 사용한다.
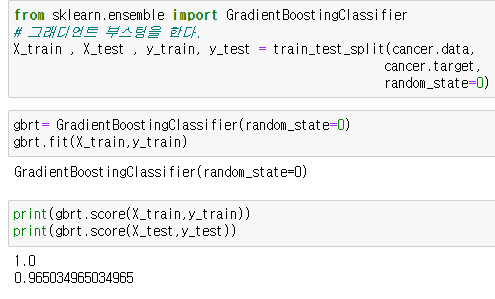
gbrt= GradientBoostingClassifier(random_state=0)
gbrt.fit(X_train,y_train)이후 나온 데이터값의 정확도를 확인한다.
print(gbrt.score(X_train,y_train))
print(gbrt.score(X_test,y_test))
상당히 높은 정확도를 가지는것을 확인 할수 있다.
이때 max_depth 로 깊이를 1로 제약을 걸어본다.
gbrt= GradientBoostingClassifier(random_state=0, max_depth=1)
gbrt.fit(X_train,y_train)
print(gbrt.score(X_train,y_train))
print(gbrt.score(X_test,y_test))
깊이를 얕게 제약을 걸어도 정확도가 높게 나오는 알고리즘이란것을 알수있다.
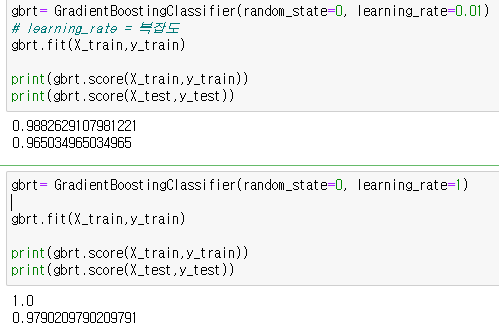
이때, 깊이가 아닌 복잡도, learning_rate 를 설정한다.
우선 0.01로 설정을 한다.
gbrt= GradientBoostingClassifier(random_state=0, learning_rate=0.01)
gbrt.fit(X_train,y_train)
print(gbrt.score(X_train,y_train))
print(gbrt.score(X_test,y_test))이번에는 1로 설정을 한다.
gbrt= GradientBoostingClassifier(random_state=0, learning_rate=1)
# learning_rate = 복잡도
gbrt.fit(X_train,y_train)
print(gbrt.score(X_train,y_train))
print(gbrt.score(X_test,y_test))
이를 통해서 복잡도를 설정하는 것에 따라서 정확도의 값이 바뀌는것을 확인할수있다.
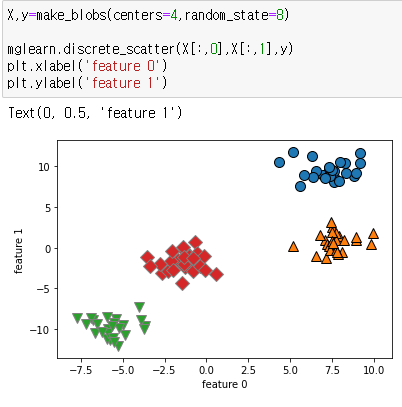
군집을 4개로 지정을 하고 데이터를 불러온다.
X,y=make_blobs(centers=4,random_state=8)
mglearn.discrete_scatter(X[:,0],X[:,1],y)
plt.xlabel('feature 0')
plt.ylabel('feature 1')
centers 를 설정함으로써 군집의 갯수를 정해서 4개로 출력을 할수있는데,
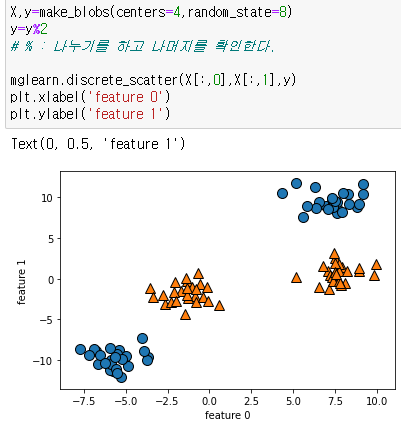
이때 이들을 홀수번째와 짝수번째를 묶어내서 같은 군집으로 만드는 것 이 가능하다.
X,y=make_blobs(centers=4,random_state=8)
y=y%2
# % : 나누기를 하고 나머지를 확인한다.
mglearn.discrete_scatter(X[:,0],X[:,1],y)
plt.xlabel('feature 0')
plt.ylabel('feature 1')%를 통해서 나누기를 하고 나머지에 따라서 y값, feature1 을 지정한다.
그렇게 되면
0/2 의 나머지는 0, 1/2의 나머지는 1, 2/2의 나머지는 0, 3/2의 나머지는 1이 됨으로써
군집을 묶어내는것이 가능하다.
2.학습내용 중 어려웠던 점
수업도중 강사님이 plot_feature_importances_cancer() 의 코드를 사용할때, 이것이 기존의 라이브러리에 이미 설정되어있는 함수일거라서 작동을 할것인데 하지않는것에 의문을 가지셨다.
3.해결방법
그래서 함수에 대해서 궁금하여서 인터넷 브라우저에 검색을 함으로써, 다른 글들에서는 해당 함수를 먼저 def, 정의를 하고 사용하는것을 확인 하여서 해당 정의하는 코드를 복사해와서 작동을 시켜보았다.
4.학습소감
원하는 결과가 출력되는것을 볼수는 있었지만, 이러한 함수들이 라이브러리에 추가되는것 뿐만이 아니라 사라질수도 있다는 점을 배우게 되었다, 앞으로 배우게 됬을때 무작정 해당 함수가 있다 라고 기억하는것이 아닌 시간대별, 버전별로 추가가 될수도, 삭제가 될수도 있는 가능성이 존재하는것을 알게되었다.