- 데이터 탐색
R 기본 데이터인 ‘Iris’ 데이터를 활용하여 Ensemble 모형을 사용하기에 앞서 데이터의 기본적인 구조를 탐색한다.
Target Variable은 Species로 설정하고, setosa와 nonsetosa로 나눈다.
iris$Species <- as.character(iris$Species)
iris$Species[iris$Species !="setosa"] <- "non setosa"
iris$Species <- as.factor(iris$Species)
- Bagging
library(ipred)
irisbag = bagging(Species ~ ., data=iris, nbagg = 135)
library(caret)
tmp = sample(1:150,120)
iris_tr = iris[tmp,]
iris_test = iris[-tmp,]
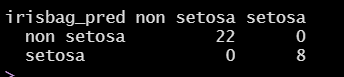
irisbag_pred = predict(irisbag, iris_test)
table(irisbag_pred, iris_test$Species)
#Parameter selection
tr_err = test_err = c()
for (i in 1:150){
irisbag = bagging(Species ~ ., data = iris_tr, nbagg = i)
iris_pred_tr = predict(irisbag, iris_tr)
iris_pred_test = predict(irisbag, iris_test)
tr_err[i] = 1-sum(diag(table(iris_pred_tr, iris_tr$Species)))/nrow(iris_tr)
test_err[i] = 1-sum(diag(table(iris_pred_test, iris_test$Species)))/nrow(iris_test)
}
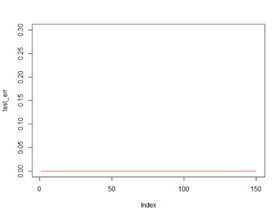
plot(test_err, type="l", ylim=c(0,0.3))
lines(tr_err,col="red")
ctrl = trainControl(method = "cv", number = 10)
train(Species ~ ., data = iris, method = "treebag",
trControl = ctrl)
Parameter selection을 통해 그래프를 확인한 결과 1부터 150까지의 구간에서 같은 error rate와 10 fold-cross validation을 통해 구해진 최적의 tree는 135개로 추정되어 135개로 수행되었다.
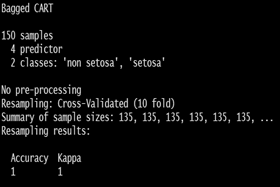
Bagging으로 Species Classification을 진행한 결과, accuracy = 1.0, error rate = 0이다.