Protein Engineering Pipeline
- RFdiffusion
- Backbone or general structure
- ProteinMPNN -> 단백질 구조를 통해, 시퀀스를 생성(단백질 역접힘 문제 예측)
- Sequence Generation
- AlphaFold 2 -> 서열을 통해, 단백질 구조 예측(단백질 접힘 문제 예측)
- Filtering
- Structure prediction problem
- Experimental Validation
다중서열정렬(Multiple Sequence Alignment, MSA)
- Tolerant Position: 서열 중 특정 아미노산이 계속 바뀌어도 영향을 받지 않고, 서열 Position을 계속 유지하는 것
=> 특정 서열이 바뀌어도 전체 구조는 유지되는 특성이 있다(tolerant, 잘 견디는) - Co-Evolution: 서열 중 특정 아미노산이 바뀔때, 구조를 안정화 시키기 위해 근처의 자주 상호작용하는 서열도 같이 바뀐다.
AlphaFold 의 아키텍쳐
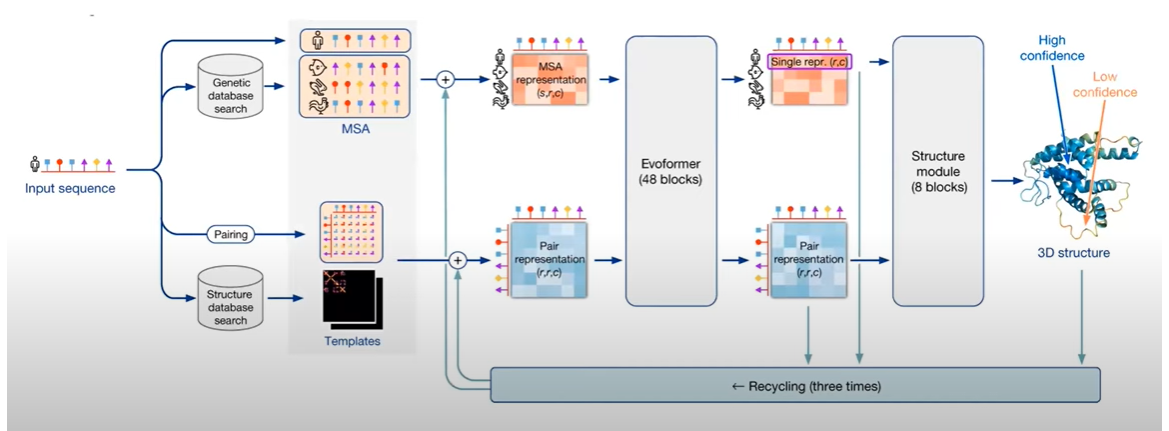
1. Input 모델
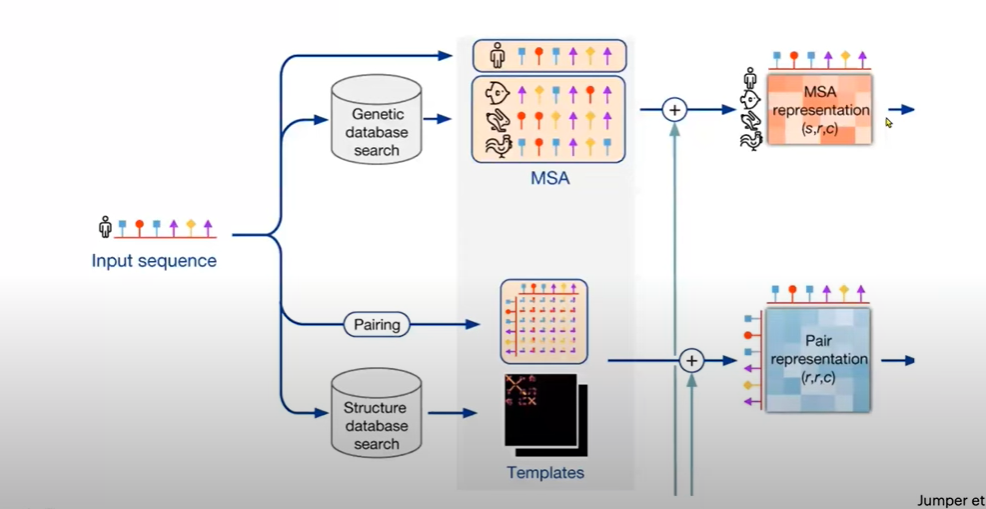
input이 들어오면 2가지로 나뉘어 검색됨
1. 유전자 데이터베이스 검색 -> MSA를 구성하는데 도움
2. 구조 데이터베이스 검색 -> 유사한 구조가 존재하는지 확인
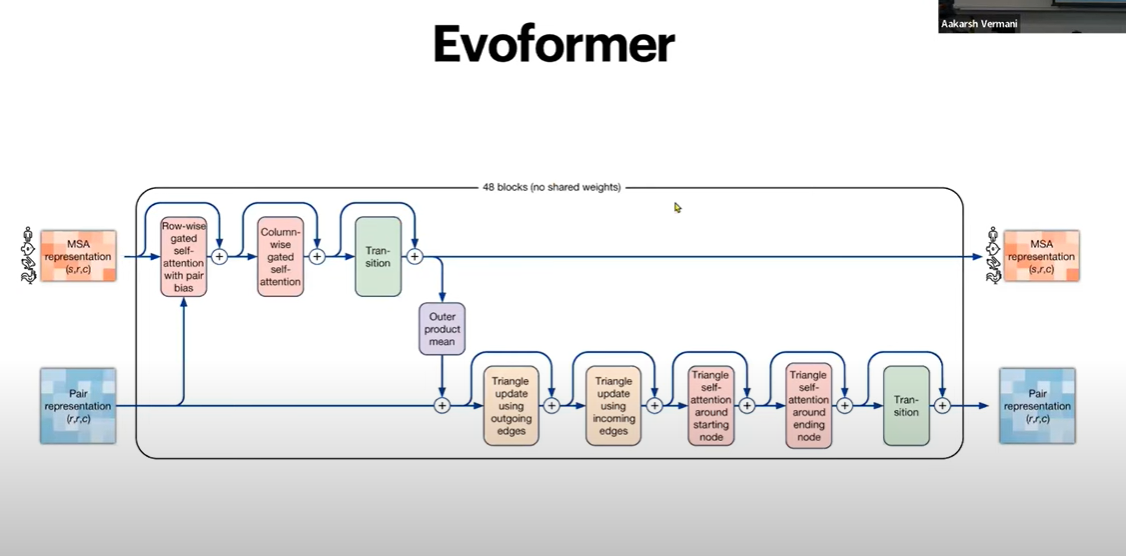
- Evoformer(수정된 transformer)
- self-Attention
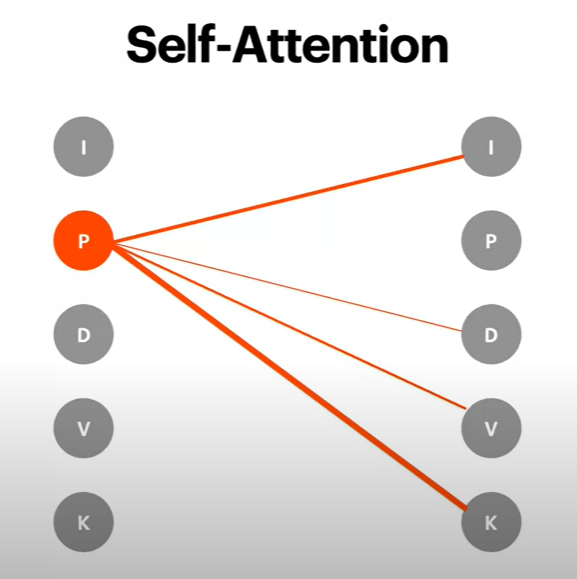
-> 각 항목이 다른 항목들과 서로 얼마나 관련되었고, 중요한지 파악
=> 사슬의 반대쪽 끝에서 서로 상호 작용하는 아미노산이 있을 수도 있기 때문에 거리가 아닌 상호연관성이 더욱 중요
- Evoformer
- Structure Module
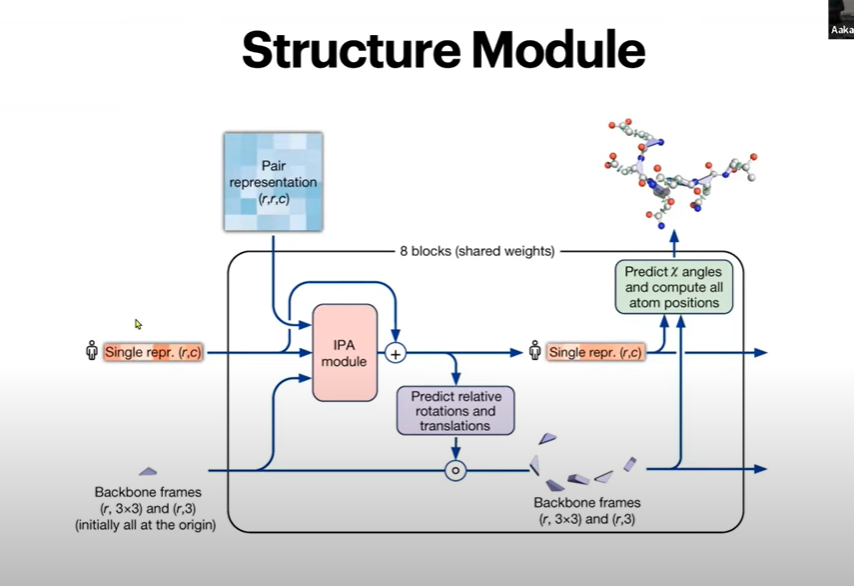
단백질에서 각 아미노산 분자들이 삼각형 구조를 이루는 것을 backbone frame이라 함
IPA 모듈의 출력은 백본 업데이트 알고리즘으로 전달 -> 백본 프레임의 위치와 구조를 업데이트
ProteinMPNN
- 단백질 구조 정보를 통해서, 시퀀스를 생성
- 핵심은 MPNN(Message Passing Neural Network)
=> 정보가 그래프를 따라 전달됨(target 노드와 엣지로 연결된 노드끼리 정보 전달)