scRNA-seq 과 snRNA-seq 차이 및 비교
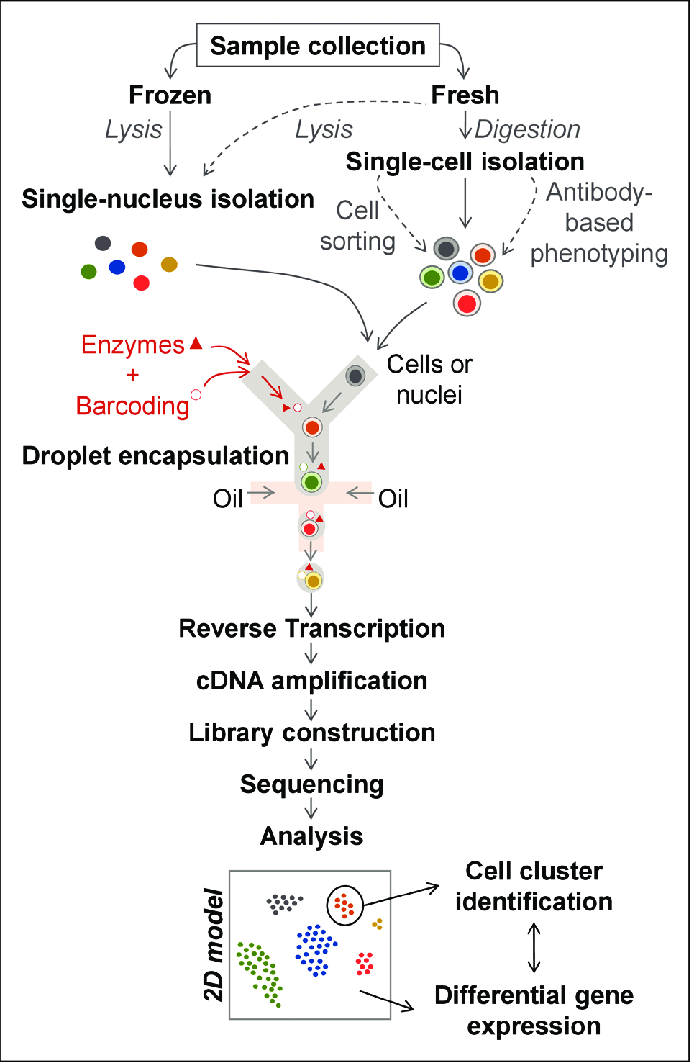
그림 출처: https://www.ahajournals.org/doi/10.1161/ATVBAHA.120.314776
가장 큰 차이점
- single-cell RNA sequencing (scRNA-seq) 의 경우, 세포질과 핵의 transcripts (RNA)를 모두 sequencing
- single-nucleus RNA sequncing (snRNA-seq)은 핵의 transcripts 만을 sequencing
scRNA-seq의 장점과 단점
scRNA-seq은 세포질과 핵의 RNA를 모두 포함하여 보다 포괄적인 전사체 (transcriptome)를 탐색할 수 있는 장점이 있음.
이는 세포의 전체적인 기능을 이해하는 데 유리함.
그러나 이 방법은 신선한 조직 샘플 (fresh tissue sample)이 필요하며, 세포 분리 과정에서 발생하는 스트레스로 인해 데이터에 편향 (bias)이 생길 수 있음.
특히, 세포가 쉽게 분리되지 않는 조직에서는 효율이 떨어질 수 있음
- 예시: 고밀도의 세포 분포에, 세포 간 결합이 강한 조직들 (brain, heart, solid tumor와 같은 tissue)에선 세포가 쉽게 분리되지 않아 효율이 떨어질 수 있음
snRNA-seq의 장점과 단점
snRNA-seq은 핵의 RNA만을 분석하지만, 다양한 샘플 유형을 사용할 수 있어 유연성이 높음.
신선한 샘플뿐만 아니라 동결된 샘플 (frozen sample)이나 고정된 샘플 (fixed sample)에서도 사용이 가능함.
또한, 세포 분리 과정에서 발생하는 스트레스가 적어, 세포의 상태를 보다 정확하게 반영할 수 있음.
그러나 핵에 존재하는 RNA만 분석하기 때문에, 특정 RNA의 발현을 파악하는 데 제한이 있을 수 있습니다.
선택 기준
세포가 쉽게 분리되고, 전체 전사체를 분석해야 하는 경우에는 scRNA-seq이 적합함. (예: 혈액 샘플)
반면, 조직이 분리하기 어렵거나 동결된 샘플을 사용하는 경우에는 snRNA-seq이 더 나은 선택일 수 있음.
연구자는 각 방법의 장단점을 고려하여 가장 적합한 기술을 선택해야 함.
최근 발전
snRNA-seq은 신경계 연구와 같이 어려운 조직을 다루는 연구에서 큰 진전을 보였음. 예를 들어, post-mortem brain 샘플에서 성공적으로 세포를 분리하여 분석할 수 있게 되었음.
또한, snRNA-seq은 전체 포유류 심장을 분석하는 데 성공했으며, 이는 scRNA-seq으로는 어려운 과제였음 (heart tissue를 손상 없이 단일 세포-single cell 단위로 분리하는 것을 매우 어려움)
Reference
-
Wu H, Kirita Y, Donnelly EL, Humphreys BD. Advantages of single-nucleus over single-cell RNA sequencing of adult kidney: Rare cell types and novel cell states revealed in fibrosis. Journal of the American Society of Nephrology. 2019;30(1).
-
Wolfien M, Galow AM, Müller P, Bartsch M, Brunner RM, Goldammer T, et al. Single-nucleus sequencing of an entire mammalian heart: Cell type composition and velocity. Cells . 2020;9(2).
-
Grindberg RV, Yee-Greenbaum JL, McConnell MJ, Novotny M, O’Shaughnessy AL, Lambert GM, et al. RNA-sequencing from single nuclei. Proceedings of the National Academy of Sciences . 2013;110(49).
-
Thrupp N, Frigerio S, Wolfs L, Skene NG, Fattorelli N, Poovathingal S, et al. Single-nucleus RNA-seq is not suitable for detection of microglial activation genes in humans. Cell Reports . 2020;32(13).
-
Andrews TS, Atif J, Liu JC, Perciani, Catia T, Ma X, Thoeni C, et al. Single‐cell, single‐nucleus, and spatial RNA sequencing of the human liver identifies cholangiocyte and mesenchymal heterogeneity. Hepatology Communications . 2022;6(4).
-
Wen F, Tang X, Xu L, Haixia Q. Comparison of single nucleus and single cell transcriptomes in hepatocellular carcinoma tissue. Molecular Medicine Reports. 2022;26(5):339.

감사합니다.