Transcriptome
1.single cell vs. single nucleus RNA sequencing 차이
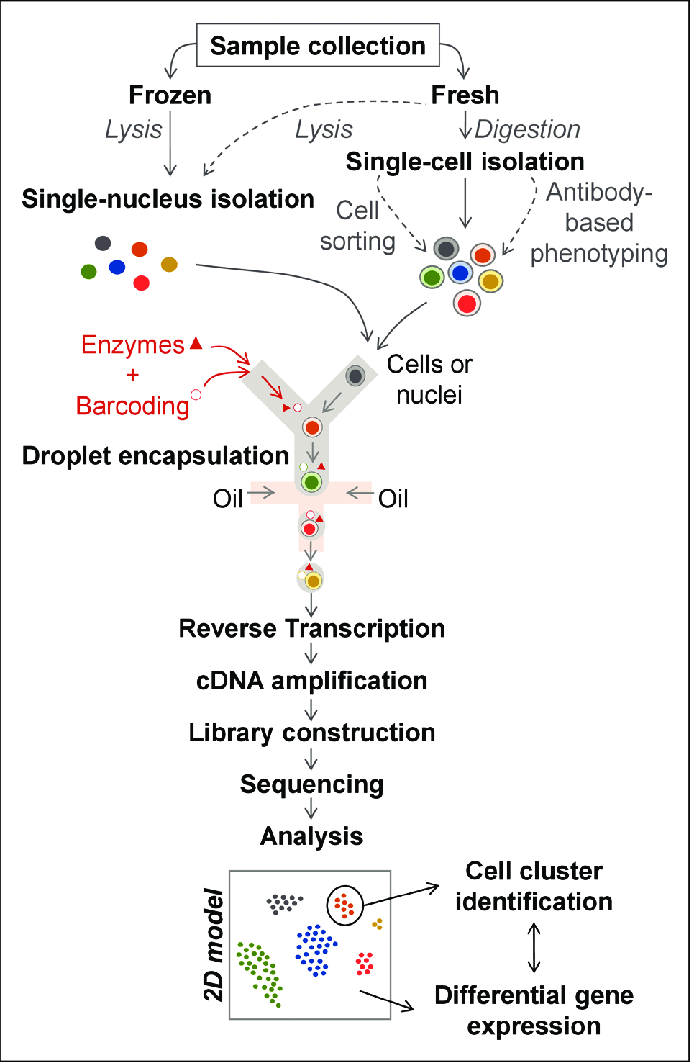
scRNA-seq 과 snRNA-seq 차이 및 비교전체 글 내용 출처 / 원문 확인하기그림 출처: https://www.ahajournals.org/doi/10.1161/ATVBAHA.120.314776single-cell RNA sequencing (scR
2.Differentially Expressed Genes 개념과 가정
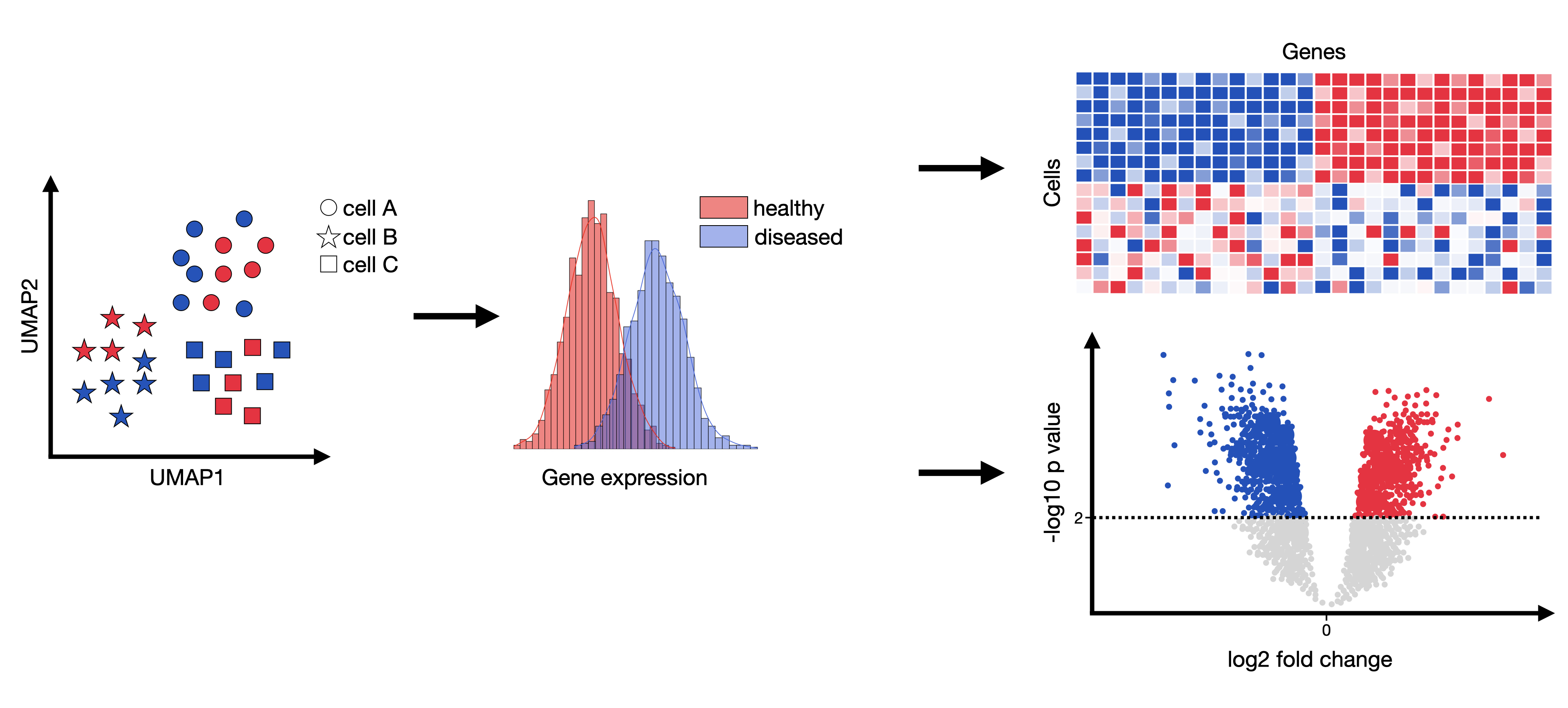
Differentially Expressed Genes (DEG) 분석 개념 DEG 분석은 두 개 이상의 샘플 그룹 간 유전자 발현 차이를 식별하는 과정이다. 이는 특정 조건이나 처리에 의해 유발된 유전자 발현 변화를 찾아내어 생물학적 메커니즘을 이해하는 데 도움을
3.Gene Set Variation Analysis 알고리즘
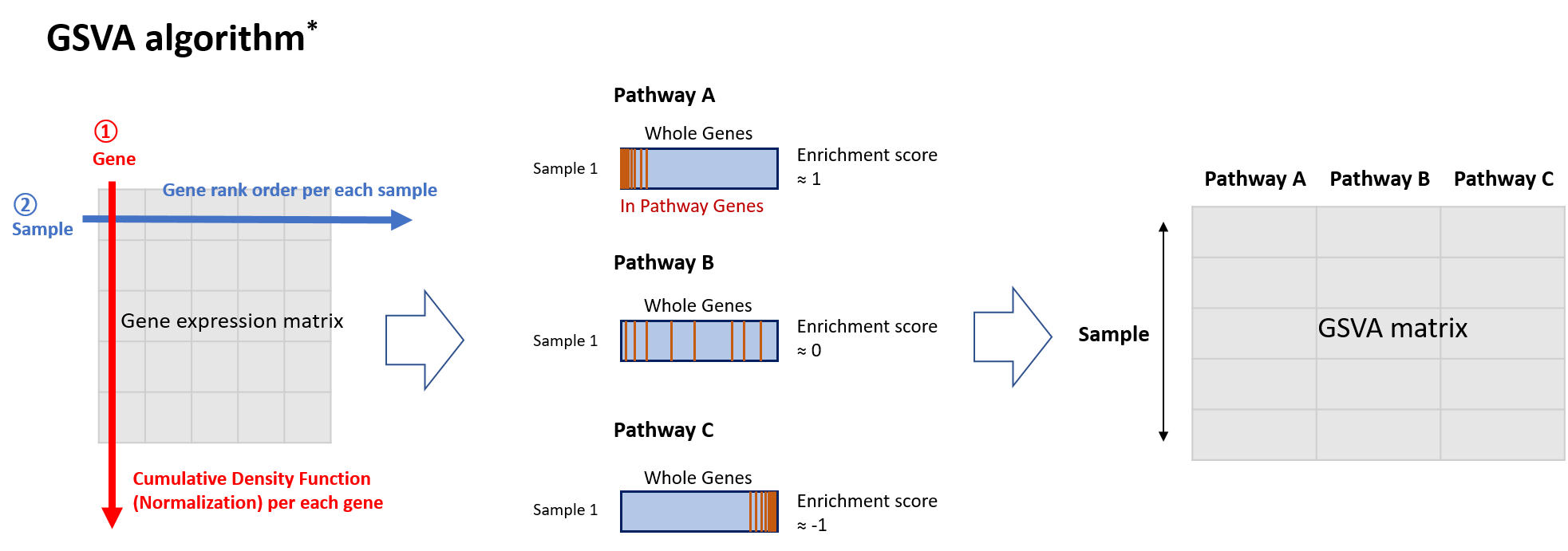
샘플 단위의 Gene set enrichment analysis (GSEA) 를 위해 많이 쓰이는 Gene Set Variation Analysis (GSVA; BMC Bioinformatics, 2013) 알고리즘을 리뷰하고자 한다.2024/05/29 및 구글 스콜라
4.RNA 시퀀싱과 음이항 분포

DEG 분석에서 음이항 분포를 사용하는 이유는 RNA-seq 데이터의 특성인 과분산을 반영하여 유전자 발현 수준을 보다 정확하게 모델링 하기 위함입니다.RNA-seq 데이터는 각 유전자에서 유래한 read의 개수를 세는 방식으로 얻어지는 이산적(discrete) 데이터
5.Louvain Clustering
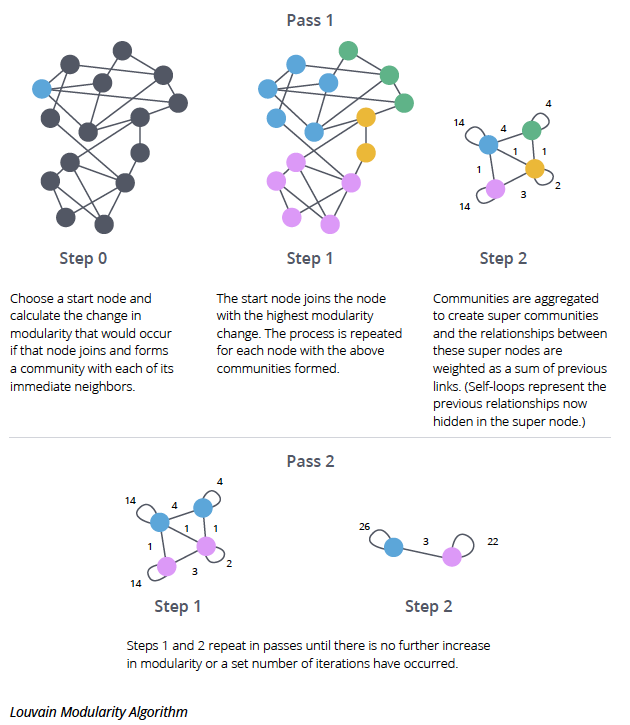
단일세포 RNA 시퀀싱(scRNA-seq)은 개별 세포 수준에서 전사체의 발현 양상을 분석할 수 있는 강력한 도구입니다. 이를 통해 세포의 이질성 (heterogeneity) 과 희귀 세포 집단 (rare cell population) 을 발견할 수 있습니다. 그러나
6.RNA 시퀀싱 데이터 정규화 / count normalization

※ 원문: HBC training 기반으로 작성하였습니다. RNA-sequencing 데이터를 얻고 Differentially expressed genes (DEGs) analysis 을 하고자 할때, 앞서 수행해야하는 것이 정규화(normalization)이다.